Introducción
La mastitis bovina es la principal causa de pérdidas económicas a nivel mundial debido a la disminución en la producción de leche, costos de tratamiento, descarte temprano de ganado, entre otros(1). Uno de los principales patógenos ambientales causante de mastitis bovina es E. coli, cuyas infecciones pueden ser desde moderadas hasta severas(2,3). Aunque a la fechano se han logrado determinar los factores de virulencia específicos de las E. coli asociadas a mastitis bovina, se ha propuesto que éstas pueden formar parte de un nuevo patotipo denominado Mammary Pathogenic E. coli (MPEC)(4). De acuerdo con esto último, estudios más recientes a nivel genómico han revelado la presencia de grupos de genes que codifican para sistemas se secreción tipo VI, biosíntesis de lipopolisacáridos, formación de biopelículas y sistemas de captación de hierro, característicos de las E. coli asociadas a mastitis bovina(5-8). Sin embargo, otros autores sugieren que no hay evidencia suficiente de que las E. coli causantes de mastitis pertenecen a un patotipo particular(9).
Diversos análisis filogenéticos de E. coli han permitido clasificarlas en cuatro grupos principales A, B1, B2 y D, en donde las bacterias comensales no patógenas pertenecen principalmente a los grupos A y B1, aunque estos filogrupos también incluyen patógenos importantes. Mientras al grupo B2 y en menor proporción al D, pertenecen las bacterias asociadas con infecciones extra-intestinales(9,10). Para el caso de E. coli causantes de mastitis bovina se ha observado que pertenecen principalmente a los grupos filogenéticos A y B1(11,12,13). Además, análisis genéticos y genómicos han revelado que las E. coli asociadas a mastitis bovina son muy diversas. En este sentido se ha reportado que las E. coliasociadas a mastitis bovina tanto persistente como transitoria, muestran diversidad fenotípica y genotípica(14). Asimismo, el análisis a nivel genómico de diversos aislados de E. coli, causantes de mastitis bovina y comensales, ha mostrado en ninguno de los casos las bacterias pueden clasificarse en un grupo filogenético en particular, sino que al parecer en ambos casos se han originado de diferentes linajes(9).
La habilidad para colonizar e invadir el epitelio mamario bovino ayuda a las bacterias a evadir la respuesta inmune e infectar de forma persistente. En este sentido, se ha demostrado que E. coli causante de mastitis persistente invade células epiteliales mamarias en forma eficiente,aunque los mecanismos empleados para tal fin no se conocen con precisión, pero se observó que las bacterias que muestran mayor movilidad tienen mayor virulencia(15). De igual forma, la matriz polimérica extracelular de las bacterias formadoras de biopelículas las hace más resistentes a diversas sustancias químicas con actividad antimicrobiana producidas por las células, además de que las protege del sistema inmune innato(16,17).
Generalmente, para el estudio de la virulencia bacteriana se utilizan diversos modelos animales; sin embargo, muchos de esos modelos son costosos y presentan algunos problemas éticos. Por lo cual, en épocas recientes, uno de los modelos que ha tomado importancia son las larvas del insecto Galleria mellonella, en las que se ha analizado la virulencia de bacterias patógenas, tanto Gram positivas como negativas, así como hongos(18,19). Por ejemplo, se ha observado que G. mellonella es un modelo adecuado para el estudio de variantes patógenas de E. coli tanto intestinales como extraintestinales(20,21,22).
El objetivo del presente trabajo fue estudiar la diversidad genética, determinar los grupos filogenéticos, la capacidad de formación de biopelículas y movilidad de E. coli aisladas de casos de mastitis bovina subclínica, así como evaluar la virulencia de aislados representativos in vivo usando como modelo de estudio larvas de G. mellonella.
Material y métodos
Preparación de extractos de ADN y determinación del grupo filogenético
Se estudiaron 34 E. coli resistentes a antibióticos, que fueron aisladas de casos de mastitis bovina subclínica en el occidente del estado de Michoacán, México, y las cuales fueron descritas previamente(23). A partir de estos aislados se prepararon extractos totales de ADN de acuerdo a lo descrito en la literatura(24). Para la determinación del grupo filogenético al que pertenecen las E. coli se siguió el esquema descrito en la literatura(10). Los primers utilizados para la amplificación de los fragmentos fueron: ChuA.1 (5’-GACGAACCAACGGTCAGGAT-3’) y ChuA.2 (5’-TGCCGCCAGTACCAAAGACA-3’), YjaA.1 (5’-TGAAGTGTCAGGAGACGCTG-3’) y YjaA.2 (5’-ATGGAGAATGCGTTCCTCAAC-3’) y TspE4C2.1 (5’-GAGTAATGTCGGGGCATTCA-3’) y TspE4C2.2 (5’-CGCGCCAACAAAGTATTACG-3’). Las reacciones de PCR se realizaron en un volumen de 25 µl conteniendo: 1X PCR Master mix (Promega, Madison, WI, USA), 1 µM de cada primer y 2 µl de extracto de ADN bacteriano (50 ng). La amplificación de los fragmentos se llevó a cabo en un termociclador C1000 (Bio-Rad, México, DF, México) en las siguientes condiciones: un ciclo inicial de desnaturalización inicial a 94 °C por 5 min, seguido de 30 ciclos en las siguientes condiciones; desnaturalización a 94 °C por 30 s, alineamiento a 55 °C por 30 s y extensión a 72 °C por 30 s. La amplificación se terminó con un ciclo de extensión a 72 °C durante 5 min. Los productos amplificados se separaron en geles de agarosa al 1.5 % y se visualizaron en un fotodocumentador Universal Hood II (Bio-Rad).
Producción de biopelículas
La producción de biopelículas se realizó de acuerdo a la literatura(25) con algunas modificaciones como se describe a continuación. Las bacterias se sembraron en cajas Petri con agar soya tripticasa (AST, BD, México, DF, México), a partir de dichos cultivos se inocularon tubos con 2 ml de caldo soya tripticasa (CST) adicionado de 0.25 % de glucosa, se incubaron toda la noche a 37 °C con agitación constante. Al día siguiente los cultivos se diluyeron hasta igualar con el tubo 0.5 de la escala de MacFarland en TSB con 0.25 % de glucosa. De la dilución bacteriana se depositaron 200 μl por triplicado en placas de Elisa de 96 pozos y se crecieron por 24 h a 37 °C sin agitación. Como control negativo se usó medio de cultivo sin inocular y como control positivo se utilizó la bacteria formadora de biopelículas Pseudomonas aeruginosa ATCC 27853. Se descartó el cultivo bacteriano y los pozos de laplaca se lavaron tres veces con solución salina estéril, para la fijación de las células la placa se dejó secar a 60 °C por 1 h. Posteriormente, se adicionaron 200 μl de cristal violeta al 1% y se dejó a temperatura ambiente por 20 min, se lavó la placa con agua corriente hasta que no se observó desprendimiento de color y se dejó secar a temperatura ambiente. A cada pozo de la placa se le adicionaron 200 μl de etanol al 96%, se agitó para solubilizar el cristal violeta, el sobrenadante se transfirió a un tubo de microcentrífuga y se repitió el proceso, el volumen del tubo se llevó a 1 ml y se midió la densidad óptica a 570 nm (D.O.) en un espectrofotómetro SmartSpec Plus (Bio-Rad). El ensayo se repitió cuatro veces por triplicado y con los datos obtenidos se calculó el promedio de la D.O. De igual forma se calculó la densidad óptica de corte (D.O.c.), para lo cual se tomó como base el promedio de la D.O. del control negativo más tres veces su desviación estándar, obteniéndose un valor de 0.22. La clasificación de las bacterias formadoras de biopelículas se realizó como sigue: formadoras fuertes D.O>4xD.O.c., moderadas 2xD.O.c.<D.O≤4xD.O.c., débiles D.O.c.<D.O.≤2xD.O.c. y negativas D.O.≤D.O.c.(25).
Detección de genes asociados a la formación de biopelículas
Se analizó mediante PCR la presencia de dos genes asociados con la formación de biopelículas, fimA (subunidad principal de la fimbria tipo I) y csgA (subunidad principal de la fimbria curli). Para lo cual se amplificó un fragmento de 119 pb de fimA con fimA-F (5’-CTCTGGCAATCGTTGTTCTGTCG-3’) y fimA-R (5’-GCAAGCGGCGTTAACAACTTCC-3’) y de 178 pb de csgA con csgA-F (5’-GATCTGACCCAACGTGGCTTCG-3’) y csgA-R (5’-GATGAGCGGTCGCGTTGTTA CC-3’)(26). Las reacciones se realizaron en 25 µl conteniendo 1X PCR Master mix (Promega), 1 µM de cada primer, 2 µl de extracto total de ADN. Las condiciones utilizadas fueron las siguientes: un ciclo inicial de desnaturalización a 95 °C por 5 min, seguido de 30 ciclos a 94 °C por 30 s, 55 °C por 30 s y 72 °C por 30 s, las reacciones se terminaron con un ciclo de extensión a 72 °C por 10 min. Los productos amplificados se separaron en geles de agarosa al 2% y se visualizaron como se describió anteriormente.
Determinación de la movilidad tipo “swarming”
El análisis de movilidad bacteriana se realizó siguiendo el protocolo ya descrito(15). Para la movilidad tipo swarming los cultivos se crecieron toda la noche en caldo LB (Lysogeny Broth). Posteriormente se depositaron 5 µl en placas con agar swarming (LB con agar al 0.5%) adicionado con 0.5 % de glucosa. Las bacterias se incubaron por 12 h a 37 °C. Posteriormente se midió el diámetro del desplazamiento bacteriano, las determinaciones se repitieron al menos tres veces y con los datos obtenidos se calculó el promedio.
Rep-PCR de E. coli causante de mastitis bovina
Para el análisis de la diversidad genética se purificó ADN genómico de acuerdo a protocolos estándar(27), con el DNA purificado se realizaron amplificaciones por PCR en 25 µl de mezcla de reacción con los siguientes componentes: 12.5 µl de 2X Master mix (Promega), 3 mM de MgCl2, 5% de DMSO, 0.16 µg/µl de albumina de suero bovino, 100 ng de ADN y 2 µM del primer (GTG)5 (5’-GTGGTGGTGGTGGTG-3’). La amplificación se realizó en un termociclador C-1000 (Bio-Rad) en las siguientes condiciones: un ciclo de desnaturalización inicial a 95 °C durante 2 min, seguido de 30 ciclos a las siguientes condiciones; desnaturalización (94 °C/3 s y 92 °C/30 s), alineamiento (40 °C/1 min), extensión (65 °C/8 min) y se finalizó con un ciclo de extensión a 65 °C durante 8 min(28). Los productos amplificados se separaron en geles de agarosa al 1.5% y se visualizaron en un fotodocumentador Universal Hood II (Bio-Rad).
Para el análisis de los patrones de bandas obtenidos se usó el software GelJ versión 2(29). El tamaño de las bandas fue normalizado usando como estándar de referencia el marcador 1 kb DNA ladder (Promega), que tiene fragmentos entre 250 y 10,000 pb. Los coeficientes de similitud fueron generados por el método de Dice con 2 % de tolerancia entre los carriles y el dendrograma se construyó usando el método UPGMA.
Virulencia en Galleria mellonella
Se analizó la virulencia in vivo de bacterias representativas que mostraron los mayores índices de formación de biopelículas, así como no formadoras en larvas de G. mellonella; para lo cual, se utilizaron larvas de 150 a 200 mg, libres de daño aparente y sin melanización (manchas necróticas) (Petmmal, Cuautitlán Izcalli, México). Las larvas se almacenaron en a 30 °C en obscuridad hasta su uso. Para los ensayos de infección se utilizaron cultivos bacterianos crecidos en caldo LB hasta la fase exponencial media de crecimiento (D.O. 600 nm= 0.5), se centrifugaron a 12,000 rpm/min, se lavaron dos veces con 1 ml de MgSO4 10 mM, al final las bacterias se re-suspendieron en 1 ml de MgSO4 10 mM. A partir de esta suspensión celular, se realizaron diluciones seriales hasta obtener una concentración bacteriana de aproximadamente 1x105 UFC/ml, lo cual fue confirmado mediante el conteo de UFC en agar LB.
Para cada una de las bacterias a estudiar se utilizaron 10 larvas de G. mellonella, a las cuales se les inyectaron 10 μl de la suspensión bacteriana (1 x 105 UFC/ml) en la última pro-pierna, con una jeringa de insulina con aguja calibre 29G. Como controles negativos se inyectaron 10 larvas con MgSO4 10 mM sin bacterias y 10 más sin inocular. Las larvas, tanto inoculadas como no inoculadas se colocaron en cajas Petri estériles y se incubaron a 30 °C en obscuridad por 96 h. Se determinó el porcentaje de sobrevivencia a las 24, 48, 72 y 96 h, se registraron como muertas aquellas larvas que no respondieron al tacto(21). Los ensayos de infección con cada bacteria se repitieron al menos dos veces de forma independiente.
Resultados
Grupos filogenéticos y relación genética de E. coli asociada a mastitis bovina
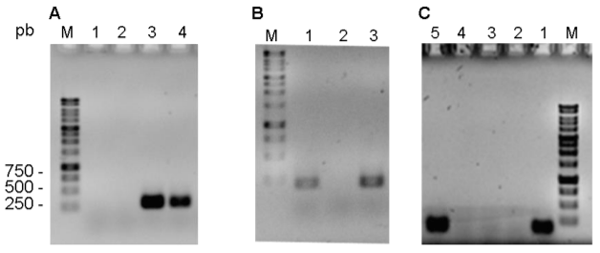
De acuerdo a los resultados obtenidos de la amplificación de los fragmentos chuA (279 pb), yjaA (211 pb) y TspE4.C2 (152 pb) (Figura 1), las 34 E. coli asociadas a mastitis bovina seclasificaron en tres grupos filogenéticos. El 67.7 % (23 aislados) pertenece al grupo A, el 17.6 % corresponde al grupo B1 (6 aislado) y el restante 14.7 % (5 aislados) corresponde al D (Figura 2B).
Figura 1: Geles de agarosa de los resultados positivos y negativos representativos de la amplificación por PCR de: A) chuA (279 pb), B) yjaA (211 pb) y, C) TspE.C2 (152 pb)
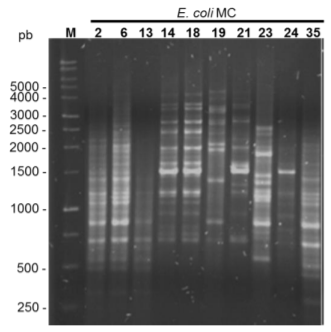
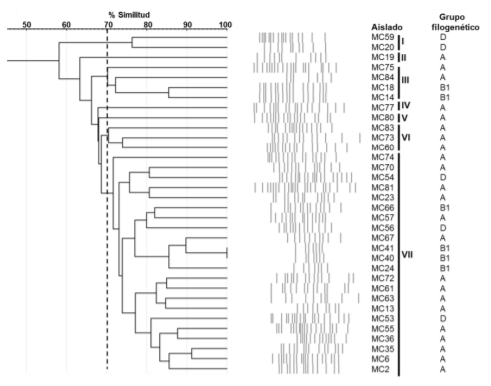
El análisis de relación genética mediante rep-PCR con el oligonucleótido (GTG)5 arrojó 32 patrones distintos de entre 7 y 21 bandas, con tamaños que oscilaron de 250 a 5,000 pb (Figura 2). El dendrograma generado a partir de los patrones de bandas usando el coeficiente de similitud de Dice por el método de UPGMA, indica que la similitud más baja entre los aislados fue de 58 %, mientras que la mayor similitud observada fue del 100 % en dos aislados. También se puede apreciar que considerando una similitud genética del 70 % (línea punteada) se identificaron 7 grupos de bacterias, de los cuales el grupo VII fue el más grande con 22 (64.7 %) aislados, el grupo III con 4 (11.8 %), el grupo VI con 3 (8.8 %) bacterias, el grupo I con 2 (5.8 %) y los grupos II, IV y V con un solo aislado (2.9 %), respectivamente (Figura 3).
Figura 2: Gel de agarosa de la separación de los productos amplificados por (GTG)5-PCR, se indica el tamaño en pares de bases de algunas bandas del marcador de 1 kb
Figura 3: Agrupamiento de los patrones de bandas obtenidos en 34 E. coli asociadas a mastitis bovina, basado en el coeficiente de similitud de Dice generado mediante UPGMA
Movilidad, formación de biopelículas y detección de genes asociados
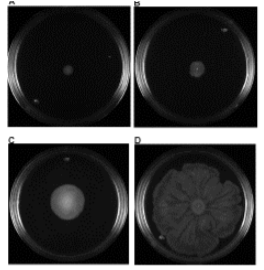
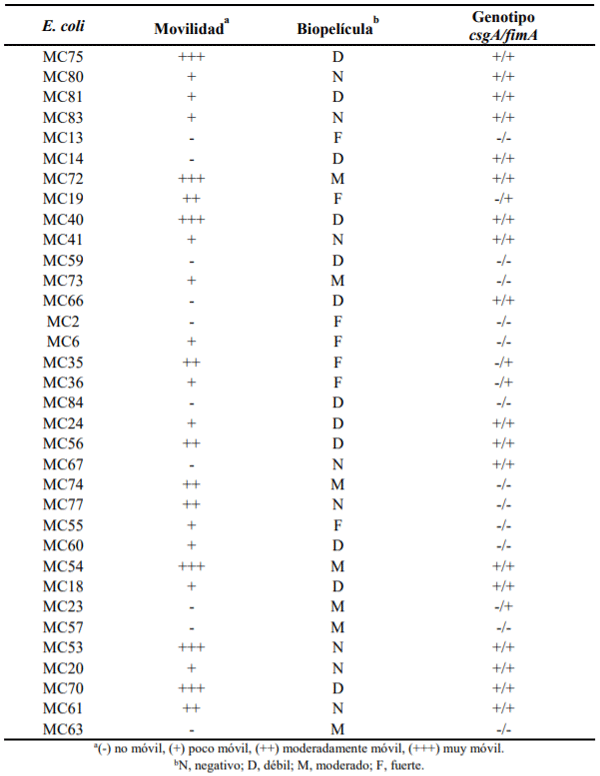
El análisis de la movilidad de las 34 E. coli, reveló que 10 no fueron móviles (-) y 24 mostraron movilidad. De éstas últimas de acuerdo al diámetro del desplazamiento 12 se consideraron poco móviles (+), 6 con movimiento moderado (++) y 6 fueron muy móviles (+++) (Figura 4 y Cuadro 1).
Figura 4: Resultados representativos de movilidad tipo ”swarming”. A) No móvil, B) Poco móvil, C) Moderadamente móvil, D) Muy móvil
Cuadro 1: Movilidad, formación de biopelículas y frecuencia de genes asociados a la formación de biopelículas de 34 aislados de E. coli provenientes de casos de mastitis bovina subclínica
Para la formación de biopelículas 26 aislados (76.5 %) formaron biopelículas en algún grado (D.O. >0.22) y las restantes 8 (23.5 %) no fueron formadoras (D.O. ≤0.22). Para el caso de las formadoras, siete se clasificaron como formadoras fuertes (D.O. >0.88) y moderadas (D.O. 0.45-0.88), mientras que 12 mostraron capacidad de formación débil (D.O. 0.23-0.44) (Cuadro 1). En 22 (64.7 %) de las 34 E. coli se detectó la presencia de alguno de los dos genes csgA/fimA, mientras que de las 26 formadoras en 15 se detectó uno o los dos genes. También los genes csgA y fimA se observaron en siete aislados no formadores de biopelículas.
Virulencia de E. coli en G. mellonella
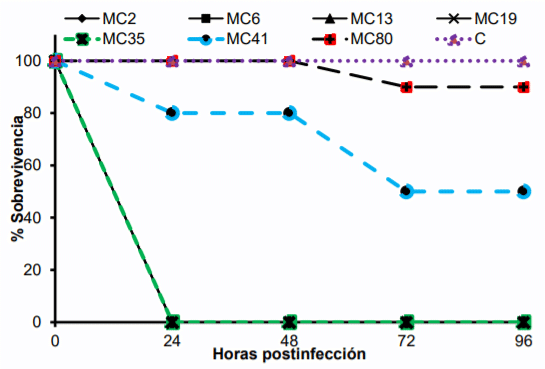
Con el propósito de analizar si hay diferencias en la patogénesis de los aislados de E. coli asociadas a mastitis bovina se realizaron ensayos de infección en larvas de G. mellonella con siete aislados representativos, cinco formadores fuertes de biopelículas y dos no formadores. Como se observa en la Figura 5, las bacterias formadoras de biopelículas mataron el 100 % de las G. mellonella a las 24 h después de la infección; mientras que, con los dos aislados no formadores de biopelículas se observó una mortalidad de entre 0 y 20 % a las 24 h y de 10 y 50 % a las 96 h. Para el caso del control no se observaron decesos durante las 96 h que duró el experimento.
Figura 5: Virulencia en G. mellonella de siete aislados representativos de E. coli, los formadores de biopelículas fueron MC2, 6, 13, 19 y 35. MC41 y MC80 no fueron formadores de biopelículas. También se muestra el control negativo (C)
Discusión
La mastitis bovina causada por E. coli es una infección que puede ser desde leve hasta severa, aunque a la fecha no se han asociado factores de virulencia específicos a dichas bacterias causantes de la enfermedad(11,30), incluso se ha sugerido que la severidad de la infección está determinada por las características del ganado más que por los microorganismos(31). Además, una línea de evidencia ha sugerido que las infecciones mamarias pueden ser producidas por cualquier cepa de E. coli(11), mientras que otra ha mostrado que no todos los aislados de E. coli son capaces de producir mastitis en modelos animales(5).
Por otro lado, se ha observado que las E. coli causantes de mastitis bovina pertenecen principalmente al grupo filogenético A, B1 y en menor proporción al grupo D(7). De acuerdo con los reportes previos, en este trabajo se encontró que el 67.6 % de las E. coli analizadas pertenecen al grupo filogenéticos A, 17.6 % al B1 y 14.7 % al grupo D. En este mismo sentido, en un estudio se observó que el 50 % de las E. coli asociadas tanto a mastitis persistente como transitoria pertenecen al filogrupo A, 28.6 % al B1 y 7.1 % al grupo B2 y D, respectivamente(14). Otros autores han descrito que el 44.88 % de las E. coli asociadas a mastitis pertenecen al filogrupo A, 38.58 % al B1 y 16.53 % al D(32). Resultados similares también han sido reportados en otros trabajos(13,33). Además, se ha observado que las E. coli aisladas de mastitis bovina presentan gran diversidad en serotipos, tipo de enfermedad (transitoria o persistente) y genotipo, incluso aunque pertenezcan a un mismo grupo filogenético determinado(3,34). En este sentido, las 34 E. coli analizadas en este trabajo presentaron gran diversidad genética, solo dos de ellas mostraron patrones de bandas idénticos, las restantes presentaron similitud entre el 92 y 58 %. Los resultados obtenidos también indican que no hay una separación clara entre los diferentes filogrupos, lo cual indica una gran heterogeneidad entre las E. coli causantes de mastitis bovina en nuestra área de estudio. Otros autores también han observado gran diversidad genética en E. coli causantes de mastitis bovina(3,30,34,35).
La habilidad de formar biopelículas en bacterias patógenas les confiere protección contra el sistema inmune del hospedero y resistencia a antibióticos, además de ser importante para la virulencia(26). En este sentido, se ha observado que las bacterias asociadas a mastitis bovina muestran un patrón variable en la formación de biopelículas(36). Aunque en las infecciones recurrentes se ha asociado a aquellas bacterias con la capacidad de formación de biopelículas(16). Los resultados descritos en este trabajo muestran que 20.6 % de los aislados pueden formar biopelículas de forma fuerte y moderada, mientras que 35.3 % lo hace de forma débil. De forma similar en otros estudios se ha observado que E. coli asociadas a mastitis bovina son capaces de formar biopelículas de forma variable, donde 18.5 % lo hace de forma fuerte, 40.7 % moderada y débil(36). En otro estudio se describió que 40 % de las E. coli asociadas a mastitis bovina fue formador fuerte de biopelículas, 12 y 4 % lo hicieron de forma moderada y débil, respectivamente(33).
De las 34 E. coli analizadas, en el 52.9 % se detectó la presencia de los genes csgA y fimA, en el 11.8 % solo se detectó fimA y en el restante 35.3 % no se detectó ninguno de los dos. En 15 de las 26 E. coli formadoras de biopelículas se detectó la presencia de al menos uno de los genes, mientras que en siete no formadores se detectó al menos uno de los dos genes. En este sentido, ambos genes se han asociado a la formación de biopelículas, al igual que otra variedad de genes y condiciones ambientales(37). Por lo cual, se requieren más estudios para caracterizar con más detalle los factores que afectan o promueven la formación de biopelículas en nuestra colección bacteriana. Por otro lado, se ha descrito que la formación de biopelículas varía dependiendo de la cepa, medio de cultivo, metodología y método de cuantificación. En este estudio solo se probó un medio de cultivo y una temperatura, por lo que pudiera ser que en otras condiciones las bacterias no formadoras de biopelículas y positivas para los genes csgA y fimA sean capaces de formar biopelículas(38).
Otro factor importante para que las bacterias se diseminen y produzcan infecciones persistentes en el ganado es la movilidad(15). De acuerdo con lo anterior se encontró que 70.6% de las E. coli estudiadas fueron móviles, esto sugiere que en nuestra colección tenemos bacterias tanto de mastitis persistente como transitoria. Además, los resultados obtenidos in vivo indican que las bacterias formadoras de biopelículas mostraron mayor virulencia contralas larvas de G. mellonella en comparación con las no formadoras. Aunque se requiere más trabajo para establecer la correlación entre la patogénesis in vivo en G. mellonella con la severidad de la enfermedad causada por E. coli. Los resultados presentados en este trabajo sugieren que este organismo puede ser un modelo adecuado para analizar la patogénesis de las E. coli causantes de mastitis.
Conclusiones e implicaciones
Los resultados presentados en este trabajo indican que las E. coli asociadas a mastitis bovina en nuestra área de estudio son muy diversas tanto a nivel genético como fisiológico. Y que las bacterias que tienen la habilidad para formar biopelículas de forma fuerte son más patógenas que las no formadoras. La diversidad genética y fenotípica de las bacterias estudiadas indica que no hay cepa, genotipo o factor de virulencia específico asociado a la enfermedad. Sin embargo, debido a que las bacterias formadoras de biopelículas se han asociado con infecciones mamarias recurrentes y persistentes, es importante estudiar mejores estrategias para el control de las infecciones causadas por esas bacterias. Lo cual podría disminuir las pérdidas económicas asociadas con la reducción en la producción y calidad de la leche.
Agradecimientos
Este trabajo fue apoyado por PRODEP (IDCA-11106) y por la UCEMICH (proyectos UCEMICH-2016-006 y UCEMICH-2017-004). Los autores agradecen al Dr. Iván Medina y al Dr. José Antonio Aguilar por su valiosa ayuda en la revisión del manuscrito.