
Contenido patrocinado por:
FARVET
Desarrollo de una vacuna multiepitópica contra el virus de influenza aviar A-H5N1 en base a herramientas inmunoinformáticas
Publicado: 9 de diciembre de 2015
Por: Manuel Ramirez Saenz, David Requena Anicama, Hugo Jesus Valdivia Olarte, Carlos Bueno, Luis Enrique Saravia, Reynaldo Ponce, Manolo Fernandez, Francesca Falconi Agapito, Ph. D. Mirko Zimic. Farvet S.A.C.

Introducción.
La gripe aviar es una enfermedad infecciosa viral de las aves. El virus de la influenza aviar infecta no sólo a las aves silvestres, sino también a las aves de corral domésticas y causan brotes a gran escala, generando repercusiones en la economía local, mundial y el comercio internacional. La mayoría de los virus de la gripe aviar normalmente no afectan a los humanos, sin embargo existen subtipos que sí lo logran, como el A-H5N1 y A-H7N9, generando graves consecuencias [1]. Se calcula que en el mundo han muerto cerca de 800 millones de aves de diferentes especies [2].
Para prevenir la infección efectiva con el virus de la influenza se recurre al uso de vacunas. En general, el desarrollo de vacunas convencionales (vivas, atenuadas) es un proceso que puede tomar entre 10 a 15 años. Además del prolongado tiempo de desarrollo, una de las mayores limitantes de las vacunas compuestas por virus atenuados es el riesgo de reactivación del patógeno. Frente a ello, existe otro tipo de vacunas basadas en proteínas recombinantes, las cuales son más seguras. Sin embargo, estas incluyen un alto costo de producción y generalmente se limitan a un número reducido de antígenos empleados, por lo cual no alcanzan una efectividad óptima. En estos últimos años, el Laboratorio FARVET S.A.C. ha venido desarrollando una investigación novedosa y pionera en inmunoinformática aviar, la cual se basa en conocimientos de genómica, bioinformática, biología molecular e inmunología aviar. Como primeros resultados, ha logrado la predicción de epítopes inmunogénicos de diversas proteínas de la influenza aviar A-H5N1 y el desarrollo de una proteína multiepitópica potencialmente protectiva contra la gripe aviar.
La gripe Aviar.
El virus de la influenza A es un virus segmentado, cuyo genoma está compuesto de 8 segmentos de RNA de una sola cadena codificante en sentido negativo. Tres de estas proteínas se expresan en la membrana y son incorporadas sobre la envoltura del virión: Hemaglutinina (HA), Neuraminidasa (NA) y la proteína matriz (M1 y M2). La bicapa lipídica del virus es una estructura de mosaico que incluye lípidos saturados, no saturados, colesterol y componentes derivados de la membrana plasmática del huésped. Incluye además otras cinco proteínas: la polimerasa B2 (PB2), la polimerasa A (PA), la proteína de nucleocápside (NP), la polimerasa B1 (PB1) y la proteína no estructural (NS1 y NS2) [3] (Figura 1).
VIRION.
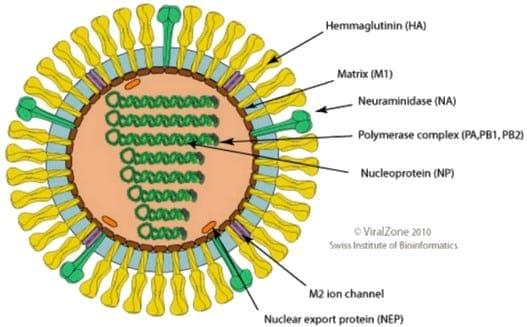
Figura 1. Estructura del virión del virus de la influenza A.
El complejo de mayor de histocompatibilidad El Complejo Mayor de Histocompatibilidad (CMH), juega un rol importante en la respuesta inmunológica mediante la presentación de fragmentos (epítopes) de bacterias, virus y/o parásitos a los linfocitos [4]. Los epítopes son la mínima unidad inmunológica capaz de estimular una respuesta inmune. Luego de darse el reconocimiento de los epítopes y la formación del complejo CMH-epítope, estos son presentados a los receptores de los linfocitos T. Las moléculas del CMH son de dos tipos: clase I y clase II, los cuales producen la activación de los linfocitos CD8+ y CD4+, respectivamente (Figura 2).
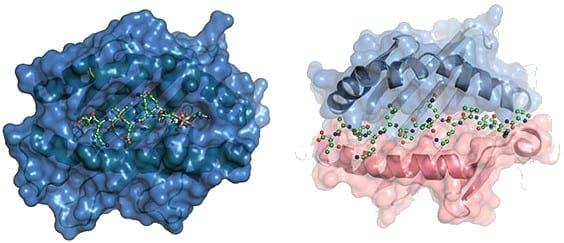
Figura 2. A. Estructura 3D del Complejo Mayor de Histocompatibilidad Tipo I conteniendo un epítope en el surco, el cual está conformado por la subunidad alfa. B. Estructura 3D del Complejo Mayor de Histocompatibilidad Tipo II conteniendo un epítope en el surco, el cual está conformado por las subunidades alfa y beta.
Inmunoinformática.
La inmunoinformática es un área novedosa, que consiste en la aplicación de la bioinformática y la inteligencia artificial para el estudio del sistema inmunológico. Esta disciplina emergente aborda aspectos importantes de inmunología y vacunología, basándose en el análisis de grandes bases de datos de pruebas experimentales para desarrollar métodos de predicción. Ello permite obtener candidatos con mayor probabilidad de éxito, permitiendo reducir el número de pruebas experimentales necesarias [5]. Entre sus ventajas, la inmunoinformática permite la identificación computacional de potenciales epítopes, mediante la predicción de afinidad entre un péptido cualquiera y el CMH y bajo la hipótesis que seleccionar a los péptidos con buena afinidad con el CMH predispone a una posterior presentación a los linfocitos T y el desarrollo de una respuesta inmune efectiva [6].
Materiales y métodos.
Secuenciamiento de los genes del CMH I y II del pollo Cobb.
Se realizó el secuenciamiento de los genes de las cadenas alfa y beta que conforman el CMH del pollo Cobb, tanto para la clase I y como para la clase II. Este procedimiento se realizó siguiendo el protocolo estándar del secuenciador de ADN 454 GS Junior de Roche en el laboratorio de Biología Molecular de FARVET S.A.C. Un total de 6 animales fueron muestreados en distintas granjas ubicadas en Chincha, ciudad importante en la crianza de pollos ubicada en el sur de Perú.
Predicción de epítopes de alta afinidad al CMH I y II del pollo Cobb.
Se analizaron 1536 secuencias de aminoácidos disponibles en el Genbank, correspondientes a las proteínas del virus de la influenza aviar A-H5N1. De ellas, se realizó la predicción de afinidad al CMH-I y CMH-II. De este análisis, se identificaron péptidos con alta afinidad (SB), con baja afinidad (WB) y no afines (NB) al CMH. Adicionalmente, se puso especial atención en las proteínas Hemaglutinina (HA) y Neuraminidasa (NA). Se compararon los péptidos SB predichos teóricamente en estas proteínas con los epítopes con evidencia experimental reportados en literatura.
Diseño de proteínas multiepitópicas.
Los epítopes teóricos seleccionados para el CMH clase I fueron concatenados linealmente en una sola cadena proteica, llamada “proteína multiepitópica”. Esto se realizó análogamente para el CMH clase II (Figura 4). La posición de los epítopes en la proteína multiepitópica se organiza de tal manera que la interface entre dos epítopes no origine un péptido SB que pueda interferir con la respuesta inmune deseada. Para producir las proteínas multiepitópicas de manera recombinante, se diseñaron genes sintéticos en plásmidos. Estos genes fueron clonados y expresados como proteínas recombinantes en E. coli.
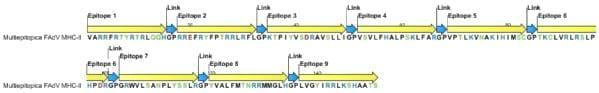
Figura 4. Cadena multiepitópica conteniendo epítopes teóricos.
Pruebas experimentales.
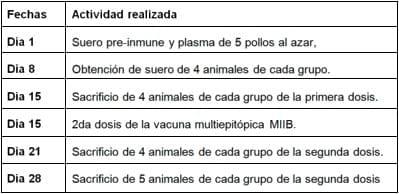
La inmunización se realizó en una población experimental de pollos Cobb SPF de aproximadamente 3-4 meses de edad y 2Kg de peso. Esta se dividió en dos grupos: un grupo vacunado (14 pollos) y un grupo control (12 pollos). La proteína MIIB fue administrada en conjunto con buffer urea 8M con 100mM de imidazol, mientras que el placebo consistió en urea 8M con 100mM de imidazol. En el día 1 se obtuvo suero pre-inmune y plasma. El monitoreo se realizó durante 28 días, en los cuales el día 1 y el día 15 se realizaron la primera y la segunda inmunización, respectivamente (Tabla 1).
Al finalizar el ensayo de vacunación, se realizó el ensayo de ELISA para todas las muestras del grupo control y el grupo vacunado. En el caso del grupo control positivo se usó el kit comercial ChR para detectar anticuerpos específicos de influenza.
Tabla 1. Cronología del ensayo de la proteína multiepitópica MIIB.
Resultados y discusión.
Del secuenciamiento de los genes del MHC de pollo Cobb, se identificaron 12 secuencias de MHC clase I y 11 secuencias de MHC clase II. Las secuencias de MHC clase I fueron alineadas para determinar la similaridad entre las secuencias y la obtención de los alelos más frecuentes en la población de pollos Cobb. Se obtuvo un alelo más abundante para el MHC clase I y 3 alelos para el MHC clase II.
De la predicción de epítopes afines a los alelos más frecuentes del CMH I, se obtuvo un total de 18 epítopes WB (de baja afinidad) en las proteínas PB2, NP, PA, HA, M1, PB1 (Tabla 2). Análogamente, de las predicciones de epítopes afines a los tres alelos de MHC clase II, se obtuvieron 40 epítopes SB (Strong Binding) de la proteína Hemaglutinina y 18 epítopes SB (Strong Binding) de la proteína Neuraminidasa.
Tabla 2. Muestreo de las proteínas correspondiente al proteoma del virus de influenza H5N1, usadas para la predicción de epítopes en el CMH I.
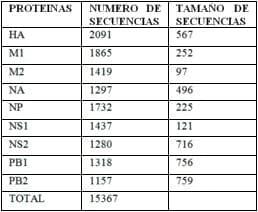
Tras la comparación de los epítopes experimentales y los predichos computacionalmente, se observó coincidencia en secuencia entre ambos. Esto se puede ver como sobrelapamiento en la Figura 5, lo cual refuerza la predicción mediante evidencia experimental.
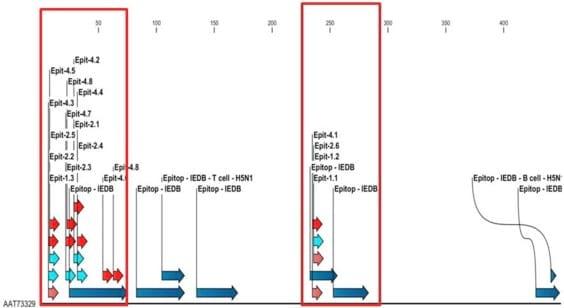
Figura 5. Comparación de los epítopes teóricos obtenidos en el proyecto contra los experimentales para el caso de la proteína Neuraminidasa. En los recuadros rojos se puede observar que existe sobrelapamiento entre los epítopes teóricos (color Rojo, Rosa y Celeste) contra los epítopes experimentales reportados (color Azul).
En el diseño de la proteína multiepitópica se obtuvieron de 3 secuencias de ADN (MIA, MIB y MIIB). Del proceso de producción de las proteínas, sólo se logró expresar y purificar la proteína MIIB, la cual se usó en las pruebas experimentales y los ensayos inmunológicos.
En la inmunización, a partir del séptimo día se detecta una estimulación de anticuerpos, la cual fue en aumento los días 14, 20 y 28. En todos los casos, se evidencia que la proteína MIIB presenta mayores niveles de anticuerpos respecto al grupo control. Además, se observa que los títulos de anticuerpos del grupo control están alrededor del cut-off (Figura 6).
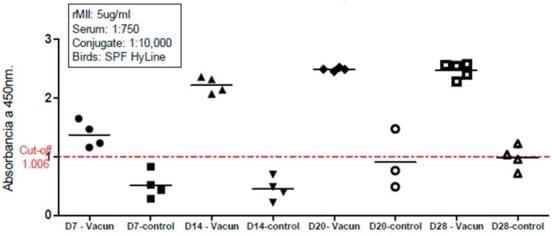
Figura 6. Medición de niveles de anticuerpos en los 28 días de ensayo de la proteína multiepitópica MIIB.
Finalmente, los resultados obtenidos en las pruebas de ELISA para el grupo pre-inmune, control positivo y la proteína MIIB, mostraron que se obtuvo un mayor número de anticuerpos contra la proteína multiepitópica MIIB (Figura 7).
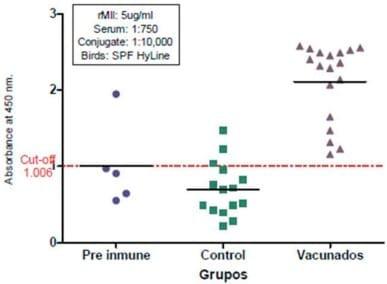
Figura 7. Título de anticuerpos contra la proteína multiepitópica MIIB y su comparación con los controles.
Conclusiones.
La proteína multiepitópica ha sido capaz de levantar una respuesta de tipo humoral en las aves inmunizadas. Este es un resultado alentador que refuerza el interés de seguir evaluando este tipo de antígenos a nivel de protección frente a una infección.
Financiamiento.
Este estudio ha sido financiado gracias al apoyo del FINCyT (Convenio N° 147-FINCyT-FIDECOM-PIPEA-2012), el laboratorio FARVET S.A.C. y con colaboración de la Universidad Peruana Cayetano Heredia.
Referencias.
1. http://www.who.int/mediacentre/factsheets/avian_influenza/es/
2. http://www.maplarevista.pe/2015/06/la-influenza-aviar-y-la-bioseguridad.html
3. http://viralzone.expasy.org/viralzone/all_by_species/6.html
4. Abbas, A.B.; Lichtman A.H. (2009). «Ch.3 Antigen capture and presentation to lymphocytes». Basic Immunology. Functions and disorders of the immune system (3rd edición). Saunders (Elsevier).
5. Flower DR. Towards in silico prediction of immunogenic epitopes. Trends in Immunology. 2003; 24(12):667-74.
6. Ole Lund, Morten Nielsen, Claus Lundegaard, Can Kesmir and Soren Brunak. Immunological Bioinformatic. 2006.
Temas relacionados:
Autores:
Mostrar más
Recomendar
Comentar
Compartir
23 de enero de 2020
Muchísimas gracias por su gran valiosa información para la influenza y la bioseguridad de la gripe aviar. Un gran saludos.
Recomendar
Responder

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Usuarios destacados

Katherine Calderón Mayo  FARVET
FARVET

Investigadores del Laboratorio de Biotecnología Molecular y Genómica
Perú
Perú









