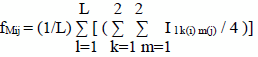Introducción
La diversidad de los recursos zoogenéticos y su conservación son importantes como fuente de variación genética de valor para la mejora genética y la adaptación a las circunstancias cambiantes ambientales y de mercado (FAO, 2010).
Es importante destacar al ovino Criollo uruguayo como uno de los recursos zoogenéticos disponible. Los mismos se encuentran clasificados como en riesgo de extinción, según los criterios establecidos por la Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO, 2010).
Los coeficientes de parentesco y consanguinidad son parámetros claves en el manejo genético de las poblaciones (Gómez-Romano et al., 2014). Cuando no se dispone de registros genealógicos, como es el caso de la población de ovinos Criollos en estudio, existe la posibilidad de recurrir a los marcadores moleculares para calcular dichos parámetros (Toro et al., 2009). El parentesco molecular se define como la probabilidad de que dos alelos de un locus elegido al azar sean idénticos por estado (Fernández et al., 2005). De forma análoga, la consanguinidad molecular de un individuo es la probabilidad de que dos alelos de un locus sean idénticos en estado (Toro et al., 2014).
El objetivo de este trabajo fue calcular el parentesco y la consanguinidad en base a la información que brinda un chip de alta densidad de SNP en el rebaño de ovinos Criollos del Parque Nacional de San Miguel.
Materiales y Métodos
Se analizó la información genómica de los ovinos Criollos del rebaño localizado en el Parque Nacional de San Miguel, el cual está administrado por el Servicio de Parques del Ejército. Se tomaron muestras de sangre de la vena yugular utilizando tubos con EDTA de la cual se extrajo ADN en base al protocolo de Sambrook modificado (Sambrook et al., 1989), en la Unidad de Biotecnología de INIA Las Brujas. El genotipado se realizó con paneles de 606.000 SNP (AgResearch, Nueva Zelanda), de 174 animales, que representaban el 59 % de la población existente. Para los análisis se utilizó el software GoldenHelix SNP & Variation Suite (SVS) (GoldenHelix Inc., Bozeman, MT, USA). La calidad de la información genómica se evaluó a través de los valores de call rate por muestra (umbral superior a 95%), y por SNP (umbral superior a 90%).
El coeficiente de parentesco molecular (fM) entre dos individuos i y j se calculó como:
siendo: L: número de marcadores; Il k(i) m(j): identidad del alelo k ésimo del individuo i con el alelo m ésimo del animal j en un locus L, que toma valor de uno si son alelos idénticos o de cero si no lo son (Saura et al., 2013).
El coeficiente de consanguinidad molecular (FM) para el individuo i se estimó como FMi = 2fMii –1 (es decir, como la proporción de genotipos homocigotos) (Saura et al., 2013).
Resultados y discusión
Del total de las muestras genotipadas, 158 muestras y 562.294 SNP cumplieron los criterios de calidad establecidos. El promedio de los coeficientes de parentesco y consanguinidad molecular fueron de 0,759 ± 0,013 y 0,755 ± 0.015, respectivamente. Resulta fundamental resaltar que los coeficientes moleculares y genealógicos se encuentran en diferentes escalas, ya que el parentesco y la consanguinidad molecular se refieren a idéntico por estado y en el caso de los genealógicos es idéntico por descendencia. En un estudio en cerdos Ibéricos con un chip de 60k de SNP el coeficiente de parentesco y consanguinidad molecular promedio fueron de 0,82 ± 0,015 y 0,81 ± 0,016, respectivamente, mientras que los genealógicos 0,42 ± 0,047 y 0,39 ± 0,037, si se comparan los valores genealógicos con los moleculares, estos últimos son mayores, representan casi el doble (Saura et al., 2013). El desarrollo de las nuevas tecnologías de genotipado masivo ha transformado el paradigma de que la genealogía es el parámetro más apropiado para medir la diversidad genética. Esto se debe a que el manejo basado en el parentesco molecular mantiene igual o es hasta usualmente mayor el nivel de diversidad genética logrado, y a su vez se logra mayor precisión en su estimación (Gómez-Romano et al., 2013).
Conclusión
Los resultados obtenidos remarcan la importancia del uso de información molecular en poblaciones sin registros genealógicos para la obtención del parentesco y consanguinidad molecular. Dichos coeficientes forman parte de la caracterización genética de estos ovinos y resultan fundamentales al momento de diseñar un plan de conservación.
Bibliografía
FAO. 2010. La situación de los recursos zoogenéticos mundiales para la alimentación y la agricultura. Comisión de recursos genéticos para la alimentación y la agricultura. Organización de las naciones unidas para la agricultura y la alimentación Roma.
Fernández J, Villanueva B, Pong-Wong R, Toro M. 2005. Efficiency of the use pedigree and molecular marker information in conservation programs. Genetics. 170:1313-1321.
Gómez-Romano F, Sölkner B, Villanueva B, Mészáros G, de Cara MAR, Pérez O’Brien AM, Fernández J. 2014. Genomic estimates of inbreeding and coancestry in Austrian Brown Swiss cattle. Consultado: 07/10/2014. Disponible en:
https://asas.org/docs/default-source/wcgalp-posters/842_paper_9791_manuscript_943_0.pdf?sfvrsn=2
Gómez-Romano F, Villanueva B, de Cara MAR, Fernández J. 2013. Maintaining genetic diversity using molecular coancestry: the effect of marker density and effective poplation size. Genet. Sel. Evol. 2;45:38.
Sambrook J, Fritsch E, Maniatis T. 1989. Molecular Cloning. A laboratoty Manual. (Eds) 2. Cold Spring Harbor Laboratory Press.
Saura M, Fernández A, Rodríguez M, Toro M, Barragán C, Fernández A, Villanueva B. 2013. Genome-wide estimates of coancestry and inbreeding in a closed herd of ancient Iberian pigs. PLOS ONE, 8 (10). SVS Golden Helix. Consultado: 10/09/2014. Disponible en:www.goldenhelix.com/SNP_Variation/SNP_Analysis_Package/index.html).
Toro M, Villanueva B, Fernández J.2014.Genomics applied to management strategies in conservation programmes. Livestock Science. 166:48-53.
Toro M, Fernández J, Caballero A. 2009. Molecular characterization of breeds and its use in conservation. Livestock Science. 120, 174-195.