Huevos contaminados
Evaluación de tres métodos de extracción de ADN de Salmonella SP en huevos de gallina contaminados artificialmente
En este estudio se evaluó la eficacia de tres métodos para la extracción de ADN de Salmonella sp. en huevos de gallinas libres de patógenos específicos (SPF). Se utilizaron 75 huevos, los que fueron divididos en 5 grupos de 15. Tres grupos de huevos fueron inoculados con cultivos de Salmonella sp., un cuarto grupo fue inoculado con Escherichia coli y el quinto fue el control negativo, que recibió una solución estéril a 0.85%. Los huevos fueron incubados a temperatura ambiente (20 - 25ºC) durante 24 horas. Después de este intervalo, se recolectaron las yemas de cada grupo, las cuales fueron homogenizadas y centrifugadas por 10 minutos, siendo el sobrenadante resultante descartado. Seguido a eso, se adicionó PBS pH 7,2 y se centrifugó de nuevo. El sedimento obtenido de cada grupo fue utilizado para la extracción de ADN genómico bacteriano. Se utilizaron tres métodos de extracción uno con el kit comercial GenElute Bacterial Genomic y los otros con partículas de sílice con diferentes sales, las cuales fueron yoduro de sodio ó reactivo Dnazol. El ADN extraído se mantuvo a una temperatura de - 20ºC hasta la evaluación por PCR. Los primers utilizados fueron relacionados con el gen invA. Los productos de amplificación se visualizaron en un transiluminador de luz ultravioleta. Los resultados obtenidos con la extracción de ADN bacteriano demostraron que el método con partículas de sílice utilizando yoduro sódico fue el más eficiente, para obtener suficiente calidad y cantidad de DNA para así conseguir un buen resultado con la técnica de PCR.
Palabras Clave: Bacterias, Agentes caotrópicos, Huevo.
La Reacción de Cadena de Polimerasa-PCR, parece ser una estrategia útil para la detección de bacterias del género Salmonella en diferentes sustratos como carnes, sangre, leche y huevos. Para el uso de la técnica de Reacción en Cadena de Polimerasa es esencial la obtención de ADN de buena calidad para así conseguir buenos resultados en las pruebas, donde los excesos de estructuras celulares y proteínas pueden inhibir el proceso de amplificación. Las extracciones de ADN deben proporcionar una buena estandarización en las bandas, con cantidad y calidad suficiente para no causar interferencia en los patrones de migración de geles de electroforesis.
Eso indica que nuevos protocolos aun necesitan ser probados para diferentes sustratos, con el objetivo de desarrollar nuevas tecnologías e incluso mejorar los ya existentes. Por ello, el objetivo de este trabajo fue evaluar la eficacia de tres métodos para la extracción de ADN de Salmonella sp. en huevos de gallinas libres de patógenos específicos (SPF).
Se utilizaron 75 huevos blancos de gallinas libres de patógenos específicos (SPF), los cuales fueron divididos en 5 grupos con 15 huevos cada uno. Los huevos fueron inoculados en la cavidad alantoidea: Inicialmente cada huevo fue desinfectado con una solución de tintura de yodo al 10% en el sitio de la perforación de la cascara, luego los huevos fueron perforados en la cámara de aire con ayuda de una aguja de acero inoxidable adaptada a una goma de calibre 30 x 12 mm. Se inocularon los huevos con una jeringa de tuberculina de 1mL, 0,1mL. Los huevos fueron inoculados con cultivos bacterianos, tres grupos de esos fueron inoculados con Salmonella sp. a una concentración de 105 UFC/ml, otro grupo fue inoculado con una cepa de E. coli en la misma concentración que los anteriores y los huevos del grupo control fueron sometidos a los mismos procedimientos empleando solución salina a 0,85%, taponada y esterilizada. Para hacer la inoculación se utilizo una aguja de calibre 12,7 X 0,33 mm que fue introducida en la cámara de aire colocado el contenido en la cavidad alantoidea, en un ángulo aproximado de 30°. Después de la inoculación los orificios fueron sellados con pegamento y los huevos transferidos a bandejas donde permanecieron por 24 horas a temperatura ambiente.
Después de este tiempo las yemas de cada grupo fueron recogidas asépticamente y colocadas en un beaker estéril y homogenizados. Luego se trasladaron 10 ml del homogenizado de yema a los tubos de centrifuga. Se centrifugaron 2.500 x g a temperatura ambiente. El sobrenadante fue descartado, y los sedimentos fueron resuspendidos en igual volumen con PBS pH 7,2. y centrifugados nuevamente eliminado el sobrenadante. El sedimento obtenido de cada grupo fue almacenado a -20°C, hasta la extracción de ADN.
La extracción de ADN bacteriano con el kit comercial- GenElute Bacterial Genomic ADN (Zigma®) se realizó según las instrucciones del fabricante. El ADN obtenido fue cuantificado y analizado en gel de agarosa (1%); el mismo fue teñido con bromuro de etidio y visualizado con luz ultravioleta.
Los dos otros protocolos que utilizaron partículas de sílice con DNAzol o NaI fueron basados en el trabajo de Boom et al. (1990).
A las muestras se le adicionaron 600 µL de NaI (o Dnazol), se calentaron a 55°C y agitadas suavemente por cinco minutos. El material obtenido fue centrifugado por 5 minutos a 12.000 x g a temperatura ambiente y el sobrenadante fue colectado con ayuda de una pipeta. A continuación se le agrego a la mezcla 50 μl de suspensión de sílice (dióxido de silício- Sigma Chemical Co., St. Louis, Mo) y fue homogenizada con un vórtex. La mezcla fue incubada en un agitador end-over-end (Speci-Mix, Thermolyne) por 10 minutos a temperatura ambiente, después se centrifugó por 30 segundos a 12.000 x g a temperatura ambiente, se desecho el sobrenadante por inversión del tubo. El sedimento fue enjuagado dos veces con 1 mL de tampón de lavado (50%, 50mM Tris-HCl pH 8, 10mM EDTA pH 8). Después de la centrifugación por 30 segundos a 14.000 x g a temperatura ambiente, todo el tampón de lavado fue removido con una pipeta.
Luego se adiciono 1 mL de acetona y fue homogenizado en el vórtex y centrifugado por 30 segundos a 14.000 x g a temperatura ambiente. El sobrenadante fue descartado y el residuo de acetona se evaporo del sedimento por el tubo con tapa abierta a una temperatura de 56°C durante 10 minutos. El ADN adherido al sílice fue eluido por adicción de 50 μl de TE (5 mM TRIS-HCl pH 8, 0,5 mM EDTA pH 8), incubado a 50°C por 5 minutos y centrifugado por 30 minutos a 14.000 x g a temperatura ambiente para solidificar el sedimento. El sobrenadante fue retirado con una pipeta, las muestras de ADN fueron extraídas analizadas y cuantificadas por lectura en espectrofotómetro NanoDrop ND-1000 (Thermo Fisher Scientific, Wilmington, DE, USA), que estima la cantidad de ADN de la muestra en μg/ μl y la cantidad del material por el valor obtenido en la razón DO260nm/ DO280nM.
Con las alícuotas de cada muestra de ADN, obtenidas a partir de los dos protocolos de extracción, se realizaron las reacciones de amplificación, con un volumen final de 50 µL que contenía: 2µL de DNA, 5 µl de tampón 10X ( 200mM Tris-HCl, pH 8, 500mM KCL- Invitrogen®) 1,0 µL de DNTP a 10mM ( Datp, dTTP, Dctp, Dgtp - Invitrogem), 1,5 µL de MgCl2 a 50 mM, 1µL de cada primer a 25 pmol , 0,3µL Taq polimerasa a 1U/µL y agua ultra pura q.s.p.
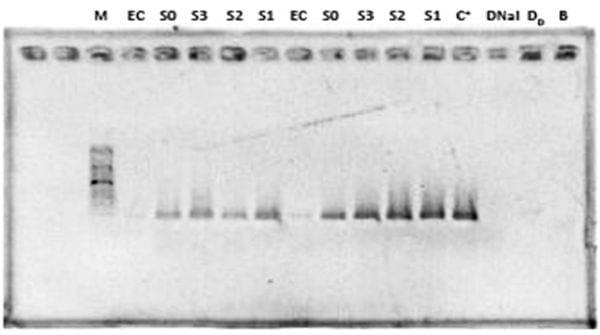
En cuanto al kit comercial - GenElute Bacterial Genomic DNA kit (Zigma®), no fue posible visualizar la amplificación de ADN en las muestras extraídas. Sin embargo, fue posible visualizar la amplificación de todas las muestras obtenidas en los tres protocolos por PCR. Fueron utilizados los primers derivados de el gen invA, obteniendo una banda de amplificación de ADN con 284 pares de bases como lo esperado en todas las muestras de Salmonella sp., no siendo observada ninguna amplificación de ADN de E. coli. Con esto podemos afirmar que el gen invA Salmonella sp. contiene secuencias únicas y demostró que este gen es un objetivo adecuado para PCR, con posibles aplicaciones diagnósticos de Salmonella sp.
El mejor resultado se obtuvo cuando el ADN fue extraído por medio de partículas de sílice con NaI, por la presencia de bandas mas intensas de ADN, indicando mayor representación de material genético (Fig. 1). Por lo tanto, la extracción de ADN por este método puede ser utilizado de manera segura en las pruebas de PCR, reduciendo así los costos de la técnica, principalmente en lo que se refiere a la adquisición de kits de extracción de ADN.
Rahn et al., (1992), usando un par de oligonucleotidos del gen invA, lograron detectar 626 cepas de Salmonella en 630 pruebas, a partir de una colonia aislada e incorporada directamente en el PCR. Ellos obtuvieron una sensibilidad del 99,4% y una especificidad del 100%, pues no observaron una reacción positiva en ningún control negativo pertenecientes a otras 33 enterobacterias evaluadas.
Según Rohland & Hofreiter (2007), el método de extracción utilizando partículas de sílice tiene algunas ventajas sobre otros protocolos, ya que es rápido y fácil, pudiendo utilizarse pequeñas cantidades de la muestra. Además, es muy fácil de implementar, ya que emplea un equipo estándar de laboratorio y pocos productos químicos. Por otro lado, posee eficiente remoción de inhibidores para PCR.






.jpg&w=3840&q=75)