
Contenido patrocinado por:
FARVET
Vacunación intranasal de hámsteres con el vector del virus de la enfermedad de Newcastle
Publicado: 14 de julio de 2022
Por: Manolo Fernández Díaz, Katherine Calderón Mayo, Aldo Rojas, Vikram Vakharia, Ricardo Choque Guevara, Angela Montalván, Astrid Poma Acevedo, Dora Rios-Matos, Andrés Agurto-Arteaga, María de Grecia Cauti-Mendoza, Norma Perez Martinez, Gisela Isasi-Rivas, Luis Tataje, Yacory Sernaque-Aguilar, Freddy Ygnacio Aguirre, Manuel Criollo-Orozco, Edison Huaccachi-Gonzalez, Elmer Delgado-Ccancce, Doris Villanueva-Pérez, Ricardo Montesinos, Kristel Gutiérrez-Manchay, Katherinne Pauyac Antezana, Ingrid Ramirez-Ortiz, Ste
Autores: Manolo Fernández Díaz, Katherine Calderón Mayo, Aldo Rojas, Vikram Vakharia, Ricardo Choque Guevara, Angela Montalván, Astrid Poma Acevedo, Dora Rios‑Matos, Andrés Agurto‑Arteaga, María de Grecia Cauti‑Mendoza, Norma Perez Martinez, Gisela Isasi‑Rivas, Luis Tataje, Yacory Sernaque‑Aguilar, Freddy Ygnacio Aguirre, Manuel Criollo‑Orozco, Edison Huaccachi‑Gonzalez, Elmer Delgado‑Ccancce, Doris Villanueva‑Pérez, Ricardo Montesinos, Kristel Gutiérrez‑Manchay, Katherinne Pauyac Antezana, Ingrid Ramirez‑Ortiz, Stefany Quiñones‑Garcia, Yudith Cauna‑Orocollo, Katherine Vallejos‑Sánchez, Angela Rios‑Angulo, Dennis Núñez‑Fernández, Mario I. Salguedo‑Bohorquez, Julio Ticona, Manolo Fernández‑Sánchez, Eliana Icochea, Luis A. Guevara‑Sarmiento, Mirko Zimic.
Introducción:
El coronavirus 2 del síndrome respiratorio agudo severo (SARS-CoV-2), es el agente causante de la enfermedad del coronavirus del 2019 (COVID-19), el cual reconoce la enzima convertidor de angiotensina-2 (ACE-2) que se encuentra presente sobre la superficie de varios tipos de células humanas incluyendo los neumocitos. La proteína spike (S) glicosilada de superficie es el principal atributo para la unión al receptor, promoviendo la endocitosis y lo cual resulta en la entrada del virus1,2.De las dos subunidades distintivas de la proteína S (S1 y S2), el extremo más distal de la subunidad S1 es el dominio de unión al receptor (RBD), que interactúa con el receptor ACE-2 a través del motivo de unión al receptor (RBM) para iniciar el proceso de infección y de entrada3. Ha sido demostrado que los anticuerpos neutralizantes de pacientes convalecientes de COVID-19 son comúnmente dirigidos hacia epítopes específicos en las subunidades S1 y S24-6. Nuestra comprensión más temprana de los coronavirus, como el coronavirus del síndrome respiratorio agudo severo (SARS-CoV) y el coronavirus del síndrome respiratorio del Oriente Medio (MERS-CoV) ha ayudado a identificar potenciales candidatos a vacunas contra el SARS-CoV-2 específico a la proteína S debido a su inmunogenicidad conocida7-10. También ha sido encontrado que la proteína S del SARS-CoV-2 lleva epítopes protectores potenciales a los linfocitos B y T con el potencial para ser vacunas candidatas11, 12. Además de la proteína S de longitud completa (pre-fusión o post-fusión), los dominios S1 y RBD son considerados objetivos de vacunas importantes13 y han sido el foco del desarrollo de vacunas.
Sin embargo, la secuencia de aminoácidos de S1/RBD se encuentra bajo una presión de selección, buscando una mayor afinidad por el ACE-214-18 o escapar de la neutralización mediante anticuerpos contra la subunidad S1 del SARS-CoV-219,20. Diferentes estrategias han sido aplicadas para el desarrollo de vacunas contra el SARS-CoV-2, buscando seguridad, efectividad y protección contra el virus, incluyendo vacunas basados sobre virus inactivado, ARNm, y usando vectores virales21-24.
El virus de la enfermedad de Newcastle (NDV), es el agente causal de la enfermedad de Newcastle (ND), ha sido usado como un vector viral para la expresión de diversos antígenos de muchos patógenos animales y humanos25-27. El NDV, también conocido como Avian orthoavulavirus 1, es un miembro de la familia Paramyxoviridae28 y lleva un genoma de ARN de sentido negativo de cadena única con un tamaño de aproximadamente 15.2 kb29,30. El NDV codifica seis proteínas estructurales en el siguiente orden: la nucleoproteína (NP), fosfoproteína (P), proteína matriz (M), proteína fusión (F), proteína hemaglutinina-neuraminidasa (HN), y la proteína larga (L), el cual es una polimerasa viral29, 30.
El NDV puede ser dividido en tres grupos de acuerdo a su virulencia en aves de corral: velogénico, mesogénico, y lentogénico29. La cepa LaSota de NDV es lentogénico (apatogenico), y es comúnmente usado como una vacuna de NDV atenuada viva en aves de corral. De forma importante, ello alcanza altos títulos en huevos embrionados de pollo, induce fuerte respuesta inmune celular y humoral, y puede ser administrado por la ruta intranasal30 debido a la abundancia de sus receptores en el tracto respiratorio superior. Ha sido demostrado que el NDV no representa una amenaza para la salud humana, y la mayoría de la población humana no muestra inmunidad pre-existente26, 31. Debido a la expresión ectópica y natural replicación citoplasmática, la vacuna vectorizada de NDV induce una respuesta inmune en la mucosa en el tracto respiratorio, y no recombina con el ADN del huésped durante la replicación32.
El NDV ha sido usado como un vector para el desarrollo de vacunas desde finales de la década de 1990 en diferentes huéspedes animales. La eficiencia de las vacunas basadas sobre este vector ha sido demostrada contra virus respiratorios, como el virus de la bronquitis infecciosa y reovirus aviar en pollos, SARS-CoV-2 en monos, y MERS-CoV en camellos33-36. Estos estudios han demostrado que es factible generar el NDV que expresa la proteína S de otros coronavirus, como el SARS-CoV y MERS-CoV, el cual confiere una fuerte inmunogenicidad y protección en ratones y primates no-humanos35,36. Recientemente, el NDV ha sido propuesto como un vector potencial para una vacuna contra el SARS-CoV-2.
Otros estudios, basados sobre vacunas vectorizadas de NDV que expresan la proteína S completa, que fue administrada tanto por la ruta nasal o intramuscular, evaluado en hámsteres y ratones, provocaron una alta respuesta inmune, provocando la producción de anticuerpos de inmunoglobulina G (IgG) e inmunoglobulina A (IgA). Los animales fueron fuertemente protegidos contra un desafío con el SARS-CoV-2. La infección, inflamación, o cualquier lesión patológica fue prevenido en los tejidos de los pulmones, mientras el peso corporal y movilidad física fue mantenido de forma normal. La carga viral en los pulmones de animales vacunados y desafiados, comparados con el control desafiado, fue reducida, así también como la diseminación viral en las turbinas nasales y pulmones de hámsteres y ratones37-40.
En este estudio, nosotros describimos el diseño y evaluación de una vacuna vectorizada de NDV intranasal en hámsteres desafiados con el SARS-CoV-2. En el presente estudio nosotros demostramos que el mismo nivel de eficiencia de protección y seguridad. En comparación, nuestra vacuna vectorizada de NDV expresó la subunidad S1 y RBD, y fueron administrados a través de la ruta nasal.
Resultados
En este estudio desarrollamos y evaluamos tres diferentes formulaciones de vacunas para uso intranasal, incluyendo un NDV que presenta el dominio RBD, un NDV que presenta la subunidad S1, y una combinación de ambos, el dominio RBD y la subunidad S1. Estas vacunas fueron administradas por vía nasal en dos dosis. La seguridad y eficacia fue confirmada en hámsteres desafiados con SARS-CoV-2. El vector NDV que expresa el S1 mostró una alta protección frente al desafío a diferencia con el vector NDV que expresa el RBD.
Desarrollo y caracterización de NDV recombinante que expresa antígenos RBD y S1 del SARS-CoV-2.Generación de rNDVs expresando individualmente el RBD y la subunidad S1 ambos genes del SAR-CoV-2. Células Vero-E6 fueron co-transfectadas con un plásmido ADNc de longitud completa de los constructos pFLC-LS1-HN-RBD y pFLC-LS1-S1-F junto con tres plásmidos de soporte codificando las proteínas NP, P, y L de NDV, esenciales para la replicación de NDV. A las 72 h post-transfección, las células mostraron varias placas visibles con un efecto citopático (ECP) típico de NDV, demostrando la recuperación exitosa de ambos virus recombinantes. Los sobrenadantes colectados cinco días después de la transfección fueron inyectados en las cavidades alantoideas de huevos embrionados de pollo SPF de 9 días de edad. El fluido alantoideo fue recolectado cuatro días después de la inoculación y analizado por HA utilizado células de glóbulos rojos de pollo. Encontramos títulos de HA positivos que oscilan desde 2 a 2048.
La presencia de los casetes de expresión HN-RBD y S1-F insertados dentro de la región no-codificante entre los genes P y M del genoma de NDV fueron verificados por RT-PCR, produciendo fragmentos de 1600 y 3028 pares de bases (pb), respectivamente. Estos fragmentos fueron subsecuentemente amplificados y secuenciados utilizando cebadores (NDV-3LS1-2020-F1 y NDV-3LS1-2020-R1), demostrando una inserción correcta dentro del genoma de NDV (Fig. 1). Estos nuevos NDV recombinantes fueron llamados rLS1-HN-RBD y rLS1-S1-F, respectivamente.
Expresión de las proteínas del SARS-CoV-2 en los virus rLS1‑HN‑RBD y rLS1‑S1‑F. En lisados de células infectadas, una banda fue detectada para cada uno de los virus recombinantes, con un peso molecular de ~90 kDa (S1-F) para el virus rLS1‑S1‑F y una banda de ~30 kDa (HN-RBD) para el virus rLS1-HN-RBD (Fig. 2A), respectivamente. Igualmente, el mismo resultado se obtuvo con el virus recombinante purificado (Fig. 2B), confirmando que las proteínas S1-F y HN-RBD fueron incorporadas en las partículas virales de rLS1-S1-F y rLS1-HN-RBD, respectivamente. Como se esperaba, estas bandas no fueron detectadas en las células infectadas con rLS1 y en las partículas virales purificadas del virus rLS1. Imágenes de exposición múltiple en Fig. 2A, B son previstas en el material complementario (Figuras complementarias 1, 2, respectivamente). Se generó un rLS1-F-RBD, expresando RBD fusionado con los dominios TM y CT del gen F de NDV.
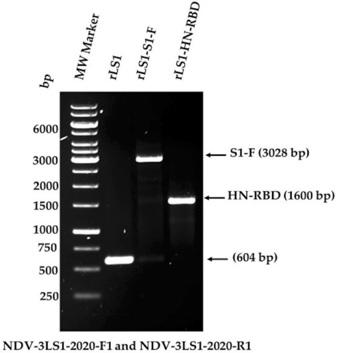
Figura 1. La inserción del casete de expresión en la región no-codificante entre los genes P/M del genoma de NDV fue verificado por RT-PCR utilizando los cebadores NDV-3LS1-2020-F1 y NDV-3LS1-2020-R1.
Sin embargo, después de insertar este constructo dentro del genoma de NDV, fuimos incapaces de detectar la expresión del RBD (tamaño esperado de 27.9 kDa) después de evaluar 4 clones mediante Western blot (Figura complementaria 3).
La expresión del RBD y de la subunidad S1 del SARS-CoV-2 fueron detectadas en células Vero-E6 infectadas con rLS1‑HN‑RBD y rLS1‑S1‑F mediante IFI. La expresión del RBD y de la subunidad S1 no fueron detectadas en células infectadas con el virus rLS1. La expresión de las proteínas de NDV fueron detectadas utilizando un antisuero en pollo específico para NDV, y un Goat Anti-Chicken IgY H&L-Alexa Fluor 488 en células Vero-E6 infectadas con los virus rLS1, rLS1-HN-RBD, y rLS1-S1-F (Fig. 2C). La detección de la subunidad S1 y el RBD del SARS-CoV-2 sobre la superficie de los virus rLS1-S1-F y rLS1-HN-RBD en células Vero-E6 fueron confirmados por citometría de flujo en dos experimentos independientes (Fig. 2D). Para el virus rLS1‑S1‑F, fueron detectadas 40.4% de células positivas, para el virus rLS1-HN-RBD fueron detectadas 10.2% células positivas, y para el virus rLS1 se detectaron hasta un 0.37% de células positivas. Un alto porcentaje de células fueron detectadas en el virus rLS1‑S1‑F que en el virus rLS1-HN-RBD.
Estabilidad genética de los virus rLS1-HN-RBD y rLS1‑S1‑F. Para demostrar la estabilidad génica y de expresión de los genes insertados, la RT-PCR se realizó en el tercer (P3) y sexto (P6) pasaje, la cual mostró la presencia del tamaño esperado de los genes insertados. El análisis de Western blot confirmó la expresión de las proteínas S1-F y HN-RBD durante estos pasajes, sugiriendo una estabilidad genética y una correcta expresión de los insertos en los genomas virales recombinantes (Fig. 3A, B) hasta el sexto pasaje (P6).
Estabilidad de la vacuna liofilizada. Antes de la liofilización, el título de la vacuna fue de 8.12 ± 0.03 log10 PFU/mL determinado por ensayo en placa y el título de HA fue 210.
Después de la liofilización, y después de almacenar las vacunas a 4°C, la vacuna fue reconstituida. Los títulos después de la reconstitución resultaron estables a los 1, 30, y 50 días con 8.37 ± 0.06, 7.69 ± 0.24, y 8.24 ± 0.05 log10 PFU/mL. Del mismo modo, los títulos de HA fueron estables después de cada uno de los tres períodos de almacenamiento (210). La expresión de las proteínas S1-F y HN-RBD en células Vero-E6 infectadas con la vacuna liofilizada de NDV fueron confirmadas después de la reconstitución a los 1, 30 y 50 días post-liofilización mediante Western blot (Fig.4).
Inmunogenicidad en hámsteres. La vacunación intranasal provocó anticuerpos específicos contra la proteína S y anticuerpos neutralizantes contra SARS-CoV-2 en hámsteres.Quince días después de la primera inmunización, el grupo de hámsteres inmunizados con las vacunas vivas rLS1-HN-RBD, rLS1-S1-F y la mezcla de ambas rLS1-HN-RBD/rLS1-S1-F desarrollaron anticuerpos específicos IgG en sueros contra S1 y RBD (Fig. 5A). A los 15 días post-refuerzo (30 días post-inmunización), hubo un incremento significativo en los títulos de anticuerpos IgG en suero. Por otro lado, el grupo control no indujo anticuerpos específicos para S1 y RBD de SARS-CoV-2. De manera importante, la inmunización con rLS1-S1-F y la mezcla de las vacunas rLS1-HN-RBD/rLS1-S1-F indujo significativamente un mayor nivel de anticuerpos IgG en suero específicos para S1 y RBD a los 15 días después del refuerzo en comparación con el rLS1-HN-RBD (Fig. 5B, C).
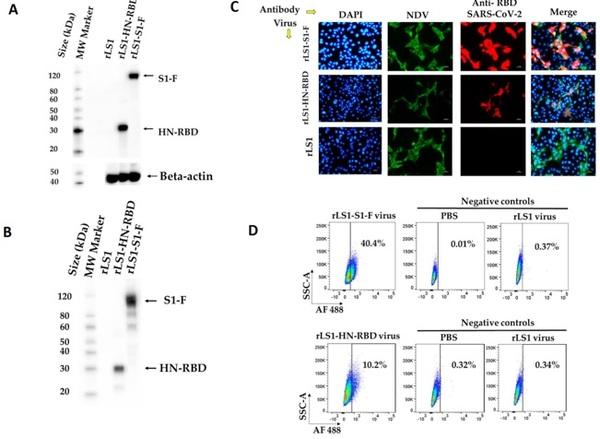
Figura 2. Expresión de las proteínas RBD y S1 del SARS-CoV-2 en células Vero-E6 y partículas de NDV. (A) Western blot para detección de la expresión de las proteínas HN-RBD y S1-F. Células Vero-E6 fueron infectadas con los virus a una MOI de 1.0. Después de 48 hpi, las células fueron lisadas y analizadas por Western blot. (B) Para verificar la incorporación de las proteínas HN-RBD y S1-F en los virus rLS1-HN-RBD y rLS1-S1-F, las partículas virales en fluido alantoideo de huevos embrionados de pollo SPF infectados con los virus recombinantes y el rLS1, fueron concentrados por ultracentrifugación, y parcialmente purificadas en sucrosa al 25%. El análisis de Western blot fue realizado utilizando los virus parcialmente purificados y los lisados de células infectadas, usando un anticuerpo en conejo específico para la proteína RBD de SARS-CoV-2 y anti-IgG de conejo conjugado con HRP. La proteína beta-actina fue usada como un control de corrida en los lisados celulares. La flecha negra indica la banda de la proteína esperada. Los geles que se muestran se ejecutaron bajo las mismas condiciones. (C) Las células Vero-E6 infectadas con rLS1, rLS1-HN-RBD, y rLS1-S1-F con una MOI de 0.5. Después de 48 hpi, la expresión de las proteínas RBD y S1 fueron detectadas por ensayo de inmunofluorescencia utilizando un anticuerpo en conejo específico para la proteína RBD de SARS-CoV-2, y un Donkey Anti-rabbit IgG H&L-Alexa Fluor 594. Por lo tanto, el NDV fue detectado utilizando un antisuero de pollo específico para NDV, y un a Goat Anti-chicken IgY H&L-Alexa Fluor 488. Los núcleos celulares fueron teñidos con DAPI. Una barra de escala de 50-µm. Aumento de imagen 200x. (D) La detección de las proteínas S1 y RBD sobre la superficie de los virus rLS1-S1-F y rLS1-HN-RBD en células Vero-E6 se realizó en dos experimentos independientes. Las células fueron incubadas con los virus purificados rLS1-HN-RBD y rLS1-S1-F, por 30 min. Subsecuentemente, las células fueron marcadas con un anticuerpo monoclonal en conejo anti-S1 de SARS-CoV-2 como anticuerpo primario, seguido por el anticuerpo secundario goat anti-rabbit IgG Alexa Fluor 488. Las células fueron luego analizadas por citometría de flujo. El porcentaje de células positivas indica la detección de las proteínas S1 o RBD sobre la superficie de los virus en Vero-E6 y se muestra en el diagrama de puntos para el virus rLS1-S1-F y el virus rLS1-HN-RBD; incluyendo controles negativos para cada ensayo determinado por células incubadas con DBPS y el virus rLS1.
Además, el ensayo de neutralización utilizando los sVNT indicó que los sueros de los grupos inmunizados con rLS1-HN-RBD, rLS1-S1-F, y rLS1-HN-RBD/rLS1-S1-F desarrollaron anticuerpos neutralizantes específicos para la proteína RBD a los 15 días post-inmunización y a los 15 días post-refuerzo.
Sin embargo, los sueros colectados de hámsteres vacunados con rLS1-S1-F y rLS1-HN-RBD/rLS1-S1-F mostraron un porcentaje de inhibición del RBD-ACE-2 superior al 50%, mientras que el grupo rLS1-HN-RBD solo mostró una inhibición del 30% hasta 15 días post-refuerzo. Los sueros del grupo control se mantuvieron por debajo del 20% de inhibición hasta 15 días post-refuerzo y no mostró anticuerpos neutralizantes contra la proteína RBD (Fig. 5D).
Figura 3. Estabilidad genética de los virus recombinantes. La estabilidad genética de los virus rLS1-HN-RBD y rLS1-S1-F fue evaluada al tercer y sexto pasaje mediante (A) el análisis de Western blot utilizando un anticuerpo policlonal en conejo específico para la proteína RBD de SARS-CoV-2, y (B) RT-PCR utilizando los cebadores NDV-3LS1-2020-F1 y NDV-3LS1- 2020-R1 para amplificar los insertos completos. P3: tercer pasaje, P6: sexto pasaje. La flecha negra indica el peso molecular esperado.
Un pool de sueros de los hámsteres vacunados con el virus rLS1-S1-F mostró un título alto de reducción de placa en el ensayo de neutralización a los 15 días post-refuerzo, conservando el 100% de su capacidad incluso en diluciones más altas de suero (1/160). La mezcla de las vacunas rLS1-HN-RBD/rLS1-S1-F mostró un título menor de reducción de placa viral (1/40) que rLS1-S1-F. Por otro lado, el rLS1-HN-RBD no tuvo efecto en la reducción de placa (Fig. 5E, F).
Cuantificación de citoquinas por ELISA. Inmunización de hámsteres no indujo un incremento significativo en los niveles de IL-2 o IFNγ en suero, evaluado por ELISA cuantitativo. Aunque no significativa, se observó una tendencia creciente de los niveles de IL-2 con rLS1-HN-RBD (P = 0.55), rLS1-S1-F (P = 0.07) y rLS1-HN-RBD/rLS1-S1-F (P = 0.07). Sin embargo, uno de los individuos vacunados con rLS1-HN-RBD/rLS1-S1-F tuvo altos niveles de producción de IL-2 circulante. Los niveles de IFNγ no fueron incrementados significativamente con rLS1-HN-RBD (P = 0.08), rLS1-S1-F (P = 1.00) y rLS1-HN-RBD/rLS1-S1-F (P = 0.56) (Figure 5 complementaria). Los niveles de IL-4 e IL-10 se mantuvieron por debajo del límite inferior de detección, y los valores de TNFα fueron bajos y detectables solo en suero de animales inmunizados con rLS1-S1-F y rLS1-HN-RBD/rLS1-S1-F (Tabla I Complementaria).
Cuantificación de citoquinas por qPCR. No se observaron diferencias significativas en la expresión génica de citoquinas para cualquiera de las vacunas utilizadas: rLS1-HN-RBD (IFNγ 1.2-fold, P = 1.00, TNFα 1.48-fold, P = 0.16, IL-10 1.7-fold, P = 0.16), rLS1-S1-F (IFNγ 1.16-fold, P = 0.16, TNFα 1.48-fold, P = 0.14, IL-10 P = 0.16) y rLS1-HN-RBD/rLS1- S1-F (IFNγ 0.81-fold, P = 0.48, TNFα 1.12-fold, P = 0.48, IL-10 0.90, P = 0.48). Aunque no significativas, se observan tendencias. (Figura 5 complementaria).
Eficacia de las vacunas contra el desafío del SARS-CoV-2. En los días 5 y 10 posteriores al desafío, no se aisló el SARS-CoV-2 en tejido de pulmón de hámsteres vacunados con rLS1-S1-F y la mezcla de las vacunas rLS1-S1-F/rLS1-HN-RBD. Los animales vacunados y desafiados mostraron un resultado de IFI negativo a los 5 y 10 días post-desafío (dpc). La vacuna rLS1-HN-RBD sola no demostró suficiente capacidad neutralizante para prevenir la infección. En este grupo, los virus pudieron ser aislados a los 5 dpc y confirmados por un IFI positivo en tejido de pulmón (Fig. 6A, Tablas complementarias II, III). Entre los hámsteres no vacunados, el SARS-CoV-2 fue aislado a los 2 y 5 dpc, pero para el día 10 no se pudo detectar ningún virus.
Figura 4. Estabilidad de la vacuna liofilizada de NDV. La expresión de las proteínas S1-F y HN-RBD en células Vero-E6 infectadas con la vacuna liofilizada de NDV fue confirmada a los 1, 30, y 50 días post-liofilización por ensayo de Western blot usando un anticuerpo policlonal en conejo específico para la proteína RBD del SARS-CoV-2. La flecha negra indica la banda de la proteína esperada.
La cuantificación de copias virales por RT-qPCR obtenida de homogeneizados de pulmón de hámster confirmó la presencia de un alto número de copias virales (10.97 ± 0.17 log10) en hámsteres no inmunizados (control) y hámsteres inmunizados con las vacunas rLS1-HN-RBD (10.96 ± 0.24 log10) y rLS1-HN-RBD/rLS1-S1-F (9.36 ± 4.09 log10) a los 2 dpc. Los animales inmunizados con rLS1-S1-F mostraron un número de copias virales significativamente más bajo y (6.92 ± 3.70 log10, P < 0.05). A los 5 dpc, la carga viral del aislado se mantuvo en el grupo control (10.99 ± 0.24 log10) y en hámsteres inmunizados con rLS1-HN-RBD (11.54 ± 0.76 log10), sin embargo, en hámsteres inmunizados con rLS1-S1-F (4.50 ± 1.17 log10, P ≤ 0.0003) y rLS1-HN-RBD/rLS1-S1-F (5.65 ± 1.41 log10, P ≤ 0.0059) la carga viral disminuyó significativamente (Fig. 6B). A los 10 dpc todos los grupos analizados, incluyendo el control, presentó un bajo número de copias virales (3.94 – 4.93 log10) (Fig. 6B), probablemente debido a la presencia de residuos de ARN, ya que no detectamos algún efecto citopático en cultivo de células Vero.
Los cambios histopatológicos de los pulmones de los hámsteres fueron monitoreados durante el desafío con SARS-CoV-2. El grupo no vacunado (control) demostró signos patológicos de la enfermedad, empezando con neumonía intersticial a los 2 dpc, evolucionando en neumonía hemorrágica a los 5 dpc, y terminando en bronconeumonía severa, caracterizado por un engrosamiento de la pared del parénquima, infiltración moderada a severa de células inflamatorias macrófagos y linfocitos, y en la luz bronquiolar, y pérdida de la arquitectura alveolar. Los grupos inmunizados con las vacunas rLS1-S1-F y rLS1-HN-RBD/rLS1-S1-F no mostraron lesiones visibles, manteniendo características del tejido de pulmón similar a aquellas vistas en el grupo no desafiado en cada uno de los puntos evaluados. Aunque el grupo inmunizado con rLS1-HN-RBD desarrolló cambios patológicos en el pulmón, aquellos fueron más leves que aquellos vistos en el grupo no vacunado, mostrando neumonía moderada 2 dpc y terminando en una neumonía moderada a severa a los 10 días (Fig. 6C).
Los animales no vacunados que pertenecen al grupo control no desafiado mostraron una variación porcentual promedio en peso corporal de no más de 3% durante los 10 días de análisis. Los animales no vacunados que fueron infectados con SARS-CoV-2 mostraron una pérdida de peso significativa, con una reducción promedio en el peso corporal de más del 5% en el día 5 y más del 10-25% en el día 10 dpc. No hubo diferencias estadísticamente significativas en el peso corporal entre los hámsteres vacunados y los no vacunados y no expuestos los días 2 y 5, pero no hubo diferencia significativa entre el grupo control desafiado y el grupo vacunado con rLS1-HN-RBD/rLS1-S1-F en el día 10 (Fig. 7A).
Las evaluaciones de velocidad, aceleración y desplazamiento de los animales confirmaron que los hámsteres vacunados con la mezcla de las vacunas rLS1-S1-F/rLS1-HN-RBD fueron los más activos en el día 5 (momento en que aparecen los síntomas) y 10 (momento en el cual los síntomas desaparecen) post-desafío. Los animales vacunados con la mezcla de las vacunas rLS1-S1-F/rLS1-HN-RBD mostró la mayor recuperación en términos de movilidad (Fig. 7B–D).
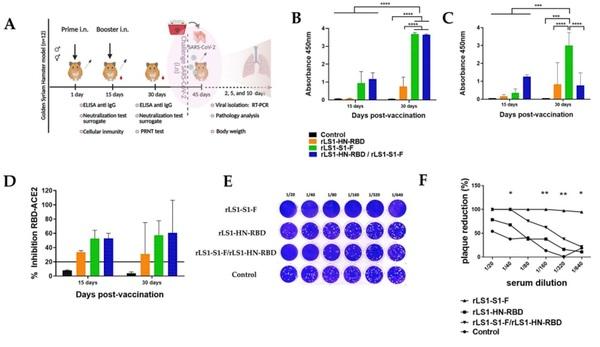
Figura 5. La vacuna intranasal induce anticuerpos específicos contra el RBD, la subunidad S1 y anticuerpos neutralizantes contra el SARS-CoV-2 en hámsteres. (A) Régimen de inmunización. Para evaluar la inmunogenicidad de las vacunas de NDV, hámsteres Sirios Dorados (hembras y machos) de cinco semanas de edad fueron utilizados en este estudio. Los hámsteres fueron divididos al azar en cuatro grupos. Los hámsteres fueron vacunados por vía intranasal con la vacuna viva de NDV, después de un régimen de refuerzo con un intervalo de dos semanas. El grupo 1 recibió la rLS1-HN-RBD (n = 12), el grupo 2 recibió la rLS1-S1-F (n = 12), el grupo 3 recibió la mezcla de ambas vacunas rLS1-HN-RBD/rLS1-S1-F (n = 12), y el grupo 4 fue el grupo control no-vacunado (n = 12) Una inmunización de refuerzo con la misma concentración de cada vacuna se aplicó en todos los grupos vacunados en la segunda semana. (B) El ensayo de ELISA para medir anticuerpos IgG específicos para RBD de SARS-CoV-2 en suero, y (C) anticuerpos IgG específicos a la subunidad S1 en suero. Los sueros de hámsteres en el pre-refuerzo y 15 días después del refuerzo fueron evaluados. La proteína recombinante purificada RBD de SARS-CoV-2 fue usada para el ELISA. El corte se fijó en 0.06. (D). Los hámsteres inmunizados fueron sangrados pre-refuerzo y 15 días después del refuerzo. Todos los sueros fueron aislados por centrifugación a baja velocidad. Las muestras de suero fueron procesadas para evaluar los títulos de anticuerpos neutralizantes contra la proteína RBD del SARS-CoV-2 utilizando los sVNT. El corte positivo y negativo para la detección de anticuerpos neutralizantes contra el SARS-CoV-2 fueron interpretados como la tasa de inhibición. La interpretación del corte de los resultados: resultado positivo ≥20% (anticuerpos neutralizantes detectados), resultado negativo < 20% (anticuerpos neutralizantes no detectables). (E) La figura muestra los títulos de PRNT de SARS-CoV-2 en células Vero con muestras pool de sueros de hámsteres inmunizados con rLS1-S1-F, rLS1-HN-RBD, y la combinación de ambas vacunas. (F) Las curvas de reducción de placas (%) utilizaron sueros combinados de diferentes grupos de hámsteres. Two-way ANOVA and Tukey’s post hoc fueron realizados. *P < 0.05; **P < 0.01; ***P < 0.001; ****P < 0.0001.
Discusión
En este estudio, nosotros desarrollamos dos candidatos a vacunas nasales vectorizadas de NDV recombinante que expresaron los antígenos de la subunidad S1 y RBD del SARS-CoV-2. Tres diferentes formulaciones de vacuna intranasal fueron evaluados: un NDV que expresó el RBD (rLS1-HN-RBD), un NDV que expresó la subunidad S1 (rLS1-S1-F), y una mezcla de ambas vacunas vectorizadas de NDV que expresaron el RBD y la subunidad S1 (rLS1-HN-RBD/rLS1-S1-F). La evaluación de la eficacia de la protección en hámsteres demostró que la inmunidad y protección más fuerte fueron provocadas por la vacuna vectorizada de NDV que expresó la subunidad S1, seguido por la combinación de ambas vacunas de NDV que expresaron la subunidad S1 y RBD, evidenciado por la ausencia de daños en las células pulmonares, viabilidad viral y carga viral considerablemente reducida, en comparación con el grupo no vacunado. Sorprendentemente, la vacuna que expresó el RBD no demostró alguna evidencia de protección. Hay al menos dos posibles explicaciones que no son necesariamente excluyentes entre sí. En primer lugar, la subunidad S1 puede incluir epítopes protectores además de los presentes en el RBD, para los cuales los anticuerpos neutralizantes podrían contribuir a interferir en la interacción S-ACE-2 a una distancia a nivel estérico. Esta posibilidad es apoyada por un previo estudio que demostró que la subunidad S1 contiene epítopes neutralizantes que no fueron encontradas en el RBD7. En segundo lugar, el RBD presente en la vacuna rLS1-HN-RBD no alcanzó un plegamiento suficientemente cercano a la estructura 3D del RBD biológicamente activo cuando se encuentra en la partícula viral del SARS-CoV-2. Además, es posible que epítopes B conformacionales desempeñen un rol más importante que epítopes T lineales en la respuesta inmune protectiva. Recientes estudios que evaluaron una similar vacuna candidata vectorizada de NDV presentando el antígeno S completo (subunidad S1 y S2), demostraron protección de hámsteres en un ensayo de desafío38, 39.
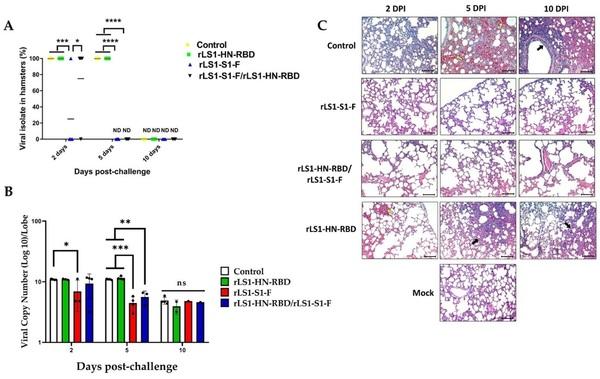
Figura 6. Eficacia de las vacunas vivas de NDV contra la infección por SARS-CoV-2 en hámsteres. Grupos de hámster Sirio Dorado vacunados con rLS1-S1-F, rLS1-HN-RBD, la mezcla rLS1-S1-F/rLS1-HN-RBD, o no vacunados (control positivo) fueron desafiados 30 días después del refuerzo con SARS-CoV-2, junto con el grupo mock que no estaba vacunado ni desafiado (control negativo). (A) Aislamiento viral (% de animales que fueron positivos para SARS-CoV-2) del pulmón de cada grupo de hámsteres (n = 4) en los días 2, 5 y 10 posteriores al desafío. Se realizaron ANOVA de dos vías y post hoc de Tukey. *P < 0,05; **P < 0,01; ***P < 0,001; ****P < 0,0001. (B) Detección por RT-qPCR de SARS-CoV-2 en sobrenadante de cultivo de células Vero, inoculadas con homogeneizados de pulmón de grupos de hámster inmunizados y desafiados. Los datos muestran una diferencia significativa en el número de copias virales *P < 0,05; **P < 0,01; ***P < 0,001; ****P < 0,0001. ns, no significativo. (C) La histopatología pulmonar de cada grupo de hámster (n = 4) se sacrificó en diferentes días posteriores a la infección (dpi). Las áreas hemorrágicas e infiltradas se indican con una flecha amarilla y negra, respectivamente. Amplitud de imagen: 20×. Barra de escala: 100 µm.
Cabe señalar, hámsteres de grupos no vacunados que fueron desafiados con SARS-CoV-2, presentaron un cultivo viral positivo a los 2 y 5 días post-desafío (dpc), y fueron negativos al día 10. Estos resultados coinciden con estudios previos en el cual demostraron que la carga viral es reducida a niveles indetectable a los 8 días post-infección en modelo de hamster41, 42. Junto con los resultados in vitro, el IFI también confirmó que la vacuna rLS1-S1-F, seguido por la mezcla de las vacunas rLS1-HN-RBD/rLS1-S1-F, provocaron una respuesta protectiva más fuerte. Esto fue evidenciado por la ausencia de un aislamiento viral de los tejidos pulmonares a los 5 dpc. A los 2 días post-desafío, solo la mitad de los animales vacunados con la vacuna rLS1-S1-F presentó un aislamiento viral, y estos presentaron un IFI negativo. Esto sugiere que probablemente la neutralización del SARS-CoV-2 está ocurriendo entre los días 2 y 3 post-desafío en el modelo de hámster.
La evaluación histopatológica de los pulmones de los hámsteres no vacunados después del desafío mostró signos patológicos severos de la enfermedad, comenzando con una neumonía intersticial a los 2 dpc, y culminando con una bronconeumonía severa, así también como la perdida de la arquitectura alveolar. En comparación, los grupos inmunizados con la vacuna rLS1-S1-F y la combinación de las vacunas rLS1-HN-RBD/rLS1-S1-F fueron protegidos, demostrando capilares alveolares y respiratorios casi intactos, sin evidencia de una reacción inflamatoria. Por el contrario, los animales vacunados con rLS1-HN-RBD desarrollaron lesiones patológicas, aunque con menor grado hemorrágico comparado al grupo control no vacunado, con neumonía a los 2 dpc y culminando con una neumonía moderada a severa a los 10 dpc. Este es un resultado alentador que necesita ser verificado ensayos clínicos en humanos.
Después del desafío intranasal, se evaluó la presencia y viabilidad del SARS-CoV-2 en los pulmones de los animales, a los días 2, 5 y 10, momento en el cual los animales fueron sacrificados y se realizó la necropsia. Al día 5 post-desafío, los animales vacunados con rLS1-S1-F y rLS1-S1-F/rLS1-HN-RBD no mostraron la presencia del virus viable en los pulmones, comparados con los grupos de animales control y vacunados con rLS1-HN-RBD. Sin embargo, ya que no medimos la diseminación viral de la cavidad nasal, no podemos especular que la diseminación viral pudiera detenerse en los animales vacunados. Se necesitan más estudios para comprender mejor este hecho.
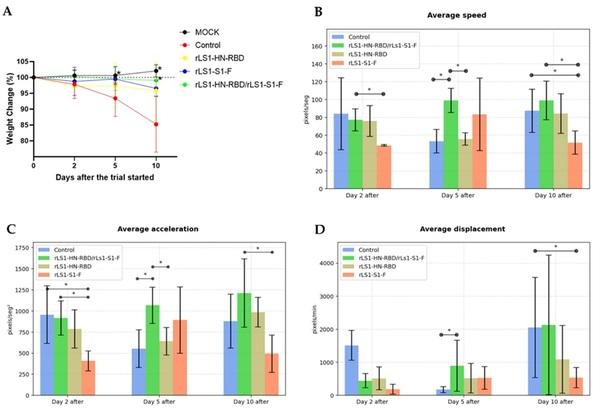
Figura 7. Análisis del peso corporal y la movilidad de los hámsteres Sirios Dorados desafiados con SARS-CoV-2. (A) Cambios en el peso corporal (porcentaje de cambio de peso en comparación con el día 0) de hámsteres inoculados con SARS-CoV-2 y grupo mock, en los días 2, 5 y 10 posteriores al desafío. Los resultados de la evaluación de movilidad muestran (B) velocidad promedio, (C) aceleración promedio y (D) desplazamiento promedio. Se muestra la media ± SD. Los asteriscos indican que los resultados fueron estadísticamente significativos en comparación con el grupo de control (*P < 0,05).
Para evaluar la respuesta inmune, dos semanas antes del desafío se recolectaron muestras de sangre de los animales vacunados. Fue importante reducir el estrés en los animales que puede temporalmente afectar la respuesta inmune en el momento del desafío. Aunque otros estudios usan un tiempo más corto. Nosotros conocemos que dos semanas es más que suficiente para tener los animales recuperados del estrés causado durante el sangrado. Sin embargo, reconocemos esta limitación en nuestro estudio, ya que no tenemos una medida del nivel de inmunidad en el momento exacto del desafío y además nuestras medidas no reflejan completamente la respuesta inmune en ese momento.
Cuando medimos anticuerpos específicos de RBD y S1, nosotros observamos que los animales vacunados con rLS1-HN-RBD y rLS1-S1-F, demostraron un refuerzo del día 15 hacia 30 post-vacunación. Sin embargo, en los animales vacunados con la mezcla de ambas vacunas rLS1-HN-RBD/rLS1-S1-F, hubo un refuerzo específico de anticuerpos RBD, pero hubo una falta de refuerzo con respecto a anticuerpos específicos para S1. Desafortunadamente, nosotros no tenemos una clara explicación para este resultado. Nosotros podemos especular que la falta de aumento de anticuerpos para S1 entre animales vacunados con rLS1-HN-RBD/rLS1-S1-F, podría ser asociado con una potencial interacción entre las dos vacunas, en la cual la mezcla de ambas, da como resultado la dominancia del antígeno RBD, que resulta de una falta de refuerzo del anticuerpo no RBD (es decir anticuerpos S1 diferentes a los anticuerpos RBD). Se requieren más estudios para aclarar este tema.
En este reporte, evaluamos el patrón de movilidad de los animales como un indicador objetivo y cuantitativo de su estado de salud. Usando videos grabados y herramientas computacionales para el análisis de patrones y seguimiento digital, medimos el promedio de velocidad, el promedio de la aceleración, y el promedio de desplazamiento de los animales en sus jaulas a los 2, 5, y 10 dpc. Los resultados mostraron que al 5 dpc, los animales del grupo control no vacunados tenían un desplazamiento, velocidad, y aceleración medios reducidos en comparación con animales vacunados. Esta marcada diferencia no se observó claramente a los 2 dpc, lo que confirma que, en ese momento, los animales infectados pueden haber sido relativamente asintomáticos. Este resultado concuerda con otros hallazgos que muestran la eliminación del virus a los 10 dpc41,42.
Un resultado prometedor que sugiere una fuerte protección de la vacuna rLS1-S1-F es la capacidad neutralización de los sueros de los hámsteres vacunados contra el virus SARS-COV-2 a los 30 días después de la inmunización en el PRNT. Por el contrario, animales vacunados con rLS1-HN-RBD no mostraron una capacidad relevante de sero-neutralización.
Este resultado es consistente con los hallazgos similares encontrados en un estudio reciente que muestran que una vacuna de SARS-CoV-2 atenuada viva intranasal vectorizada con NDV indujo un alto nivel de inmunidad humoral en un tiempo relativamente corto, así como la producción de IgA de la mucosa39. Una respuesta anticuerpos mucosa es considerada importante contra las infecciones que utilizan el tracto respiratorio como vía de entrada, lo que convierte a la mucosa del tracto respiratorio en la primea línea de defensa contra esta infección.
Actualmente, estamos completando un ensayo para evaluar la presencia de anticuerpos IgA de la mucosa anti S1/RBD en ratones vacunados con rLS1-S1-F. Los datos preliminares muestran evidencia de producción de IgA en células de médula ósea (datos no mostrados). Por lo tanto, se requieren más estudios para confirmar este hallazgo.
Otros estudios recientes evaluaron vacunas similares vectorizadas de NDV en hámsteres y ratones. A diferencia de las vacunas descritas aquí, otras usando la proteína Spike S completa37-40.
En esos reportes, la vacuna vectorizada de NDV se administró por vía nasal y por la ruta intramuscular. Estas vacunas provocaron una alta respuesta inmune, con anticuerpos específicos IgG e IgA. La evaluación de la eficacia con el desafío del SARS-CoV-2 después de la inmunización confirmó que no se encontró infección, inflamación, ni ninguna lesión patológica en los tejidos pulmonares. Se redujo la carga viral en los pulmones de los animales desafiados, en comparación con el control, así como la excreción del virus en los cornetes o turbinas nasales y los pulmones en hámsteres y ratones. El peso corporal y la movilidad física no se vieron alterados37-40.
Nuestros resultados que evalúan la seguridad y la eficacia de la vacuna propuesta son muy similares hacia los otros descritos previamente en la literatura. Aunque se reconoce que la ruta nasal tiene ventajas, en cuanto a la obtención de anticuerpos IgA en las mucosas, si la respuesta inmunitaria provocada es mucho menor, la ventaja puede perderse.
El NDV atenuado se usa más comúnmente como vacuna en todo el mundo contra la infección de ND, con dosis infecciosa de cultivo de tejido medio (TCID50) que oscilan entre 104 y 105, cuales son administradas hacia animales por ruta oral, nasal ocular o polverización43-45. Dado que el NDV está restringido al tracto respiratorio, no se detecta en otros órganos ni en la sangre, por lo que se espera que la replicación en humanos sea espera que la replicación en humanos sea limitada y benigna46-48. En raras ocasiones, se ha observado que los seres humanos expuestos al NDV mesogénico desarrollan conjuntivitis, laringitis, o síntomas similares a los de la gripe que desaparecen dentro de 1 a 2 días49. Por lo tanto, los vacunadores y cuidadores que están expuestos frecuentemente al NDV no han reportado ningún efecto secundario hasta la fecha50-52. Es de destacar que las pruebas de seguridad y toxicidad se realizaron con dosis que no se produjeron bajo la estricta certificación de buenas prácticas de fabricación (GMP), lo que significa que cualquier efecto adverso será sobreestimado. Dado que los ensayos se realizaron con dosis de la vacuna que no son GMP, significa que cualquier los eventos adversos se sobreestimará y se esperarían tasas más bajas en una evaluación de la vacuna en dosis producidas bajo condiciones GMP.
El desarrollo de la vacuna candidata vectorizada de NDV que se presenta aquí incluye un paso final de liofilización. Esto le confiere estabilidad: la vacuna puede conservarse a 4°C durante varios meses sin perder más del 5% de su actividad, similar a otras vacunas liofilizadas53. El hecho de que la vacuna rLS1-S1-F se administre por vía nasal le da una ventaja adicional al simplificar los requisitos logísticos para las inmunizaciones. No hay necesidad de un ejército de vacunadores ni de un gran número de jeringas. Es posible que las dosis de la vacuna rLS1-S1-F puedan administrarse en viales de 500 dosis con un sistema de dispensador manual activo por gatillo que utiliza dispositivos desechables individuales. De esta manera, se podría administrar una vacuna nasal en campañas a gran escala en comunidades rurales remotas con gran facilidad
La pandemia de COVID-19 tiene el potencial de volverse endémica, y si esto sucediera, las vacunas deben administrarse de forma rutinaria con cierta frecuencia54,55. El SARS-CoV-2 en los últimos meses ha mostrado un intenso nivel de mutaciones en los antígenos virales utilizados en las diversas vacunas actualmente disponibles. Estas mutaciones han sido seleccionados de forma natural, ante la presión inmunológica que ejercen los individuos curados de COVID-19. Por lo tanto, ahora se han identificado mutaciones que pueden dar al virus la capacidad de escapar de la inmunidad adquirida. (resistencia inmune), y esto puede conducir a un aumento en los casos de reinfección por SARS-CoV-2. Estas mismas mutaciones seleccionadas naturalmente también han sido seleccionadas in vitro, bajo presiones de selección inmunológica usando anticuerpos neutralizantes de suero convaleciente. Esto sugiere una gran posibilidad de que las vacunas basadas en el antígeno S1 circulante en el principio de 2020 pueden verse comprometidos hasta cierto punto en su nivel de efectividad contra las nuevas variantes del SARS-CoV-2.
Por lo tanto, la forma más eficiente de enfrentar la pandemia de COVID-19 será utilizar vacunas personalizadas para áreas geográficas específicas, en función de la distribución de variantes circulantes durante un período de tiempo determinado, y que puedan ser producidas y administradas de manera oportuna. La vacuna de la subunidad S1 puede actualizarse y llevar un antígeno vacunal correspondiente a una cepa más relevante en un tiempo relativamente corto.
El NDV se puede transformar dentro de 30 a 45 días y generar un criobanco para comenzar a producir lotes de vacunas actualizadas. Por lo tanto, es importante contar con programas de vigilancia epidemiológica permanente para identificar cualquier variación en la distribución de las cepas circulantes en una región de interés.
En conclusión, nosotros hemos demostrado que el candidato a vacuna de la subunidad S1 se muestra prometedor en estudios preclínicos. Esta vacuna candidata demostró ser segura e inmunogénica, y proporcionó una fuerte protección contra el desafío de SARS-COV-2. Ahora se necesitan ensayos clínicos para evaluar su seguridad y eficacia en humanos.
Métodos
Declaración de ética. La investigación con animales se realizó siguiendo las directrices y reglamentos pertinentes. Todos los protocolos fueron aprobados por el comité de bioética de la Universidad Nacional Hermilio Valdizán, Huánuco, Perú. El estudio se llevó a cabo en conformidad con las directrices de ARRIVE.
El virus SARS-CoV-2 (28549) utilizado en el desafío fue proporcionado por el Instituto Nacional de Salud (INS), Lima, Perú, de acuerdo con las pautas y reglamentos apropiados. El aislamiento del virus SARS-CoV-2 fue aprobado por la Dirección General del Centro de Salud Pública del INS.
Animales. Cien hámsteres Sirios Dorados (Mesocricetus auratus) machos y hembras de 4-5 semanas de edad, fueron obtenidos del Instituto Nacional de Salud (INS) del Perú. Para el ensayo in vivo, todos los hámsteres fueron transferidos y aclimatados en instalaciones de bioseguridad animal de nivel 3 (BSL-3) durante 1 semana. Ellos fueron vacunados con la vacuna contra SARS-CoV-2 vectorizada con NDV y posteriormente desafiados con SARS-CoV-2.
Desarrollo y caracterización de NDV recombinante que expresa antígenos RBD y S1 del SARS-CoV-2. Cultivo celular.Células de riñón de mono verde africano, clon E6 (Vero-E6, ATCC® CRL-1586TM) y células DF-1 (derivadas de fibroblastos de pollo), fueron mantenidas en medio Eagle modificado por Dulbecco (DMEM) suplementado con 5% de suero fetal bovino inactivado por calor (SFB) (HyCloneTM GE Healthcare Life Science, USA). Las células Vero (Vero 81, ATCC® CCL-81TM) fueron cultivadas en el medio esencial mínimo de Eagle (EMEM) suplementado con SFB al 10%, 100IU/ml de penicilina y 100µg/ml de estreptomicina. Todas las líneas celulares fueron cultivadas a 37°C en una atmósfera de 5% de CO2.
Desarrollo y caracterización de NDV recombinante que expresa antígenos RBD y S1 del SARS-CoV-2. Cultivo celular.Células de riñón de mono verde africano, clon E6 (Vero-E6, ATCC® CRL-1586TM) y células DF-1 (derivadas de fibroblastos de pollo), fueron mantenidas en medio Eagle modificado por Dulbecco (DMEM) suplementado con 5% de suero fetal bovino inactivado por calor (SFB) (HyCloneTM GE Healthcare Life Science, USA). Las células Vero (Vero 81, ATCC® CCL-81TM) fueron cultivadas en el medio esencial mínimo de Eagle (EMEM) suplementado con SFB al 10%, 100IU/ml de penicilina y 100µg/ml de estreptomicina. Todas las líneas celulares fueron cultivadas a 37°C en una atmósfera de 5% de CO2.
Las secuencias genéticas de RBD y de la subunidad S1 de la proteína S que corresponde a la cepa del SARS-CoV-2 Wuhan-Hu-1 aislada en China (No. de acceso de GenBank MN908947.3). En el caso de S1, debido al mayor tamaño de su secuencia, para facilitar la replicación y la producción de la vacuna en huevos embrionados de pollo, el codón utilizado fue optimizado para Gallus gallus.
Para mejorar la incorporación de RBD y S1 en el virión NDV, nosotros diseñamos dos casetes. Primero el casete transcripcional HN-RBD (1013 nt) que contiene secuencias génicas de RBD (636 nt), seguido del dominio transmembrana completo (TM), y cola citoplasmática (CT) del gen de la hemaglutinina-neuraminidasa (HN) del NDV. En segundo lugar, el casete transcripcional S1-F (2441 nt), el cual contiene la secuencia genética de la subunidad S1 (2043 nt), tomado del gene S (3822 nt). Esta secuencia fue fusionada con el TM y CT del gen de fusión (F) (Fig. 8A). Estas secuencias génicas TM y CT de los genes HN y F se obtuvieron del plásmido pFLC-LS1. Ambos casetes trasncripcionales fueron flanqueados con señales trasncripcionales específicas de gen end (GE) y de gen start (GS) del genoma del paramyxovirus57. Además, estos casetes, flanqueados con sitios de restricción de BbvCI, fueron químicamente sintetizados y posteriormente clonados en el plásmido pUC57 por GenScript. (Piscataway, NJ, USA). Estos plásmidos se purificaron y el ADN fue extraído con el Kit de QIAGEN Plasmid Midi (100), de acuerdo con las instrucciones del fabricante.
El plásmido pFLC-LS1, que contenía un sitio BbvCI único, fue digerido con la enzima BbvCI para obtener le plásmido linealizado. Los casetes trasncripcionales HN-RBD y S1-F fue digerido con la misma enzima e insertado en la unión P/M del pFLC-LS1 para expresarse como ARNm. Los plásmidos resultantes se designaron como pFLC-LS1-HN-RBD (20,315 nt) y pFLC-LS1-S1-F (21,743 nt) (Fig. 8B).
Recuperación del virus rLS1-HN-RBD y rLS1-S1-F. Los virusrLS1-HN-RBD y rLS1-S1-F fueron recuperados por co-trasnfección de ADN complementario (ADNc) de cada constructor, pFLC-LS1-S1-F o pFLC-LS1-HN-RBD respectivamente, como se describió previamente56. Los virus recuperados fueron inoculados dentro de las cavidades alantoideas de huevos embrionados de pollo libres de patógenos específicos (SPF) de 9 días de edad (Charles River, Norwich, Connecticut, EE. UU.). Después de una incubación por cuatro días a 37 °C, el fluido alantoideo que contiene el virus recuperado fue recolectado, clarificado, alicuotado y almacenados a -80°C. Se confirmó la presencia y recuperación mediante el ensayo de hemaglutinación (HA) utilizando glóbulos rojos de pollo al 1%. La identidad de los virus recombinantes fue confirmada por reacción en cadena de la polimerasa con transcripción reversa (RT-PCR) y por secuenciación de Sanger, como se ha descrito anteriormente48.
Ensayo de inmunofluorescencia indirecta (IFI). Para examinar la expresión de las proteínas RBD y subunidad S1 del SARS-CoV-2, se infectaron células Vero-E6 con los virus recombinantes rLS1-HN-RBD, rLS1-S1-F y rLS1 con una multiplicidad de infección (MOI) de 0.5. Después de 48 horas post-infección, las células fueron fijadas con paraformaldehído al 4% por 25 minutos (min), y luego la monocapa se lavó tres veces con solución salina tamponada con fosfato de Dulbecco (DPBS) y se permeabilizó con Tritón 0.1% X-100 durante 15 min a temperatura ambiente (RT). Después del lavado de las células con DPBS, la monocapa se incubó con el anticuerpo policlonal de conejo específico para la proteína SARS-CoV-2-RBD (1:200) (Sino Biological, Beijing, China), y un antisuero de pollo específico para el virus de la enfermedad de Newcastle (1:200) (Charles River, Norwich, CT, USA) por 1.5 h a temperatura ambiente. Posteriormente, la monocapa se incubó con Donkey anti-Rabbit IgG H&L-Alexa Fluor® 594 (1:250) y Goat Anti-Chicken IgY H&L-Alexa Fluor® 488 (1:1000) (Abcam, Cambridge, MA, USA) por 60 min a RT. Finalmente, las células se revelaron con 4', 6-diamidino-2-fenilindol (DAPI) durante 5 min y se observaron con un microscopio de fluorescencia ObserverA1. (Carl Zeiss, Alemania). Las imágenes digitales se tomaron con un aumento de 400× y se procesaron con la cámara AxioCam MRc5 (Carl Zeiss, Jena, Alemania).
Análisis de Western Blot. Para evaluar la expresión de las proteínas RBD y la subunidad S1 de SARS-CoV-2, las células Vero E-6 fueron infectadas con los virus recombinantes mencionados anteriormente con una MOI de 1.0. A las 48 horas post-infección, las células se recolectaron, lisaron y analizaron mediante Western blot. Adicionalmente, para verificar la incorporación de las proteínas RBD y S1 en los virus rLS1-HN-RBD y rLS1-S1-F, las partículas virales de fluido alantoideo de huevos embrionados de pollo SPF infectados con los virus recombinantes y rLS1, fueron concentradas por ultracentrifugación (Ultracentrífuga, Beckman Coulter) a 18.000 revoluciones por minuto (rpm) a 4°C, y parcialmente purificado en una gradiente de sucrosa al 25%. El análisis de western blot se llevó a cabo usando virus parcialmente purificados de fluido alantoideo y lisado de células infectadas, utilizando un anticuerpo policlonal de conejo específico para la proteína RBD del SARS-CoV-2 (Sino Biological, Beijing, China) (2/5000) como anticuerpo primario y anti-IgG de conejo conjugado con HRP (GenScript, Piscataway, NJ, EE. UU.) (2/5000) como anticuerpo secundario. Para la detección de beta-actina como control de carga de lisado de células se usó el anticuerpo monoclonal de ratón contra beta actina Abcam, Cambridge, MA, EE. UU.) (5/5000) como anticuerpo primario y goat anti-mouse IgG conjugado con HRP (GenScript, Piscataway, Nueva Jersey, EE. UU.) (3/5000) como el anticuerpo secundario. La expresión de la proteína se visualizó con una cámara CCD del sistema de imágenes Azure c600 (Azure Biosystems, Inc., Dublin, OH, USA.).
Figura 8. La estrategia utilizada para la generación de los NDV recombinantes que expresan RBD y S1 del SARS-CoV-2. (A) La representación esquemática de la estrategia de construcción de los NDV recombinantes. Se diseñaron dos casetes transcripcionales para expresar RBD y S1: 1) HN-RBD se fusionó con el dominio transmembrana completo (TM) y el tallo citoplasmático (CT) del gen de la hemaglutinina-neuraminidasa (HN), 2) S1 fue fusionado con el TM/CT del gen de fusión (F) del pFLC-LS1 de longitud completa. (B) El completo anti-genoma del clon LaSota de NDV (pFLC-LS1) se utilizó como clon, pFLC-LS1-HN-RBD y pFLC-LS1-S1-F fueron generados a partir de casetes que expresan genes de RBD y S1 insertados en el genoma de NDV bajo el control de las señales transcripcionales gen end (GE) y del gen start (GS). Los nombres, la posición y dirección de los cebadores utilizados se muestran con flechas (negras) que indican el tamaño de los productos.
Detección de la proteína RBD y de la subunidad S1 en la superficie viral por citometría de flujo. Para determinar la presencia de RBD en la superficie viral de rLS1-HN-RBD, y la presencia de la subunidad S1 en virus rLS1-S1-F, las partículas virales fueron purificadas con una gradiente de sucrosa al 25 %. Las células Vero-E6 fueron recolectadas y lavadas con DPBS con SFB al 5%. Aproximadamente, 1×106 células fueron bloqueadas con DPBS con 5% de suero de ratón naive por 30 min a 37 °C. Luego, las células fueron incubadas con los virus purificados rLS1 (0.36 mg/mL), rLS1-S1-F (0.09 mg/mL) y rLS1-HN-RBD (0.2 mg/mL) por 30 min a 37 °C. Para remover las partículas residuales no unidas a Vero-E6, las células fueron lavadas con DPBS y SFB al 5% dos veces. Seguidamente, las células fueron incubadas con anticuerpo monoclonal de conejo anti-SARS-CoV-2 S1 (1:200) (Sino Biological, Beijing, China) como anticuerpo primario por 1 h a 37 °C, seguida Goat anti-rabbit IgG Alexa Fluor® 488 (1:200) como anticuerpo secundario.
Finalmente, las células fueron analizadas por citómetro de flujo FACS Canto II (BD Biosciences, USA). Los datos obtenidos fueron analizados usando el software FlowJo v.10.6 (BD Biosciences, USA), donde el porcentaje de células positivas fue tomado para indicar detección de RBD o la subunidad S1 del SARS-CoV-2 en la superficie viral del virus unido a Vero-E6.
Detección de genes del RBD y de la subunidad S1 mediante RT-PCR. Para la detección del de los virus recombinantes rLS1-HN-RBD y el rLS1-S1-F, se extrajo el ARN viral a partir de fluido alantoideo stocks usando el Kit QIAamp MinElute Virus Spin. El ADNc se produjo a partir de ARN usando el kit ProtoScript II cDNA Synthesis (New England Biolabs, USA), según las instrucciones del fabricante. El ADNc se amplificó usando el ADN polimerasa de alta fidelidad Master Mix Q5 (New England Biolabs, USA), con los cebadoresNDV-3LS1- 2020-F1 (5′-GATCATGTCACGCCCAATGC-3′) y NDV-3LS1-2020-R1 (5′-GCATCGCAGCGGAAAGTAAC-3′) para amplificar los insertos completos. El protocolo de ciclo térmico comprendió un ciclo de desnaturalización inicial a 98°C por 30 segundos (s), seguido por 35 ciclos de 98°C por 10 s, 72 °C por 20 s, 72 °C por 30 s para la detección del rLS1-HN-RBD, y 40 s para la detección de rLS1-S1-F. La etapa de extensión final fue a 72°C por 2 min.
Propiedades de la replicación in vitro de los virus rLS1-HN-RBD y rLS1-S1-F, ensayo en placa y patogenicidad. Se comparóla infectividad y las propiedades de crecimiento entre los virus rLS1-HN-RBD, rLS1-S1-F, y rLS1. Las células DF-1 fueron sembradas a una confluencia del 70 % en placas de 12 pocillos e infectadas con los virus rLS1-HN-RBD, rLS1-S1-F, y rLS1con una MOI de 0.5. Las células fueron mantenidas con DMEM que contenían 1 % de SFB y 5 % de fluido alantoideo e incubadas a 37°C con 5 % de CO2. Los sobrenadantes de las células infectadas fueron colectados a las 12, 24, 36, 48, 60, y 72 horas post-infección y conservadas a -80 °C. Los títulos de los sobrenadantes recolectados fueron determinados usando ensayo en placa, como se ha descrito previamente56. Estos experimentos fueron repetidos 3 veces en tiempos independientes. Además, la morfología y el tamaño de las placas de los dos virus recombinantes fueron comparados con aquellos formados con infección por rLS1. Para determinar la patogenicidad, los virus fueron evaluados mediante los ensayos de tiempo medio de muerte (MDT) y el índice de patogenicidad intracerebral (IPIC) en huevos embrionados de pollo SPF de 10 días de edad y pollos SPF de un día (Charles River Avian Vaccine Services, Norwich, CT, USA), respectivamente, usando procedimientos estándar58.
Estabilidad genética de los virus rLS1-HN-RBD y rLS1-S1-F. La estabilidad genética de los virus recombinantes en múltiples pasajes fue evaluada en huevos embrionados de pollo SPF de 9 días de edad, se extrajo el ARN viral a partir de virus purificados del tercer y sexto pasaje, y la presencia de los genes insertados fueron confirmados por RT-PCR utilizando cebadores específicos. La expresión de los insertos de RBD y la subunidad S1 del SARS-CoV-2 también fueron evaluados usando virus purificados del tercer y sexto pasajemediante Western blot.
Preparación y estabilidad de la vacuna liofilizada. Para evaluar la estabilidad de la vacuna liofilizada, los virus rLS1-RBD-HN y rLS1-S1-F fueron inoculados de forma separada en cavidades alantoideas de huevos embrionados de pollo SPF de 9 a 11 días de edad. Luego de 4 días de incubación a 37°C, los fluidos alantoideos fueron recuperados, clarificados, y filtrados utilizando filtros de 0.22 µm. La presencia de los virus en fluido alantoideo fue detectado y confirmado por HA. Finalmente, el fluido alantoideo que contenía los virus rLS1-RBD-HN, rLS1-S1-F, y la combinación de ambos (equimolar 1:1), fueron dispensados en viales (2 mL/vial) y liofilizados usando un liofilizador MX5356 (Millrock Technology). La vacuna liofilizada de la combinación de ambos virus rLS1-RBD-HN y rLS1-S1-F fueron conservadas a 4°C y fueron evaluadas mediante ensayo de placa, HA, y ensayos de Western blot a los 1, 30, y 50 días luego de la liofilización. Las vacunas liofilizadas fueron usadas en las siguientes pruebas in vivo en hámsteres.
Inmunogenicidad en hámsteres. Cuarenta y ocho hámsteres Sirios Dorados, con pesos entre 120 y 140 g, fueron divididos en 4 grupos (por grupo n = 12): grupo 1 (rLS1-HN-RBD), grupo 2 (rLS1-S1-F), grupo 3 (rLS1-HN-RBD/rLS1-S1-F), y el grupo 4 de control no vacunado. Fueron inmunizados por vía intranasal con 5×106 unidades formadoras de placa (PFUs)/hámster (volumen de 40 µl) siguiendo un régimen de refuerzocon un intervalo de dos semanas. Los hámsteres inmunizados fueron inmediatamente desangrados antes del refuerzo y quince días post-refuerzo (en los días 15 y 30, respectivamente), para medir el anticuerpo IgG sérico específico para RBD y S de SARS-Cov-2 mediante ensayo por inmunoabsorción ligado a enzima indirecto (ELISA), títulos de anticuerpos neutralizantes (nAbs) utilizando una Prueba de Neutralización de Virus sustituta (sVNT), y para realizar la Prueba de Neutralización de Reducción de Placa (PRNT) contra el virus SARS-CoV-2 (Fig. 5A).
Ensayo de inmunoabsorción ligado a enzima indirecto (ELISA) para determinar los niveles de IgG en suero. Los hámsteres inmunizados fueron desangrados a los 15 y 30 días después de la inmunización. Todos los sueros fueron aislados por centrifugación a 2500 rpm por 5 min. Para realizar el ensayo, las placas de fondo plano Nunc MaxiSorp de 96 pocillos se recubrieron con 100 μL de SARS-CoV-2 RBD y proteínas purificadas de la subunidad S1 (1 µg/mL) (GenScript, Piscataway, NJ, USA), disuelto en buffer carbonato-bicarbonato (pH 9.6) e incubado a 4°C durante la noche. Luego de cubrir las placas, se siguió el protocolo estándar de ELISA tal como se describió antes48.
Pruebas de neutralización utilizando un virus sustituto de SARS-CoV-2. Muestras de sueros fueron procesadas para evaluar títulos de nAbs contra el SARS-CoV-2. Todos los ensayos fueron realizados con sVNT, (GenScript, Piscataway, NJ, USA), siguiendo las instrucciones del fabricante. El punto de corte de positivo y negativo para la detección de nAbs de SARS-CoV-2 fueron interpretados como la tasa de inhibición, como: positivo, si ≥ 20% (anticuerpos neutralizantes detectados) y negativo, si < 20% (anticuerpos neutralizantes no detectables).
Prueba de neutralización por reducción de placa (PRNT). El SARS-CoV-2 (28549) fue aislado a partir de una muestra de hisopado nasofaríngeo colectado de un paciente con infección de SARS-CoV-2 confirmado en abril del 2020 en Lima, Perú. La identidad del virus fue confirmada mediante secuenciamiento de genoma completo. El aislamiento del virus se realizó utilizando células Vero 81 mantenidas en EMEM suplementado con SFB al 10%, 100 IU/ml de penicilina y 100 µg/ml de estreptomicina y cultivados a 37°C con una atmosfera humidificada de 5% de CO2.La muestra fue filtrada con una membrana con poros de 0.22 µm. Luego, 100 µL fueron usados para infectar las células Vero 81. Las células fueron observadas diariamente para detectar la presencia de efecto citopático y se colectó el virus para la confirmación. Los virus fueron propagados en cultivo de células Vero 81 para producción de stock viral y almacenadas a -80 °C y el título fue determinado por PFU.
Un pool de las muestras de suero de hámsteres colectadas en el día 30 después de la inmunización fueron inactivadas por calor a 56°C por 30 min. Luego, después de dos diluciones seriadas, las muestras de suero fueron mezcladas e incubadas con 40-50 PFUs de SARS-CoV-2 (28549) por 1 h a 37°C con 5% de CO2.Los sueros fueron mezclados con el SARS-CoV-2 (suero-virus), fueron añadidos sobre las células Vero-E6 (en una placa de 24 pocillos) e incubados a 37°C por 1 h. Luego de la adsorción, se removió las mezclas de suero-virus, y se añadió a las monocapas de células un medio de recubrimiento líquido (L-OM) que comprendía el 0.75% de carboximetilcelulosa (CMC) (Sigma-Aldrich) suplementado con 2 % de SFB y se incubó a 37°C/5 % de CO2 durante 5 días. Las placas fueron fijadas y coloreadas con formaldehido al 10 % y cristal violeta al 0.5 %59. Cada muestra se analizó por duplicado. Las placas fueron enumeradas para el cálculo de PRNT50, considerado como el método gold standard60.
Cuantificación de citoquinas por qPCR. Quince días después de la inmunización, se recolectaron bazos de hámsteres inmunizados con los diferentes virus recombinantes y fueron almacenados en el reactivo ARN-later a 4°C durante toda la noche y luego a -80°C. El ARN se extrajo con el Kit RNeasy Mini, se convirtió en ADNc con el Kit de síntesis de ADNc ProtoScript® II y se almacenó a -20°C hasta el análisis. El interferón-gamma (IFNγ), el factor de necrosis tumoral alfa (TNF-α) y la interleuquina-10 (IL-10), y el gen de referencia β-actina se evaluaron con pares de cebadores previamente reportados61,64. Se realizaron curvas estándar para todos los cebadores, obteniendo valores aceptables de eficiencia y R2 (datos no mostrados). Las mezclas fueron preparadas utilizando el Kit Luna® Universal qPCR Master Mix (New England Biolabs), según las instrucciones del fabricante. Brevemente, se usaron 5µl de la muestra (~2 ng/µL ADNc) con 2-3 repeticiones técnicas. Los experimentos cuantitativos de PCR en tiempo real (qPCR) se realizaron en el equipo Rotor-Gene Q (Qiagen, Hilden, Alemania) y se utilizó el método ΔΔCT65 para el análisis de datos.
Cuantificación de citoquinas por ELISA. Quince días post-inmunización, la sangre obtenida de los hámsteres inmunizados con: rLS1-HN-RBD, rLS1-S1-F, rLS1-HN-RBD/rLS1-S1-F y con fluido alantoideo (mock) fueron centrifugados a 1000 gravedades por 20 min a 4°C para obtener el suero, el cual fue diluido en alícuotas y almacenado a -80°C hasta su análisis. Para el ELISA cuantitativo varios kits precisos para la detección de citoquinas en hámsteres tales como, TNF-α, IFNγ, IL-2, IL-4, and IL-10 fueron adquiridos de MyBioSource, Inc., San Diego, CA. Las cuantificaciones de citoquinas fueron realizadas siguiendo las instrucciones del fabricante. Las placas fueron analizadas en el espectrofotómetro EON (Biotek, USA) a 450 nm. El nivel de citoquinas (pg/ml) detectado en el suero de los animales vacunados con rLS1-HN-RBD, rLS1-S1-F, y rLS1-HN-RBD/rLS1-S1-F fueron comparados con animales no vacunados (mock).
Eficacia de las vacunas contra el desafío de SARS-CoV-2. Cuarenta y ocho hámsteres Sirios Dorados, divididos en 4 grupos (n = 12): grupo 1 (rLS1-HN-RBD), grupo 2 (rLS1-S1-F), grupo 3 (rLS1-HN-RBD/rLS1-S1-F), y el grupo 4 control no vacunado, fueron desafiados por vía intranasal con 1×105 PFU de SARS-CoV-2/hámster (cepa Wuhan B.1.1, ID de acceso: GISAID EPI_ISL_1092347, gentilmente donado por el Instituto Nacional de Salud, Lima, Perú) en DMEM (volumen de 40µl) a los 45 días después de la primera inmunización. Cuatro animales de cada grupo fueron anestesiados y sacrificados con una sobredosis de 1 mL de una mezcla de ketamina (100 mg), xilazina (20 mg) y sulfato de atropina (1 mg) por inyección intramuscular a los 2, 5 y 10 dpc.
Las muestras de tejido pulmonar (pulmones derecho e izquierdo) se separaron en dos partes: (1) El lóbulo derecho fue utilizado para el examen patológico y (2) el pulmón izquierdo fue inmediatamente congelado a -80°C hasta su uso, este pulmón fue utilizado para virus infecciosos vivos por aislamiento viral. Todo el trabajo y manejo con SARS-CoV-2 fue realizado en un laboratorio BSL-3 siguiendo los lineamientos de bioseguridad del INS.
Análisis histopatológico. Los pulmones obtenidos de los hámsteres sacrificados en los días 2, 5, y 10 post-desafío con SARS-CoV-2 fueron fijados en formalina tamponada al 10% durante 48 h. Luego los tejidos fueron cortados y se colocaron en recipientes durante 24 h con formalina tamponada. Los recipientes con los órganos fueron procesados en una procesadora automática de tejidos (marca Microm) realizando los siguientes procesos: deshidratación, diafanización, enjuague e impregnación en un ciclo de 8 h. Los órganos embebidos en parafina fueron cortados a 5 micras de espesor (Microtome Leica RM2245), colocados en una solución de flotación en un baño de agua, luego se fijaron en una lámina portaobjetos, y se secaron en la estufa (a 37°C durante 1 a 2 h). La tinción fue realizada con Hematoxilina y Eosina (H&E). Finalmente, los portaobjetos fueron teñidos con H&E y analizados con una cámara AxioCam MRc5 y un microscopio AxioScope.A1 (Carl Zeiss, Alemania) con un aumento de 20 y 40x por un patólogo veterinario certificado.
Viabilidad viral: Cultivo y ensayo de inmunofluorescencia indirecta (IFI). Para determinar la viabilidad del virus, se trituraron y homogenizaron 60 muestras de tejido pulmonar de animales desafiados en DMEM al 5% p/v con antibiótico antimicótico al 1% y se centrifugaron a 10,000 rpm por 10 min a 4°C. El sobrenadante se filtró con una membrana de 0.22µm, luego se inocularon 100 µl en una monocapa confluente de células Vero 81 y se cultivaron a 37°C con una atmosfera humidificada al 5% de CO2. Los cultivos se observaron diariamente durante 10 días a través de un microscopio invertido. El IFI se realizó utilizando el anticuerpo policlonal contra SARS-CoV-2 de pacientes convalecientes de COVID-19 y un anti-IgG humano conjugado con HRP (Sigma).
Carga viral en pulmón. El aislamiento pulmonar del virus se confirmó mediante PCR de transcripción inversa cuantitativa (RT-qPCR), como se describió previamente66. Los niveles de carga viral de SARS-CoV-2 se cuantificaron utilizando el Kit Allplex n-CoV-2019 RT-PCR (Seegene®) para la detección de SARS-CoV-2. El sobrenadante o suspensión obtenida del homogeneizado pulmón de hámster se sometió a extracción y purificación del ARN y ADN total mediante el kit de perlas magnéticas Maxwell® RSC Viral Total Nucleic Acid Purification Kit y el Maxwell® 16 Instrument semi automated robot. El ARN purificado de las muestras se añadió a la mezcla de reacción de RT-PCR del Kit Allplex y analizado de acuerdo con las recomendaciones del Kit mediante el Kit de termociclador CFX96 (BIORAD®). El análisis de cuantificación de copias virales se realizó en función del valor Ct del gen N del virus SARS-CoV-2.
Movilidad animal. Para evaluar la movilidad de los hámsteres (en los grupos 1 al 4) post-desafío, la velocidad media, aceleración media, y el desplazamiento medio fueron calculados basado en videos con una cámara posicionada encima de los hámsteres. Los videos fueron analizados a los 2, 5, y 10 dpc.
Se debe notar que las condiciones de los registros de videos (distancia y enfoque) fueron mantenidos igual; por lo tanto, los pixeles siempre reflejaban la misma distancia. Dado que los hámsteres no necesariamente se mueven demasiado en los bordes de la caja, se estimó la velocidad, aceleración y desplazamiento medio basado en cualquier movimiento que se alejara de los bordes de la caja (Figura Suplementaria 4). Los movimientos a lo largo de los bordes de la caja fueron excluidos y registramos movimientos de un periodo de 2 a 3 min. Se registraron observaciones subsecuentes solo cuando los hámsteres estuvieron en el centro de la caja. El seguimiento fue llevado a cabo utilizando el Kernelized Correlation Filter (FCK)67. La implementación de este algoritmo de seguimiento fue desarrollada usando la librería OpenCV y el lenguaje Python. El resultado del seguimiento de los hámsteres fue un registro de las posiciones (X e Y) del hámster en la imagen. Finalmente, una vez que el seguimiento fue obtenido en los intervalos de interés, se calcularon la velocidad media, la aceleración media, y el desplazamiento medio para cada uno de los hámsteres.
Variación en el peso animal. El cambio del peso corporal fue medido a los 2, 5, y 10 dpc. Adicionalmente un grupo de animales (mock) no vacunados y no desafiados (n = 12) ubicados fuera del BSL-3 fueron evaluados. Estas medidas fueron usadas para calcular el porcentaje de la variación del peso corporal, comparado con el día 0 para cada animal.
Análisis estadístico. Para el análisis estadístico de la variación de peso en los grupos de hámsteres, se usó el análisis de varianza (ANOVA) de un factor en el paquete estadístico del software Stata v.16. Para la comparación de los tratamientos de la cuantificación de Citoquinas por qPCR y ELISA, se usó la prueba no paramétrica Mann–Whitney–Wilcoxon. Ambos se realizaron usando el software estadístico Stata v.16. Para evaluar la significancia estadística del cambio en el peso corporal en los grupos de hámsteres, y un ANOVA de un factor con múltiples comparaciones para todos los tratamientos involucrados se realizó en el software GraphPad Prism v.8.0.1. Para evaluar los cambios en la movilidad de los hámsteres durante el tiempo se usaron las pruebas estadísticas no paramétricas Mann–Whitney y Kruskal–Walls en el paquete SciPy v1.5.2. En todos los análisis, P < 0.05% fue considerado estadísticamente significativo. Para evaluar la reducción de placa (%) de neutralización a partir de los diferentes grupos de hámsteres, se usó ANOVA de dos factores y la prueba Tukey’s como prueba post hoc usando GraphPad Prism v.8.0.1.
- Gallagher, T. M. & Buchmeier, M. J. Coronavirus spike proteins in viral entry and pathogenesis. Virology 279, 371–374 (2001).
- Gorbalenya, A. E. et al. The species severe acute respiratory syndrome-related coronavirus: Classifying 2019-nCoV and naming it SARS-CoV-2. Nat. Microbiol. 5, 536–544 (2020).
- Walls, A. C. et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell 181, 281-292.e6 (2020).
- Xiaojie, S., Yu, L., Lei, Y., Guang, Y. & Min, Q. Neutralizing antibodies targeting SARSCoV-2 spike protein. Stem Cell Res. 50, 102125 (2021).
- Gavor, E., Choong, Y. K., Er, S. Y., Sivaraman, H. & Sivaraman, J. Structural basis of SARS- CoV-2 and SARS-CoV antibody inter- actions. Trends Immunol. 41, 1006–1022 (2020).
- Zhang, H. et al. Identification of an antigenic determinant on the S2 domain of the severe acute respiratory syndrome coronavirus spike glycoprotein capable of inducing neutralizing antibodies. J. Virol. 78, 6938–6945 (2004).
- Huang, J. et al. Priming with SARS CoV S DNA and boosting with SARS CoV S epitopes specific for CD4+ and CD8+ T cells promote cellular immune responses. Vaccine 25, 6981–6991 (2007).
- Yu, M. et al. Determination and application of immunodominant regions of SARS coronavirus spike and nucleocapsid proteins recognized by sera from different animal species. J. Immunol. Methods 331, 1–12 (2008).
- Du, L. et al. The spike protein of SARS-CoV—A target for vaccine and therapeutic development. Nat. Rev. Microbiol. 7, 226–236 (2009).
- Ma, C. et al. Searching for an ideal vaccine candidate among different MERS coronavirus receptor-binding fragments-the impor- tance of immunofocusing in subunit vaccine design. Vaccine 32, 6170–6176 (2014).
- Ahmed, S. F., Quadeer, A. A. & McKay, M. R. Preliminary identification of potential vaccine targets for the COVID-19 coronavirus (SARS-CoV-2) based on SARS-CoV immunological studies. Viruses 12, 254 (2020).
- Requena, D., Médico, A., Chacón, R. D., Ramírez, M. & Marín-Sánchez, O. Identification of novel candidate epitopes on SARS- CoV-2 proteins for South America: A review of HLA frequencies by country. Front. Immunol. 11, 1–16 (2020).
- Dai, L. & Gao, G. F. Viral targets for vaccines against COVID-19. Nat. Rev. Immunol. 21, 73–82 (2021).
- Guruprasad, L. Human SARS CoV-2 spike protein mutations. Proteins 89, 569–576 (2021).
- Zhang, L. et al. SARS-CoV-2 spike-protein D614G mutation increases virion spike density and infectivity. Nat. Commun. 11, 1–9 (2020).
- Greaney, A. J. et al. Complete mapping of mutations to the SARS-CoV-2 spike receptor binding domain that escape antibody recognition. Cell Host Microbe 29, 44-57.e9 (2021).
- 17. Van Egeren, D. et al. Risk of evolutionary escape from neutralizing antibodies targeting SARS- CoV-2 spike protein. PLoS ONE
16, e0250780 (2021).
- Ou, X. et al. Characterization of spike glycoprotein of SARS-CoV-2 on virus entry and its immune cross-reactivity with SARS-CoV.
Nat. Commun. 11, 1620 (2020).
- Garcia-Beltran, W. F. et al. Multiple SARS-CoV-2 variants escape neutralization by vaccine- induced humoral immunity. Cell 184, 2372-2383.e1-e9 (2021).
- Zhou, D. et al. Evidence of escape of SARS-CoV-2 variant B.1.351 from natural and vaccine-induced sera. Cell 184, 2348–2361 (2021).
- World Health Organization. Covid-19 Situation report. World Health Organ. 31, 61–66 (2020).
- Buschmann, M. D. et al. Nanomaterial delivery systems for mRNA vaccines. Vaccines 9, 1–30 (2021).
- Dong, Y. et al. A systematic review of SARS-CoV-2 vaccine candidates. Signal Transduct. Target. Ther. 5, 237 (2020).
- Amanat, F. & Krammer, F. SARS-CoV-2 Vaccines: Status report. Immunity 52, 583–589 (2020).
- Bello, M. B. et al. Exploring the prospects of engineered Newcastle disease virus in modern vaccinology. Viruses 12, 1–23 (2020).
- 26. Kim, S. H. & Samal, S. K. Newcastle disease virus as a vaccine vector for development of human and veterinary vaccines. Viruses
8, 183 (2016).
- Abozeid, H. H. et al. Development of a recombinant Newcastle disease virus-vectored vaccine for infectious bronchitis virus variant strains circulating in Egypt. Vet. Res. 50, 1–13 (2019).
- ICTV. International committee on taxonomy of viruses. https://talk.ictvonline.org/taxonomy/ (2019).
- Diel, D. G. et al. Complete genome and clinicopathological characterization of a virulent Newcastle disease virus isolate from South America. J. Clin. Microbiol. 50, 378–387 (2012).
- Miller, P. J. & Koch, G. Newcastle disease. In: Swayne, D.E. et al. Diseases of poultry 13, 89–107 (2013).
- Bukreyev, A. & Collins, P. L. Newcastle disease virus as a vaccine vector for humans. Curr Opin Mol Ther. 10, 46–55 (2008).
- Duan, Z., Xu, H., Ji, X. & Zhao, J. Recombinant Newcastle disease virus-vectored vaccines against human and animal infectious diseases. Future Microbiol. 10, 1307–1323 (2015).
- Shirvani, E., Paldurai, A., Manoharan, V. K., Varghese, B. P. & Samal, S. K. A recombinant Newcastle disease virus (NDV) express- ing S protein of infectious bronchitis virus (IBV) protects chickens against IBV and NDV. Sci. Rep. 8, 1–14 (2018).
- Saikia, D. P. et al. Recombinant Newcastle disease virus (NDV) expressing Sigma C protein of avian reovirus (ARV) protects against both ARV and NDV in chickens. Pathogens 8, 145 (2019).
- DiNapoli, J. M. et al. Newcastle disease virus, a host range-restricted virus, as a vaccine vector for intranasal immunization against emerging pathogens. Proc. Natl. Acad. Sci. U.S.A. 104, 9788–9793 (2007).
- Liu, R. Q. et al. Newcastle disease virus-based MERS-CoV candidate vaccine elicits high level and lasting neutralizing antibodies in Bactrian camels. J. Integr. Agric. 16, 2264–2273 (2017).
- Sun, W. et al. A Newcastle disease virus (NDV) expressing a membrane-anchored spike as a cost- effective inactivated SARS-CoV-2 vaccine. Vaccines 8, 1–14 (2020).
- Sun, W. et al. Newcastle disease virus (NDV) expressing the spike protein of SARS-CoV-2 as a live virus vaccine candidate. EBio‑ Medicine 62, 103132 (2020).
- Park, J.-G. et al. Immunogenicity and protective efficacy of an intranasal live-attenuated vaccine against SARS-CoV-2 in preclinical animal models. iScience 24, 102941 (2021).
- Rohaim, M. A. et al. A scalable topical vectored vaccine candidate against SARS-CoV-2. Vaccines 8(3), 472 (2020).
- Imai, M. et al. Syrian hamsters as a small animal model for SARS-CoV-2 infection and countermeasure development. Proc. Natl. Acad. Sci. USA 117, 16587–16595 (2020).
- Sia, S. F. et al. Pathogenesis and transmission of SARS-CoV-2 in golden hamsters. Nature 583, 834–838 (2020).
- Mebrahtu, K., Teshale, S., Esatu, W., Habte, T. & Gelaye, E. Evaluation of spray and oral delivery of Newcastle disease I2 vaccine in chicken reared by smallholder farmers in central Ethiopia. BMC Vet. Res. 14, 1–7 (2018).
- Zhao, W. et al. Newcastle disease virus (NDV) recombinants expressing infectious laryngotracheitis virus (ILTV) glycoproteins gB and gD protect chickens against ILTV and NDV challenges. J. Virol. 88, 8397–8406 (2014).
- Ma, J. et al. Newcastle disease virus-based H5 influenza vaccine protects chickens from lethal challenge with a highly pathogenic H5N2 avian influenza virus. npj Vaccines 2, 1–10 (2017).
- Lam, H. Y. et al. Corrigendum to ‘safety and clinical usage of Newcastle disease virus in cancer therapy’. Biomed Res. Int. 2017, 4529437 (2017).
- Bukreyev, A. et al. Recombinant Newcastle disease virus expressing a foreign viral antigen is attenuated and highly immunogenic in primates. J. Virol. 79, 13275–13284 (2005).
- Izquierdo-Lara, R., Chumbe, A., Calderón, K., Fernández-Díaz, M. & Vakharia, V. N. Genotype-matched Newcastle disease virus vaccine confers improved protection against genotype XII challenge: The importance of cytoplasmic tails in viral replication and vaccine design. PLoS ONE 14, 1–16 (2019).
- Nelson, C. B., Pomeroy, B. S., Schrall, K., Park, W. E. & Linderman, R. J. An outbreak of conjunctivitis due to Newcastle disease virus (NDV) occurring in poultry workers. Am. J. Public Health Nations. Health 42, 672–678 (1952).
- Dimitrov, K. M., Afonso, C. L., Yu, Q. & Miller, P. J. Newcastle disease vaccines—A solved problem or a continuous challenge?.
Vet. Microbiol. 206, 126–136 (2017).
- Cardenas-Garcia, S. et al. Molecular epidemiology of Newcastle disease in Mexico and the potential spillover of viruses from poultry into wild bird species. Appl. Environ. Microbiol. 79, 4985–4992 (2013).
- Cornax, I., Miller, P. J. & Afonso, C. L. Characterization of live LaSota vaccine strain induced protection in chickens upon early challenge with a virulent Newcastle disease virus of heterologous genotype. Avian Dis. 56, 464–470 (2012).
- Boumart, Z. et al. Thermal stability study of five Newcastle disease attenuated vaccine strains. Avian Dis. 60, 779–783 (2016).
- Torjesen, I. Covid-19 will become endemic but with decreased potency over time, scientists believe. BMJ 372, 494 (2021).
- Alexandra B Hogan et al. Report 33—Modelling the allocation and impact of a COVID-19 vaccine. Imp. Coll. London Rep. (2020).
- Chumbe, A., Izquierdo-Lara, R., Calderón, K., Fernández-Díaz, M. & Vakharia, V. N. Development of a novel Newcastle disease virus (NDV) neutralization test based on recombinant NDV expressing enhanced green fluorescent protein. Virol. J. 14, 1–11 (2017).
- Peeters, B. P. H., Gruijthuijsen, Y. K., De Leeuw, O. S. & Gielkens, A. L. J. Genome replication of Newcastle disease virus: Involve- ment of the rule-of-six. Arch. Virol. 145, 1829–1845 (2000).
- World Organization for Animal Health. OIE Manual of diagnostic tests and vaccines for terrestrial animals (mammals, birds and bees). OIE Manual of Diagnostic Test and Vaccines for Terrestrial Animals Seventh Edition, 2012 vol. 2 (2012).
- Mendoza, E. J., Manguiat, K., Wood, H. & Drebot, M. Two detailed plaque assay protocols for the quantification of infectious SARS-CoV-2. Curr. Protoc. Microbiol. 57, 1–15 (2020).
- Timiryasova, T. M. et al. Optimization and validation of a plaque reduction neutralization test for the detection of neutralizing antibodies to four serotypes of dengue virus used in support of dengue vaccine development. Am. J. Trop. Med. Hyg. 88, 962–970 (2013).
- Boudewijns, R. et al. STAT2 signaling restricts viral dissemination but drives severe pneumonia in SARS-CoV-2 infected hamsters.
Nat. Commun. 11, 1–10 (2020).
- Chan, J. F., Zhang, A. J., Yuan, S. & Kwok-Y, Y. Simulation of the clinical and pathological manifestations of Coronavirus Disease 2019 (COVID-19) in golden Syrian hamster model: Implications for disease pathogenesis and transmissibility. Clin Infect Dis. 2019, 1–50 (2019).
- Ribeiro-Romão, R. P., Saavedra, A. F., Da-Cruz, A. M., Pinto, E. F. & Moreira, O. C. Development of real-time PCR assays for evaluation of immune response and parasite load in golden hamster (Mesocricetus auratus) infected by Leishmania (Viannia) braziliensis. Parasit. Vectors 9, 1–12 (2016).
- Espitia, C. M. et al. Duplex real-time reverse transcriptase PCR to determine cytokine mRNA expression in a hamster model of New World cutaneous leishmaniasis. BMC Immunol. 11, 31 (2010).
- Livak, K. J. & Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method.
Methods 25, 402–408 (2001).
- Padilla-Rojas, C. et al. Near-complete genome sequence of a 2019 novel coronavirus (SARS-CoV-2) strain causing a COVID-19 case in Peru. Microbiol. Resour. Announc. 9, e00303-20 (2020).
- Henriques, J. F., Rui, C., Pedro, M. & Horge, B. High-speed tracking with kernelized correlation filters. IEEE Trans. Pattern Anal. Mach. Intell. 37, 583–596 (2015).
Temas relacionados:
Autores:











Mostrar más
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Usuarios destacados

Katherine Calderón Mayo  FARVET
FARVET

Investigadores del Laboratorio de Biotecnología Molecular y Genómica
Perú
Perú














