
Contenido patrocinado por:
FARVET
Patotipificación y caracterización filogénica de cepas del virus de la enfermedad de Newcastle en Perú
Publicado: 28 de marzo de 2017
Por: Ana Luz Chumbe Mendoza (Farvet SAC y UNMSM), Ray William Izquierdo Lara (Farvet SAC y UNMSM), Luis Tataje Lavanda (Farvet SAC), Gonzalez R.(UNMSM), Cribillero G (UNMSM), González A.E.(UNMSM), Manolo Fernandez Diaz (Farvet SAC), Eliana Icochea (Laboratory of Avian Pathology, Universidad Nacional Mayor de San Marcos UNMSM, School of Veterinary Medicine), Peru
Patotipificación y caracterización filogénica de cepas del virus de la enfermedad de newcastle en Perú: Definiendo dos nuevos subgenotipos dentro del genotipo XII
Resumen
Introducción:
Las infecciones de aves de corral con cepas virulentas de Paramixovirus aviar 1 (APMV-1), también conocido como virus de la enfermedad de Newcastle (NDV), causan la enfermedad de Newcastle (ND). Esta enfermedad altamente contagiosa afecta a aves de corral y muchas otras especies de aves alrededor del mundo. En países donde la enfermedad es prevalente, el monitoreo constante y la caracterización de aislados que causan brotes de la enfermedad son necesarios.
Las infecciones de aves de corral con cepas virulentas de Paramixovirus aviar 1 (APMV-1), también conocido como virus de la enfermedad de Newcastle (NDV), causan la enfermedad de Newcastle (ND). Esta enfermedad altamente contagiosa afecta a aves de corral y muchas otras especies de aves alrededor del mundo. En países donde la enfermedad es prevalente, el monitoreo constante y la caracterización de aislados que causan brotes de la enfermedad son necesarios.
Metodología:
En este estudio, reportamos los resultados de las pruebas de patogenicidad y los análisis filogenéticos basados en el gen de fusión (F) de siete cepas de NDV aisladas de varias regiones de Perú desde 2004 hasta 2015. Resultados: Seis de los virus exhibieron un índice de patogenicidad intracerebral (ICPI) entre 1.75 y 1.88, correspondiente al de un patotipo velogénico. El virus restante tuvo un ICPI de 0.00, correspondiente a un patotipo lentogénico. Estos resultados fueron consistentes con las secuencias de aminoácidos en el sitio de clivaje de la proteína F. Todos los aislados velogénicos presentaron la secuencia de aminoácidos polibásicos 112RRQKR↓F117 en el sitio de clivaje de la F. Los análisis filogenéticos de las secuencias completas del gen F revelaron que todos los aislados están clasificados en la clase II de los APMV-1. Las cepas velogénicas fueron clasificadas en el genotipo XII, mientras que el virus lentogénico pertenece al genotipo II y esta altamente relacionada a la cepa vacunal LaSota. Además, la topología del árbol, los valores de bootstrap y las distancias genéticas observadas dentro del genotipo XII resultaron en la identificación de los nuevos subgenotipos XIIa (en América del Sur) y XIIb (en China), y posiblemente dos clados dentro del genotipo XIIa. Todos los virus velogénicos aislados en Perú pertenecen al subgenotipo XIIa.
Conclusión:
En general, nuestros resultados confirman la presencia del genotipo XII en Perú y sugieren que es el genotipo prevalente actualmente circulando en nuestro país. La caracterización filogenética de estos aislados ayudará para caracterizar la evolución de NDV, y puede ayudar al desarrollo de vacunas específicas acorde a nuestras necesidades regionales.
En este estudio, reportamos los resultados de las pruebas de patogenicidad y los análisis filogenéticos basados en el gen de fusión (F) de siete cepas de NDV aisladas de varias regiones de Perú desde 2004 hasta 2015. Resultados: Seis de los virus exhibieron un índice de patogenicidad intracerebral (ICPI) entre 1.75 y 1.88, correspondiente al de un patotipo velogénico. El virus restante tuvo un ICPI de 0.00, correspondiente a un patotipo lentogénico. Estos resultados fueron consistentes con las secuencias de aminoácidos en el sitio de clivaje de la proteína F. Todos los aislados velogénicos presentaron la secuencia de aminoácidos polibásicos 112RRQKR↓F117 en el sitio de clivaje de la F. Los análisis filogenéticos de las secuencias completas del gen F revelaron que todos los aislados están clasificados en la clase II de los APMV-1. Las cepas velogénicas fueron clasificadas en el genotipo XII, mientras que el virus lentogénico pertenece al genotipo II y esta altamente relacionada a la cepa vacunal LaSota. Además, la topología del árbol, los valores de bootstrap y las distancias genéticas observadas dentro del genotipo XII resultaron en la identificación de los nuevos subgenotipos XIIa (en América del Sur) y XIIb (en China), y posiblemente dos clados dentro del genotipo XIIa. Todos los virus velogénicos aislados en Perú pertenecen al subgenotipo XIIa.
Conclusión:
En general, nuestros resultados confirman la presencia del genotipo XII en Perú y sugieren que es el genotipo prevalente actualmente circulando en nuestro país. La caracterización filogenética de estos aislados ayudará para caracterizar la evolución de NDV, y puede ayudar al desarrollo de vacunas específicas acorde a nuestras necesidades regionales.
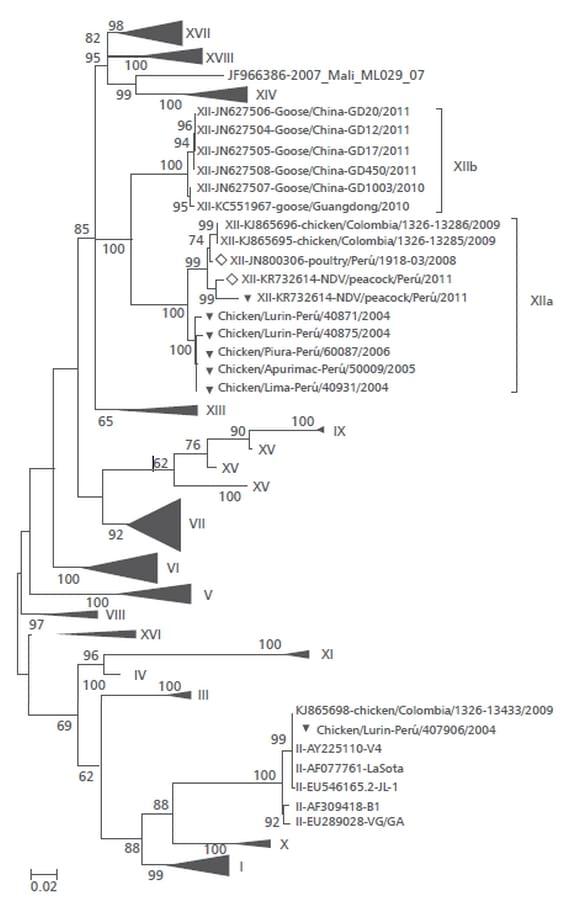
Figura 1. Análisis filogenético basado en el gen F de NDV. Se muestra en detalle las cepas pertenecientes a los genotipos XII y II. Las cepas descritas en este trabajo están denotadas como triángulos negros invertidos, mientras que el resto de cepas peruanas están denotadas con rombos blancos.
- El artículo original se encuentra en ingles, aquí
Temas relacionados:
Autores:
FARVET
FARVET
Universidad Nacional Mayor de San Marcos (Perú)
FARVET
Mostrar más
Referentes que Recomendaron :
Katherine Calderón MayoRecomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.





