INTRODUCCIÓN
El síndrome reproductivo y respiratorio porcino (PRRS) es una enfermedad de distribución mundial con efectos devastadores en la industria porcina. En Estado Unidos las pérdidas estimadas en la industria porcina debidas a esta enfermedad son alrededor de $560 millones de dólares al año (Neuman et al., 2005).
Esta enfermedad es causada por un virus RNA del género Arterivirus, de la familia Arteviridae. Debido a las diferencias en la secuencia de sus nucleótidos se han clasificado en dos genotipos, el tipo Europeo y el tipo Americano, compartiendo aproximadamente 63% de identidad a nivel genómico (Allende et al., 1999). Este virus tiene la capacidad de mutar rápidamente por lo que se pueden encontrar variantes del mismo en una sola granja e incluso en un mismo animal (Goldberg et al., 2003). Como consecuencia de estas mutaciones la respuesta inmune y la virulencia del virus de PRRS puede variar.
Diferentes estrategias de control han sido utilizadas contra esta enfermedad con el fin de estabilizar la respuesta inmune de los animales a través de la producción de anticuerpos y de reducir la presencia y circulación de variantes en las poblaciones animales. Existen pruebas diagnósticas para evaluar lo anterior, como la prueba de Ensayo de Inmunoabsorbancia Ligado a Enzimas (ELISA) utilizada para detectar la presencia de anticuerpos, así como para conocer la dinámica de circulación en la población animal, y por otro lado las técnicas moleculares que se utilizan para la detección del virus y variantes como es el caso de la prueba de RT-PCR en tiempo real, utilizando la técnica SYBR Green (Egli et al., 2001; Wasilk et al., 2004; Chung et al., 2005).
Entre las estrategias de control se mencionan, el uso de vacunas con virus vivos atenuados (MLV), el uso de autógenos (inóculos) con cepas obtenidas de la misma granja y la infección natural de los animales (Batista et al., 2004; Fano et al., 2004). La principal característica de las estrategias de control es la producción de las granjas de sus propios reemplazos. A pesar del uso, generalizado, de estas estrategias, en México y en Yucatán existe poca información sobre el éxito o fracaso de las mismas. Los objetivos del presente estudio fueron evaluar la respuesta humoral específica y la determinar la frecuencia de variantes del virus de PRRS en cerdos de la línea de producción, en tres granjas porcinas con diferentes esquemas de control.
MATERIAL Y MÉTODOS
Área de estudio
El estudio se realizó en tres granjas en el estado de Yucatán, México, localizado al sureste de México, entre las coordenadas 19º 30’ y 21º 35’ latitud norte y 87º 30’ y 90º 24’ longitud oeste del meridiano de Greenwich. El Estado limita al norte con el Golfo de México, al suroeste con el estado de Campeche y al sureste con el estado de Quintana Roo. El clima de la región es tropical subhúmedo (Aw0) con régimen de lluvias en verano (mayo a octubre), precipitación pluvial anual de 997 mm (rango de 700 a 2000 milímetros), temperatura media de 26.5º C (rango de 7 a 42º C), humedad relativa entre 61 y 87% y vientos predominantes de norte a sureste. Yucatán está libre del virus de la enfermedad de Aujeszky y Fiebre Porcina Clásica
Granjas de estudio
Las granjas de estudio fueron seleccionadas por conveniencia teniendo una población promedio de 2000 vientres cada una e instalaciones tecnificadas. La alimentación fue a base de alimento comercial y el destete se realizaba aproximadamente a los 21 días de edad; teniendo programas de limpieza y desinfección. El personal era exclusivo de cada área. Las tres granjas tenían programas de vacunación y medicación según las enfermedades presentes en cada granja. La reproducción de las piaras se realizaba mediante inseminación artificial y la tasa de reemplazo anual era de 40% con hembras provenientes del área de engorda (auto remplazo). Las tres granjas denominadas como A, B y C eran positivas al virus de PRRS y cada una utilizaba un método diferente de control contra el virus del PRRS.
En la granja A se utilizaba una vacuna comercial con virus vivo atenuado (Ingelvac ® PRRS MLV de Lab. Boehringer Ingelheim) con aplicación masiva en el hato reproductor cada 4 meses y a los cerdos a los 21 días de edad en la línea de producción (destete y engorda). Los reemplazos eran seleccionados a las 18 semanas de edad del área de engorda y eran llevados a un área de cuarentena donde recibían doble vacunación con el virus del PRRS a intervalos de 4 semanas y posteriormente eran integrados al hato reproductor cuando tenían una edad de 210 días y un promedio de peso de 140 kg.
En la granja B la estrategia se basó en el monitoreo serológico al ingreso y al final del periodo de aclimatación de los reemplazos (con un intervalo de 10 a 12 semanas). Los criterios de inclusión al hato reproductor dependieron de sí el animal era positivo cuando menos en alguno de los dos muestreos, asegurando así una previa exposición. El otro criterio era que hubiera 30% o más de animales seropositivos en la prueba de ELISA (kit CIVTEST SUIS PRRS A/S de laboratorios HIPRA). De igual forma se consideraba una edad de no menos de 210 días y un peso de 140 kg.
La granja C utilizaba inóculos de sueros positivos al virus de PRRS obtenidos de animales previamente clasificados como positivos, mediante la prueba de RT-PCR en tiempo real. Estos tenían un proceso de almacenamiento y crioconservación. Posteriormente, de acuerdo a la carga viral, se preparaban dosis individuales para ser inoculadas a cerdos de 70 días de edad en la línea de producción. Posteriormente, los remplazos permanecían en un área de cuarentena y eran introducidos al hato reproductor a los 210 días de edad con un peso de 140 Kg.
Diseño del estudio
Se realizó un estudio prospectivo longitudinal en 30 animales de cada granja. El tamaño de muestra fue obtenido considerando un nivel de prevalencia de variantes del virus del PRRS igual o mayor al 10% por granja y un nivel de confianza de 95% (Dean et al., 1995). Los animales fueron identificados individualmente en la oreja. Se tomaron muestras de sangre de los animales en los días 21, 49, 77, 105 y 160 de edad. Las muestras se obtuvieron por punción de la vena yugular. Las muestras fueron almacenadas en neveras de unicel con refrigerantes y enviadas al laboratorio para realizar la separación de suero y almacenadas en alícuotas.
Análisis de Laboratorio
Para detectar anticuerpos contra el virus de PRRS se utilizó el kit comercial (IDEXX HerdCheck PRRS, USA) que es una prueba de ensayo de inmunoabsorbancia ligado a enzimas (ELISA). Se realizó una dilución de 1:40 y posteriormente se siguió el protocolo recomendado por el fabricante. El punto de corte para determinar un suero positivo fue el coeficiente del valor de la absorbancia de la muestra sobre el valor de la absorbancia del suero control positivo (S/P) de modo que coeficientes S/P ≥0.40 fueron considerados positivos. La prueba tenía 97.4% de sensibilidad y 99.6% de especificidad. La prueba de ELISA se realizó en los laboratorios de Investigación Aplicada S.A. de C.V. en Tehuacán, Puebla.
La extracción de RNA se realizó a partir de 200µl de suero, utilizando el reactivo de extracción RNA WizTM de Ambion® RNA CompanyTM. El protocolo se realizó de acuerdo a las recomendaciones del fabricante (IDEXX HerdChek PRRS 2XR test kit Handbook. USA). La prueba de RT-PCR en tiempo real se realizó utilizando el kit comercial QuantiTect® SYBR® Green RT-PCR de QIAGEN®, siguiendo el protocolo descrito por el fabricante (QuantiTect® SYBR® Green RT-PCR Handbook, USA). Se formaron grupos de 5 muestras de suero cada uno. Se utilizaron iniciadores, diseñados a partir de la secuencia del marco de lectura abierta (ORF 7) correspondiente a la sepa Americana y Europea: sentido (forward) 5’-GGGGAATGGCCAGYCAGTCAA-3’ y antisentido (reverse) 5’- GCCAGRGGAAAATGKGGCTTCTC-3’. La mezcla de reacción fue constituida por iniciadores a una concentración final de 1µM, 10 µl de SYBR Green RT-PCR Master Mix, 0.2 μL de Quanti Tect RT mix y 8.8 µl de la muestra; obteniendo un volumen final de 20µl. La transcripción reversa se realizó a 50°C por 30 min, y posteriormente se realizó una preincubación a 95°C por 15 min. El protocolo de corrida constó de 40 ciclos de 95°C por 15 seg (desnaturalización), 60°C por 20 seg (alineación) y 72°C por 20 seg (extensión) colocando en este último paso los canales de lectura FAM/Sybr.
Posteriormente, con el fin de determinar las variantes del virus de PRRS a través del análisis de curvas de desnaturalización, los grupos de muestras que resultaron positivos a la corrida con ORF7, se analizaron nuevamente de manera individual por la prueba de RT-PCR en tiempo real utilizando como iniciadores la secuencia del ORF 5: sentido (forward) 5’- GTGGTGTATCGTGCCGTTCT-3’) y antisentido (reverse) 5’- GCCATTCAGCTCACATAGCG-3’. La mezcla de reacción fue constituida por los iniciadores a una concentración final de 0.5 µM, 10 µl de SYBR Green RT-PCR Master Mix, 0.2 μL de Quanti Tect RT mix y 8.8 µl de la muestra; obteniendo un volumen final de 20µl. La prueba transcriptasa reversa se realizó a 50°C por 30 min, posteriormente se realizó una preincubación a 95°C por 15 min. El protocolo de corrida constó de 40 ciclos de 95°C por 15 seg (desnaturalización), 50°C por 20 seg (alineación) y 72°C por 20 seg (extensión) colocando en este último paso los canales de lectura FAM/Sybr. El protocolo de curvas de desnaturalización consistió en rampas de 60°C a 95°C, incrementos de 0.5°C en cada paso, con intervalos de 5 seg.
En ambas amplificaciones se utilizó el SYBR Green RT-PCR Master Mix del kit comercial el cual contiene: HotStarTaq® DNA Polimerasa, QuantiTect SYBR Green RT-PCR Buffer, dNTP’s, SYBR Green I, ROX (colorante de referencia) y 5 µM de MgCl2. Se utilizó como control positivo la vacuna comercial con virus vivo modificado (Ingelvac ® PRRS MLV de Lab. Boehringer Ingelheim) y el inóculo de suero positivo al virus de PRRS. Para establecer la curva estándar de los controles positivos se realizaron 20 repeticiones.
Análisis de datos
Los datos fueron analizados utilizando estadística descriptiva y analítica. Los promedios de valores de S/P fueron comparados a partir de los 77 días de edad mediante análisis de varianza y pruebas de Tukey para comparaciones múltiples de medias (SPSS, 2006).
Las temperaturas de desnaturalización (Tm), fueron ordenadas y agrupadas considerando 2°C de diferencia entre variantes de acuerdo a los criterios descritos por Pham et al. (2005). Las curvas de desnaturalización de los grupos fueron comparadas a través de pruebas no paramétricas de Kolmogorov-Smirnov y Mann Whitney. La similitud entre las temperaturas de desnaturalización fue evaluada mediante análisis de similitudes (ANOSIM) de una vía usando como variable independiente los valores temperatura de las variantes (Clarke y Gorley, 2001). Estos análisis se basaron en matrices de similitud de Bray-Curtis construidas con las temperaturas de las variantes no transformadas. El valor de RANOSIM generado por ANOSIM es una medida relativa de las variantes definidas a priori que se encuentra entre -1 y 1. Un valor de cero indicando que no hay diferencias entre las variantes, mientras que un valor de uno indica que las variantes son diferentes (Clarke y Gorley, 2001; Clarke y Walmirck, 2001). La significancia estadística se probó con 10,000 permutaciones y se utilizó un escalonamiento multidimensional no métrico (nMDS) con la finalidad de observar gráficamente las diferencias entre variantes del virus detectadas por ANOSIM. Asimismo, se implementó de manera conjunta un análisis de clusters con la finalidad de apoyar los resultados observados en el nMDS. Todos los análisis previamente mencionados fueron realizados con el programa Primer 5 (Clarke y Gorley, 2001).
RESULTADOS
Ensayo de Inmunoabsorbancia ligado a enzimas (ELISA)
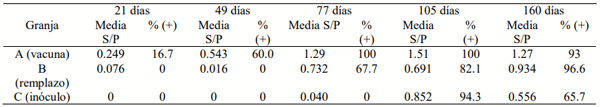
Los resultados de la prueba de ELISA mostraron que en la granjas A los mayores porcentaje de animales positivos y promedios de S/P se encontraron a los 77 y 105 días de edad; en la granja B a los 160 días, y en la granja C a los 105 (Tabla 1). Los promedios de S/P de los animales de la granja A fueron mayores que los obtenidos en las granjas B y C a 77, 105 y 160 días de edad (p< 0.05).
Tabla 1. Promedios de coeficientes S/P por edad y porcentaje de cerdos positivos a la prueba de ELISA.
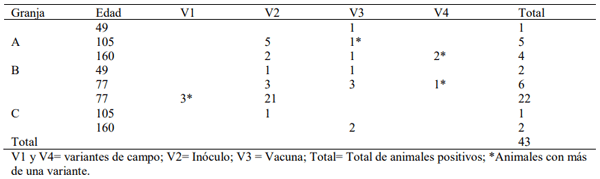
Tabla 2. Número y clasificación de animales positivos a la prueba de RT-PCR en tiempo real, por edad y por granja.
RT-PCR en tiempo real
Se obtuvieron 43 animales positivos de los 90, distribuidos en las tres granjas. La edad en la que se presentó el mayor número de animales positivos fue diferente en cada granja (Tabla 2).
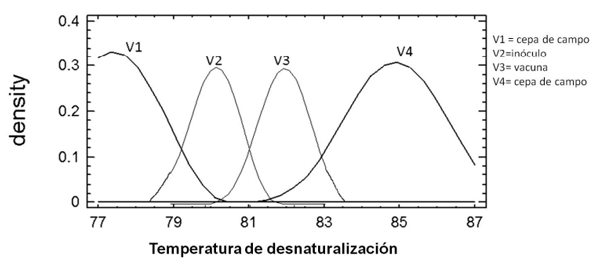
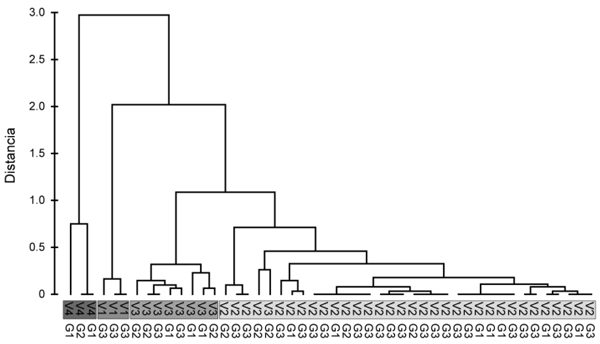
Según los rangos establecidos de las temperaturas de desnaturalización, se encontraron cuatro variantes del virus de PRRS (p< 0.05); una correspondiente al inóculo, otra a la vacuna y dos silvestres (Figuras 1, 2, 3). En las tres granjas se presentaron tres de las cuatro variantes del virus de PRRS; sin embargo, la frecuencia de presentación y el tipo de variante varió entre las granjas (Tabla 2). En las tres granjas, se observó la presencia de más de una variante de PRRS en un mismo animal.
Figura 1. Distribución de las curvas de desnaturalización correspondientes a cada variante encontrada.
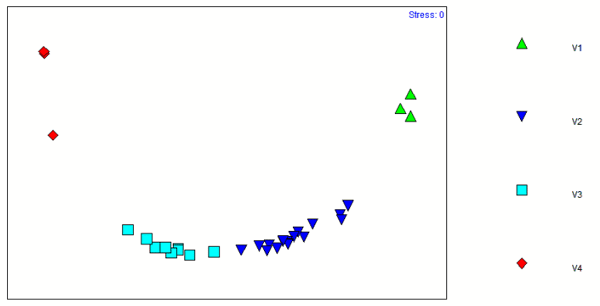
Figura 2.- Mapas de ordenamiento multidimensional de similitud (MDS)
Figura 3.- Dendograma de la clasificación de variantes según la temperatura de desnaturalización y granjas en las cuales se encontraron dichas variantes (G1= Granja A, G2=Granja B, G3= Granja C).
DISCUSIÓN
Los mayores porcentajes de animales positivos se observaron en los días 28, 56 y 35 después de la inmunización con el virus vacunal (Granja A), infección natural (Granja B) e inóculo respectivamente (Granja C), respectivamente. Estos resultados concuerdan con los de Zuckermann et al. (2007), quienes encontraron que los animales que recibieron una dosis de vacuna con virus vivo modificado (MLV), seroconvirtieron a los 28 días postvacunación y se mantuvieron seropositivos hasta el final del estudio (42 días), con valores de S/P entre 1.20 y 1.40. Similares resultados han sido reportados por otros autores (Diaz et al., 2006; Osorio et al., 1998; Cano et al., 2007).
El resultado en la granja C, concuerda con un estudio en el cual los animales inoculados con virus de campo, mostraron seroconversión a los 35 días post inoculación y se mantuvieron así hasta los 63 días post inoculación (Cano et al., 2007). Asimismo, Batista et al. (2004) encontraron que en animales inoculados con virus de campo, éstos fueron positivos a la prueba de ELISA a partir del día 14 postinoculación y se mantuvieron hasta el día 135 incrementando paulatinamente los títulos de anticuerpos. Sin embargo, en ese estudio, al realizar la prueba de seroneutralización se observó que sólo el 50% de los animales fueron positivos a los 14 días postinoculación, y hasta el día 50 postinoculación se encontró al 100% de los animales positivos. Estos resultados concuerdan con otros estudios en los que se han observado que los anticuerpos neutralizantes (AN) aparecen a partir de la tercera semana post-inoculación; sin embargo, existen estudios que muestran la presencia de AN en la segunda semana post-inoculación. Estas diferencias en la presencia de AN se deben a la variabilidad en la respuesta de los cerdos al virus del PRRS. En algunos cerdos no es posible detectar niveles significativos de AN durante todo el ensayo; sin embargo, los AN que se producen pueden permanecer durante periodos prolongados pero con títulos bajos (Loemba et al., 1996; Yoon et al., 1995; Mateu y Diaz, 2008).
Se ha descrito que la respuesta de las células T específicas contra el virus del PRRS también varía en cuanto a su aparición y esto puede deberse a la variedad de cepas existentes (Meier et al., 2003). Con respecto a lo anterior, en este estudio también se evaluó la frecuencia de variantes del virus del PRRS a través de las curvas de desnaturalización y la relación de éstas variantes con la presencia de anticuerpos. Las diferencias entre las temperaturas de desnaturalización para cada variante descrita coinciden con diferentes estudios de la genotipificación del virus del PRRS, encontrándose diferencias menores a los 10°C entre el genotipo Americano y el Europeo (Martínez et al., 2008;
Lurchachaiwong et al., 2008). García et al. (2007) plantea el uso de Sybr Green como una alternativa para el diagnóstico rápido del virus de PRRS, encontrando una temperatura de desnaturalización del producto amplificado de PCR similar al reportado por otros autores y el encontrado en el presente trabajo.
Con respecto a la relación entre los animales positivos a RT-PCR en tiempo real, el número de variantes y la presencia de anticuerpos; en la granja A, únicamente se encontró un cerdo positivo a los 49 días de vida (28 días post vacunación) y se obtuvo un resultado muy diferente en la prueba de ELISA como se mencionó anteriormente. Esto es similar a los reportados por Zuckermann et al. (2007) quienes no encontraron viremia de los 32 a los 42 días post vacunación en cerdos vacunados con virus vivos modificados. Así mismo, estos autores encontraron partículas virales en tejidos de los mismo cerdos vacunados, después del desafío con otra cepa viral. Sin embargo, la carga viral fue menor en ese grupo en comparación con el grupo de animales no vacunados. Otros autores (Cano et al., 2007; Nodelinjk et al., 2001) han demostrado que el uso de la vacuna MLV reduce significativamente las lesiones y signos clínicos, se reduce la proporción de cerdos infectados en forma persistente, y el tiempo de excreción viral, utilizando cepas homólogas del virus vacunal. Sin embargo, es claro que la vacuna no previene la reinfección con cepas homólogas, sólo disminuye los signos de la enfermedad. Con respecto a cepas heterólogas, se presenta el mismo escenario pero la protección es menor. En el presente estudio se observó una mayor cantidad de cerdos positivos al virus del PRRS por RT-PCR en tiempo real a los 105 y 160 días de edad, es decir a los 84 y 139 días post vacunación y al mismo tiempo la aparición de una nueva variante (Tm = 85.3 y 84.1) diferente a la vacunal y al inóculo. Esto explicaría los porcentajes elevados de cerdos positivos a la prueba de ELISA y los títulos elevados a estas edades quizás producidos por las nuevas variantes o bien por cierta protección heteróloga que pudiera producir la vacuna lo cual coincide con lo reportado por Cano et al. (2007).
En la granja B se encontraron animales positivos a los 49 días de vida, lo cual indica una infección natural; sin embargo, la presencia de anticuerpos se observó 56 días después. También se encontraron variaciones en las temperaturas de desnaturalización a partir de los 49 hasta los 77 días de edad, es decir que en esta granja existía una circulación temprana de diferentes variantes del PRRS y que no existe una fuerte respuesta inmunológica específica ya que el promedio de valores de S/P no rebasó el valor de 0.934 durante todo el estudio. Utilizando el gen ORF 5 se determinó, en este estudio, que existen variantes del virus en una sola granja durante una infección natural, llamadas cuasiespecies. Así mismo, los valores bajos de S/P en la granja B en comparación con las otras granjas podría explicarse a que el gen ORF 5 es el epítope encargado de la producción de anticuerpos neutralizantes y al existir variaciones en dicho gen la respuesta puede verse disminuida. Contrario a esto, Opriessnig et al. (2005) mencionan que la homología entre las secuencias del ORF 5 de diferentes aislados y la MLV no es un indicativo del grado de protección de la misma. Esto pudiera explicar lo que ocurre en los animales de la granja A.
En el caso de los animales inoculados con virus de campo (Granja C), se obtuvieron resultados positivos a RT-PCR en tiempo real a los 7 días post inoculación con 3 Tm diferentes, manteniendo menor cantidad de animales positivos hasta los 90 días post infección. Sin embargo, la respuesta inmunológica fue similar al de los animales vacunados, es decir, rápida y uniforme. A pesar de esto, los animales continuaron virémicos y con títulos bajos a los 160 días de vida, lo cual podría representar un riesgo ya que en las tres granjas los animales monitoreados eran reemplazos y podrían crearse sub-poblaciones que conlleven a la diseminación y mayor persistencia del virus. Los resultados en la granja C son similares a los encontrados en un estudio anterior en el que al inocular hembras de reemplazo negativas al virus del PRRS durante su crecimiento, se encontró que los anticuerpos declinaban a los 41 días post infección; a su vez esta práctica puede ser riesgosa ya que no se tiene un control sobre el contenido del inóculo ya que éste pudiera contener otros patógenos, cantidades variables del virus y variantes del mismo (Batista et al., 2002).
CONCLUSIONES
Las diferentes estrategias utilizadas en las granjas indujeron respuesta inmune en la mayoría de los animales. En la granja A se obtuvieron las medias más altas de S/P. Las estrategias de control no evitaron la presencia de variantes del virus del PRRS por lo que pudieran representar un riesgo para la población animal.