Resistencia Antimicrobiana en Producción Animal
Seguimiento de microorganismos resistentes a antibióticos de importancia crítica de alta prioridad en el sistema productivo avícola en el marco de “Una Salud”
Publicado: 31 de octubre de 2022
Por: Dr. Rafael Vignoli, Facultad de Medicina de la Universidad de la República (UdelaR). Uruguay
En el decimotercer programa general de trabajo de la OMS para llevarse a cabo entre el 2019 - 2023, la resistencia a antimicrobianos es considerada una de las grandes amenazas sanitarias de la humanidad (1), a la vez que se incluye el combate de la misma como una de las 5 plataformas a desarrollar durante el quinquenio(1). Para esto se enmarca el problema de la RAM en el contexto de “Una Salud” y se realizan estrategias en el marco de la tripartita OMS, OIE, FAO(1).
En este sentido el 75% del consumo de antibióticos a nivel mundial se destina a la producción de alimentos de origen animal, siendo la cadena avícola, la segunda en magnitud en relación al consumo de antibióticos por kilo de carne producida, bastante por debajo de la producción de cerdos y levemente superior a la producción de ganado bovino (2).
En trabajos previos en Uruguay, detectamos la presencia de resistencia a antibióticos críticos para la salud humana en aves reproductoras tanto adultas, como directamente en los fondos de caja de las aves de 1 día recién ingresadas al país desde Brasil (3, 4).
En este contexto, nos propusimos determinar hasta qué etapa de producción de los pollos parrilleros y las gallinas ponedoras, se detectaban los microorganismos o genes de resistencia encontrados en los pollitos de un día importados a nuestro país.
Se procesaron dos embarques de pollitos de un día, denominados lote 43 y lote 44.
Se realizaron muestreos de fondo de caja (al ingreso al país) y a las semanas 5, 18, 30 y 50 denominadas L43A-E y L44A-E respectivamente.
Las muestras fueron sembradas en caldo lactosado durante 24 horas y luego sembradas en placas de agar Mac Conkey lactosa suplementado con los siguientes antibióticos: ceftriaxona (1ug/ml), ciprofloxacina (0,25 ug/ml) y colistina (3ug/ml).
Las colonias seleccionadas de las placas crecidas en colistina fueron repicadas y se le realizó concentración inhibitoria mínima con inóculo ajustado a 1/10 de una dilución de Mac Farland 0.5 por método de dilución en agar de acuerdo a lo establecido por CLSI 2021. Solo aquellas que mostraban crecimiento en placas de Mueller Hinton conteniendo 1 ug/ml o mayores, fueron consideradas como resistente a colistina para su posterior estudio.
En este sentido el 75% del consumo de antibióticos a nivel mundial se destina a la producción de alimentos de origen animal, siendo la cadena avícola, la segunda en magnitud en relación al consumo de antibióticos por kilo de carne producida, bastante por debajo de la producción de cerdos y levemente superior a la producción de ganado bovino (2).
En trabajos previos en Uruguay, detectamos la presencia de resistencia a antibióticos críticos para la salud humana en aves reproductoras tanto adultas, como directamente en los fondos de caja de las aves de 1 día recién ingresadas al país desde Brasil (3, 4).
En este contexto, nos propusimos determinar hasta qué etapa de producción de los pollos parrilleros y las gallinas ponedoras, se detectaban los microorganismos o genes de resistencia encontrados en los pollitos de un día importados a nuestro país.
Se procesaron dos embarques de pollitos de un día, denominados lote 43 y lote 44.
Se realizaron muestreos de fondo de caja (al ingreso al país) y a las semanas 5, 18, 30 y 50 denominadas L43A-E y L44A-E respectivamente.
Las muestras fueron sembradas en caldo lactosado durante 24 horas y luego sembradas en placas de agar Mac Conkey lactosa suplementado con los siguientes antibióticos: ceftriaxona (1ug/ml), ciprofloxacina (0,25 ug/ml) y colistina (3ug/ml).
Las colonias seleccionadas de las placas crecidas en colistina fueron repicadas y se le realizó concentración inhibitoria mínima con inóculo ajustado a 1/10 de una dilución de Mac Farland 0.5 por método de dilución en agar de acuerdo a lo establecido por CLSI 2021. Solo aquellas que mostraban crecimiento en placas de Mueller Hinton conteniendo 1 ug/ml o mayores, fueron consideradas como resistente a colistina para su posterior estudio.
Resultados disgregados por lotes:
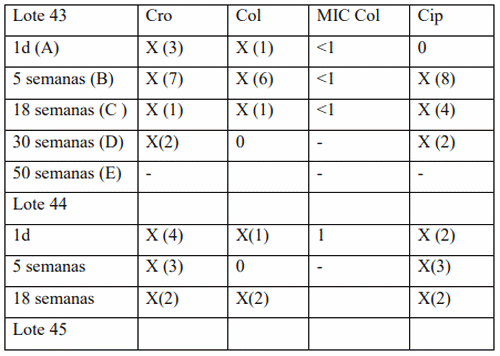
Tabla 1: Registro de crecimiento de microorganismos en placas con antibióticos. X: crecimiento; 0: sin crecimiento. Entre paréntesis se señala el número de colonias seleccionadas para estudiar de acuerdo a perfil de resistencia y crecimiento en placas. Para el caso de colistina, solo se consideran resistentes una vez que dan un valor de Concentración Inhibitoria Mínima mayor o igual a 1.
Se buscó la presencia de beta lactamasas de espectro extendido (BLEE) correspondientes a los grupos de CTX-M relacionados a CTX-M-1, CTX-M-2, CTX-M-8, CTX-M-9 y CTX-M-25.
Se determinó la búsqueda de genes transferibles de resistencia a quinolonas (qnrA, B, C, D, E, S y VC) y de resistencia transferible a colistina mediada por los genes mcr-1 a 9.
Se estudiaron 55 aislamientos de E. coli aislados de los diferentes muestreos procesados. A continuación se presentan los resultados de los 55 aislamientos estudiados.
De los 55, 31 procedía del lote 43 y 24 del lote 44.
Dentro del lote 43: se estudiaron 3 aislamientos al día de ingreso (L43A), 19 en la semana 5 (L43B), 4 en la semana 18 (L43C) y 3 en la semana 30 (L43D).
Por otro lado, en el Lote 44: se detectaron 11 al ingreso (L44A) 7 en la muestra a las 5 semanas (L44B) y 6 a las 30 semanas (L44C)
Resultados de las BLEE detectadas:
En los lotes 43 y 44 se detectaron 2 tipos de BLEE CTX-M-8 y CTX-M-55
En L43A se detectó un aislamiento productor de CTX-M-8, uno de CTX-M-55 y uno de ambas. Mientras que en L43B se estudiaron 5 aislamientos productores de CTX-M-8, 1 productor de CTX-M-8 y CTX-M-55 y uno productor de una BLEE de tipo CTX-M cuya variante alélica se está estudiando en este momento. Como ya se comentó en L43C no se detectaron aislamientos de E. coli productores de BLEE.
Mientras que en L43D se detectó un aislamiento productor de CTX-M-8
En L44A se detectaron 12 aislamientos portadores de BLEE, 9 productores de CTX-M-8 y 3 productores de CTX-M-8 y CTX-M-15. Por su parte en L44B se detectaron 6 aislamientos portadores de BLEE del tipo de CTX-M-15 y uno portando CTX-M-8 y CTX-M-15.
En la muestra L44C se detectaron 2 aislamientos portadores de CTX-M-55.
Resistencia a quinolonas:
En relación a la resistencia a quinolonas se estudiaron 33 aislamientos:
En el Lote 43 16 aislamientos: 10 en L43B, 4 en L43C y 2 en L43D.
En el lote 44 17: 6 en L44A, 9 en L44B y 2 en L44C
En relación a la resistencia transferible a quinolonas se detectaron:
En L43 13 asilamientos portador de algún gen:
7 qnrA, 4 qnrA y B, 1 qnrA y S y 1 qnrB
En L44, los 17 aislamientos resistentes a floroquinolonas presentaron algún gen de resistencia transferible:
13 qnrA, 3 qnrA y B y 1 qnrA, B y D
Teniendo en cuenta que los genes de resistencia en muchos aislamientos eran idénticos, se seleccionaron algunos aislamientos provenientes del mismo embarque para compararlos mediante electroforesis de campo pulsado. Todos los aislamientos fueron productores de CTX-M-55 y se incluyeron los aislamientos BB47 y RAM189-2 que habían sido aislados de reproductoras y pollitos de un día de trabajos anteriores.
En L43A se detectó un aislamiento productor de CTX-M-8, uno de CTX-M-55 y uno de ambas. Mientras que en L43B se estudiaron 5 aislamientos productores de CTX-M-8, 1 productor de CTX-M-8 y CTX-M-55 y uno productor de una BLEE de tipo CTX-M cuya variante alélica se está estudiando en este momento. Como ya se comentó en L43C no se detectaron aislamientos de E. coli productores de BLEE.
Mientras que en L43D se detectó un aislamiento productor de CTX-M-8
En L44A se detectaron 12 aislamientos portadores de BLEE, 9 productores de CTX-M-8 y 3 productores de CTX-M-8 y CTX-M-15. Por su parte en L44B se detectaron 6 aislamientos portadores de BLEE del tipo de CTX-M-15 y uno portando CTX-M-8 y CTX-M-15.
En la muestra L44C se detectaron 2 aislamientos portadores de CTX-M-55.
Resistencia a quinolonas:
En relación a la resistencia a quinolonas se estudiaron 33 aislamientos:
En el Lote 43 16 aislamientos: 10 en L43B, 4 en L43C y 2 en L43D.
En el lote 44 17: 6 en L44A, 9 en L44B y 2 en L44C
En relación a la resistencia transferible a quinolonas se detectaron:
En L43 13 asilamientos portador de algún gen:
7 qnrA, 4 qnrA y B, 1 qnrA y S y 1 qnrB
En L44, los 17 aislamientos resistentes a floroquinolonas presentaron algún gen de resistencia transferible:
13 qnrA, 3 qnrA y B y 1 qnrA, B y D
Teniendo en cuenta que los genes de resistencia en muchos aislamientos eran idénticos, se seleccionaron algunos aislamientos provenientes del mismo embarque para compararlos mediante electroforesis de campo pulsado. Todos los aislamientos fueron productores de CTX-M-55 y se incluyeron los aislamientos BB47 y RAM189-2 que habían sido aislados de reproductoras y pollitos de un día de trabajos anteriores.
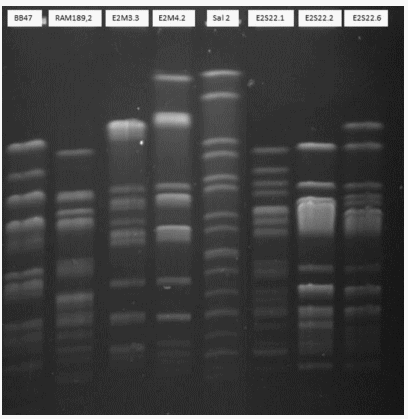
Figura 1: Electroforesis de cmpo pulsado de aislamientos de E. coli seleccionados provenientes de aves de 1 día y 5 semanas de vida.
Los primeros resultados demuestran que la presencia de microorganismos es policlonal, lo que sugiere una amplia diseminación horizontal de los plásmidos de resistencia.
Se seleccionaron 8 aislamientos de E. coli obtenidos de aves, 6 provenientes de los fondo de caja al momento del ingreso al país y 2 a las 5 semanas de ingresados, dos aislamientos causantes de infecciones del tracto urinario en humanos y un aislamiento obtenido de perro para estudiar mediante secuenciación.
Las principales características genómicas de los aislamientos se detallan en la tabla 2.
El análisis de las secuencias confirma que los aislamientos no se encontraban relacionados, como puede verse por la diversidad de secuenciotipos y serotipos.
De los 11 aislamientos estudiados, 7 presentaron la combinación de genes de resistencia CTXM-55 /fosA3 incluyendo 4 aislamientos de aves, los dos humanos y el procedente de perros.
Esta asociación de genes estaba codificada en todos los aislamientos en un plásmido del grupo de incompatibilidad IncFII_33 A-,B
Se seleccionaron 8 aislamientos de E. coli obtenidos de aves, 6 provenientes de los fondo de caja al momento del ingreso al país y 2 a las 5 semanas de ingresados, dos aislamientos causantes de infecciones del tracto urinario en humanos y un aislamiento obtenido de perro para estudiar mediante secuenciación.
Las principales características genómicas de los aislamientos se detallan en la tabla 2.
El análisis de las secuencias confirma que los aislamientos no se encontraban relacionados, como puede verse por la diversidad de secuenciotipos y serotipos.
De los 11 aislamientos estudiados, 7 presentaron la combinación de genes de resistencia CTXM-55 /fosA3 incluyendo 4 aislamientos de aves, los dos humanos y el procedente de perros.
Esta asociación de genes estaba codificada en todos los aislamientos en un plásmido del grupo de incompatibilidad IncFII_33 A-,B
Nuestro resultados sugieren que si bien existe una gran variabilidad de clones de E. coli (lo cual podría ser una consecuencia de la alta transferibilidad plasmídica horizontal), si se podrían estar movilizando los plásmidos entre las especies, de manera de representar un problema tanto de salud humana como animal.
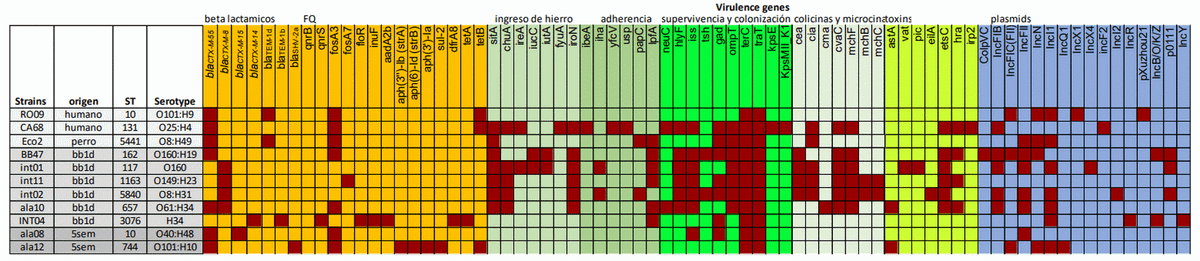
Tabla 2: Análisis genómico de 11 aislamientos de E. coli estudiados mediante secuenciación de genomas completos. Se muestran origen, secuenciotipo (ST), serotipo, genes de resistencia a antibióticos, genes de virulencia, grupos de incompatibilidad plasmídicos.
Referencias.
1. World Health Organization. 2019. Thirteenth General Programme of Work 2019–2023. .
2. Tiseo K, Huber L, Gilbert M, Robinson TP, van Boeckel TP. 2020. Global Trends in Antimicrobial Use in Food Animals from 2017 to 2030. Antibiotics 9:918.
3. Coppola N, Freire B, Umpiérrez A, Cordeiro NF, Ávila P, Trenchi G, Castro G, Casaux ML, Fraga M, Zunino P, Bado I, Vignoli R. 2020. Transferable Resistance to Highest Priority Critically Important Antibiotics for Human Health in Escherichia coli Strains Obtained From Livestock Feces in Uruguay. Front Vet Sci 7.
4. Coppola N, Cordeiro NF, Trenchi G, Esposito F, Fuga B, Fuentes-Castillo D, Lincopan N, Iriarte A, Bado I, Vignoli R. 2022. Imported One-Day-Old Chicks as Trojan Horses for MultidrugResistant Priority Pathogens Harboring mcr-9 , rmtG , and Extended-Spectrum β-Lactamase Genes. Appl Environ Microbiol 88.
1. World Health Organization. 2019. Thirteenth General Programme of Work 2019–2023. .
2. Tiseo K, Huber L, Gilbert M, Robinson TP, van Boeckel TP. 2020. Global Trends in Antimicrobial Use in Food Animals from 2017 to 2030. Antibiotics 9:918.
3. Coppola N, Freire B, Umpiérrez A, Cordeiro NF, Ávila P, Trenchi G, Castro G, Casaux ML, Fraga M, Zunino P, Bado I, Vignoli R. 2020. Transferable Resistance to Highest Priority Critically Important Antibiotics for Human Health in Escherichia coli Strains Obtained From Livestock Feces in Uruguay. Front Vet Sci 7.
4. Coppola N, Cordeiro NF, Trenchi G, Esposito F, Fuga B, Fuentes-Castillo D, Lincopan N, Iriarte A, Bado I, Vignoli R. 2022. Imported One-Day-Old Chicks as Trojan Horses for MultidrugResistant Priority Pathogens Harboring mcr-9 , rmtG , and Extended-Spectrum β-Lactamase Genes. Appl Environ Microbiol 88.
Trabajo de investigación financiado por el Programa de Investigación ALA y USPOULTRY (IPPE) . Ver más en ALA y USPOULTRY: Presentan investigaciones seleccionadas
Temas relacionados:
Autores:
Universidad de la República de Uruguay (UdelaR)
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.





.jpg&w=3840&q=75)