Estudio de asociaciones de genoma completo para producción de leche total en ganado holstein
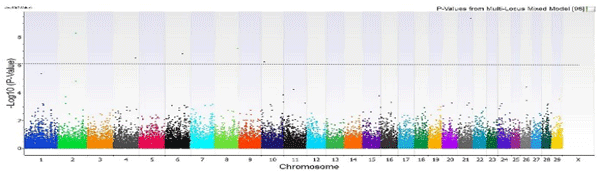
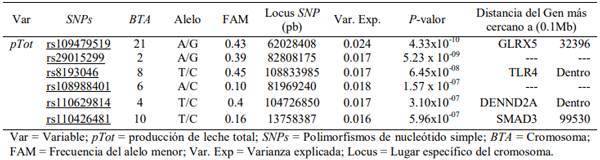
Avances en tecnologías moleculares para la selección animal han permitido la identificación de polimorfismos de nucleótido simple (SNPs), ubicados dentro o próximos a genes relacionados con caracteres de importancia fisiológica y económica. El objetivo fue realizar un análisis asociativo de genoma completo (GWAS) para identificar SNPs asociados con producción de leche total (pTot) en vacas Holstein. Se analizaron 430 lactancias con 53,443 registros diarios de leche (rLeche). Se utilizó el chip Illumina para el genotipado de 50,000 SNPs, con los cuales se realizó un análisis estadístico asociativo mediante un modelo lineal mixto multilocus. Se identificaron 6 SNPs, de los cuales dos de ellos están en una secuencia intrónica y dos en una posición intergénica cercana al gen (<0.1Mb). Se proponen los genes GLRX5, TLR4, DENND2A, SMAD3 como candidatos para pTot. El estudio de genoma completo permitió identificar SNPs y genes relacionados con la producción de leche total de vacas Holstein.
Baker, P. R., Friederich, M. W., Swanson, M. A., Shaikh, T., Bhattacharya, K., Scharer, G. H., Aicher, J., CreadonSwindell, G., Geiger, E., Maclean, K. N., Lee, W. T., Deshpande, C., Freckmann, M. L., Shih, L. Y., Wasserstein, M., Rasmussen, M. B., Lund, A. M., Procopis, P., Cameron, J. M., … Van Hove, J. L. K. (2014). Variant non ketotic hyperglycinemia is caused by mutations in LIAS, BOLA3 and the novel gene GLRX5. Brain, 137(2), 366–379. https://doi.org/10.1093/brain/awt328
Britt, J. H., Cushman, R. A., Dechow, C. D., Dobson, H., Humblot, P., Hutjens, M. F., Jones, G. A., Ruegg, P. S., Sheldon, I. M., & Stevenson, J. S. (2018). Invited review: Learning from the future—A vision for dairy farms and cows in 2067. Journal of Dairy Science, 101(5), 3722–3741. https://doi.org/10.3168/jds.2017-14025
Cai, H., Li, M., Jian, W., Song, C., Huang, Y., Lan, X., Lei, C., & Chen, H. (2020). A novel lncRNA BADLNCR1 inhibits bovine adipogenesis by repressing GLRX5 expression. Journal of Cellular and Molecular Medicine, 24(13), 7175–7186. https://doi.org/10.1111/jcmm.15181
Hernández-Cordero, A. I., Sánchez-Castro, M. A., Zamorano-Algandar, R., Luna-Nevárez, P., Rincón, G., Medrano, J. F., Speidel, S. E., Enns, R. M., & Thomas, M. G. (2017). Genotypes Within the Prolactin and Growth Hormone Insulin-Like Growth Factor-I Pathways Associated with Milk Production in Heat Stressed Holstein Cattle. Genetics and Molecular Research, 16(4), 1–10. https://doi.org/10.4238/gmr16039821
Leyva-Corona, J. C., Reyna-Granados, J. R., Zamorano-Algandar, R., Sanchez-Castro, M. A., Thomas, M. G., Enns, R. M., Speidel, S. E., Medrano, J. F., Rincon, G., & Luna-Nevarez, P. (2018). Polymorphisms within the prolactin and growth hormone/insulin-like growth factor-1 functional pathways associated with fertility traits in Holstein cows raised in a hot-humid climate. Tropical Animal Health and Production, 50(8), 1913–1920. https://doi.org/10.1007/s11250-018-1645-0
Luna-Nevarez, G., Kelly, A. C., Camacho, L. E., Limesand, S. W., Reyna-Granados, J. R., & Luna-Nevarez, P. (2020). Discovery and validation of candidate SNP markers associated to heat stress response in pregnant ewes managed inside a climate-controlled chamber. Tropical Animal Health and Production, 52(6), 3457–3466. https://doi.org/10.1007/s11250-020-02379-3
Marat, A. L., Dokainish, H., & McPherson, P. S. (2011). DENN domain proteins: Regulators of Rab GTPases. Journal of Biological Chemistry, 286(16), 13791–13800. https://doi.org/10.1074/jbc.R110.217067
Salcedo-Tacuma, D., Parales-Giron, J., Prom, C., Chirivi, M., Laguna, J., Lock, A. L., & Contreras, G. A. (2020). Transcriptomic profiling of adipose tissue inflammation, remodeling, and lipid metabolism in periparturient dairy cows (Bos taurus). BMC Genomics, 21(1), 1–13. https://doi.org/10.1186/s12864-020-07235-0
Wu, J., Li, L., Sun, Y., Huang, S., Tang, J., Yu, P., & Wang, G. (2015). Altered molecular expression of the TLR4/NF-κB signaling pathway in mammary tissue of Chinese Holstein cattle with mastitis. PLoS ONE, 10(2), 1–15. https://doi.org/10.1371/journal.pone.0118458
Zhang, L., Wu, Z. Q., Wang, Y. J., Wang, M., & Yang, W. C. (2020). MiR-143 regulates milk fat synthesis by targeting smad3 in bovine mammary epithelial cells. Animals, 10(9), 1–11. https://doi.org/10.3390/ani10091453





