INTRODUCCIÓN
En su mayoría las razas bovinas se encuentran genéticamente aisladas en virtud de la selección que ha realizado el hombre buscando rasgos de interés económico (Lourenco 2013). Los programas de mejoramiento genético en el país son pocos y por lo general se enfocan en características de crecimiento, como el peso a distintas edades y las ganancias de peso, ya que son fáciles de medir y presentan heredabilidades medias-altas (Jiménez et al. 2010). Sin embargo, características como la tolerancia o resistencia a enfermedades, a condiciones ambientales extremas, la buena calidad de los productos derivados o la mansedumbre no se han considerado en dichos programas. Dentro de estas características, la mansedumbre o temperamento del animal es fundamental en la regulación biológica y manifiesta del bienestar animal, lo que afecta directamente la productividad del animal (Mormède 2005). La disminución en la producción relacionada con el temperamento se ha estimado que ocasiona pérdidas anuales de 120 y de 31 millones de USD$ en Estados Unidos y México, respectivamente (Hall et al. 2011; Sutherland et al. 2012).
Se han estudiado variaciones del comportamiento y sus causas genéticas en ratones (Brodkin et al. 2002), perros (Topál et al. 2009), lobos (Hejjas et al. 2007), caballos (Momozawa et al. 2005), gallinas (Sugiyama et al. 2004), monos (Bailey et al. 2007) y bovinos (Laurenco 2013; Arredondo y Saavedra 2016); en estos últimos se ha demostrado que existen diferencias significativas en el temperamento de razas de fondo genético Bos taurus y Bos indicus y, por tanto, diferentes eficiencias reproductivas (Burdick et al. 2009) y productivas (Hiendleder et al. 2003).
Se han analizado diversos polimorfismos en genes que codifican enzimas biosintéticas, receptores o enzimas del metabolismo de los neurotransmisores de las rutas, tanto dopaminérgica, como serotoninérgica (Tochigi et al. 2006), que explican el temperamento del animal. Entre ellos, se destaca el gen que codifica para la dopamina β-hidroxilasa (DBH), enzima indispensable para la síntesis de la noradrenalina a partir de la hidroxilación de la dopamina en los gránulos cromafines (Lourenco 2013). La DBH también es un modulador fisiológico que actúa en el desarrollo fetal y en el sistema inmune mediado por células T. La noradrenalina es el mayor neurotrasmisor posganglionar del sistema nervioso simpático, liberándose desde la médula suprarrenal como respuesta a situaciones de estrés, fuga o defensa; su liberación masiva genera cambios en todo el organismo, como la liberación de glucosa, el mantenimiento de la homeotermia y el aumento de la respuesta cardiaca y respiratoria, control del comportamiento alimenticio y control de aspectos sensomotores (Romero et al. 2011). En humanos la DBH se ha asociado con diversos comportamientos como la respuesta a las drogas, el comportamiento materno, el comportamiento impulsivo, la motivación y el aprendizaje (Luksys et al. 2009).
El gen DBH en los bovinos se localiza en el cromosoma 11, entre los nucleótidos 104554824 y 104572619 (NM_180995.2) y está formado por 12 exones y 11 intrones; su producto es una proteína de 597 aminoácidos y 66 kda. En este gen se han reportado 62 polimorfimos, de los cuales ocho son no sinónimos (Lourenco-Jaramillo et al. 2012) y se localizan en los exones I, III y XII, formando parte del dominio funcional de la enzima y siendo el último exón el más polimórfico. El analisis de los polimorfismos genéticos existentes en el exón XII del gen DBH puede servir como estrategia para determinar si existen diferencias genéticas importantes que determinen el temperamento de las razas y su correlación con los rasgos productivos.
Por tanto, el objetivo de la presente investigación fue caracterizar los haplotipos genéticos del exón XII del gen DBH en las razas bovinas Blanco Orejinegro, Ganado de lidia y Brahman, mediante secuenciación directa bidireccional.
MATERIALES Y MÉTODOS
Animales y muestras
Se colectaron muestras de sangre mediante punción de la vena coccígea con el sistema Vacutainer® en tubos con anticoagulante (K2 EDTA 7,2 mg) en 52 animales de tres grupos raciales distribuidos así: 16 muestras de la raza criolla Colombiana Blanco Orejinegro (BON), 20 de la raza Brahman (BRA) y 16 de Ganado de lidia (GLI), provenientes de ganaderías ubicadas en La Virginia (Risaralda) y en Obando, La Victoria y El Cerrito (Valle del Cauca). Las muestras fueron transportadas a 4°C hasta el laboratorio y almacenadas a −20°C hasta su procesamiento.
Extracción del ADN
El ADN se extrajo mediante el método de precipitación salina; a 500 µl de sangre se añadieron 1 ml de solución de lisis I (0,32 M de sacarosa, 10 mM de Tris HCl pH 7,5, MgCl2 5 mM y 1% de Triton X100) y se mezclaron por inversión a temperatura ambiente durante 1 minuto. Se centrifugó a 14.000 rpm durante un 1 minuto y se descartó el sobrenadante; se realizaron sucesivos lavados con la solución de lisis I hasta obtener una pastilla limpia de glóbulos blancos. Se agregaron 400 µl de solución de lisis II (10 mM de Tris HCl pH 7,5, EDTA 10 mM pH 8,0, NaCl 50 mM y 0,2% de SDS) y 20 µl de proteinasa K (10 mg/µl) y se incubó a 56°C durante 1 hora. Se añadieron 400 µl de NaCl 5 M, se hizo vórtex durante 1 minuto y se centrifugó a 14.000 rpm durante 14 minutos. 350 µl del sobrenadante se mezclaron por inversión con 900 µl de etanol absoluto frío, se centrifugó a 14.000 rpm durante 12 minutos y se descartó el sobrenadante. Se agregó 1 ml de etanol al 70%, se mezcló por inversión durante 2 minutos y se centrifugó a 14.000 rpm durante 5 minutos. Se dejó secar el pellet de ADN durante 20 minutos a 37°C y luego se resuspendió en TE 1X (1 mM de Tris HCl pH 7,5, y 0,1 mM de EDTA pH 8,0) (Hernández et al. 2015).
La calidad y concentración del ADN se evaluó usando geles de agarosa al 0,8% en una cámara Sub-Cell® GT, (BIO-RAD, USA) teñidos con bromuro de etidio y visualizados con luz ultravioleta; la concentración se determinó mediante la comparación de las muestras con concentraciones conocidas del bacteriófago lambda.
Amplificación y secuenciación del gen DBH
Se amplificó el exón XII del gen DBH usando los cebadores: sentido 5` TTTGGTTTGGGAGAAGG 3´ y antisentido 5´ TGCACCCGTCACACTCAA 3´ (Lourenco-Jaramillo et al. 2012) a una concentración final de 0,2 µM, 20 ng de ADN, 2 mM MgCl2 , 25 µM dNTPs, 1X de tampón de PCR y 0,1 U Taq DNA polimerasa.
El perfil térmico de la PCR incluyó una desnaturalización inicial a 95°C por 5 min; 5 ciclos de 95°C por 45 segundos, 62°C por 45 segundos, disminución de 2°C cada ciclo, 72°C por 45 segundos; 30 ciclos de 95°C por 45 segundos, 55°C por 45 segundos, 72°C por 45 segundos para finalizar con una incubación final a 72°C por 10 minutos. Las amplificaciones fueron llevadas a cabo en un termociclador PTC-100® Teltier Thermal Cycler (BIO-RAD, USA).
Los amplificados se observaron en geles de agarosa al 1,2% teñidos con bromuro de etidio en una cámara Sub-Cell® GT, (BIO-RAD, USA) y visualizados con luz ultravioleta. El producto de la PCR fue un fragmento de aproximadamente 702 pb, y la concentración mínima de los amplificados fue 20 ng/µl. El secuenciamiento de las muestras se realizó en Macrogen Sequenciation Service, Maryland, USA.
Análisis estadístico
Las secuencias y sus electroferogramas fueron editados usando el programa Geneious 6.1 (Kearse et al. 2012) y alineadas usando el programa Mega 6 (Tamura et al. 2013), tomando como referencia la secuencia con el número de accesión NCBI NM_180995.2 perteneciente a Bos taurus.
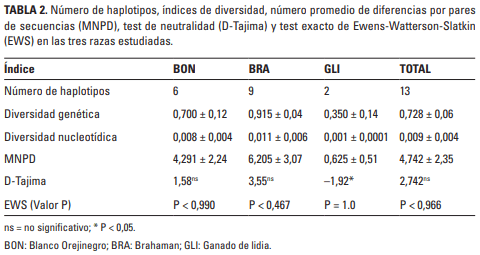
En cada raza calculó el número de haplotipos y sus frecuencias, la diversidad genética, la diversidad nucleotídica, el número promedio de diferencias por pares de secuencias (MNPD), se realizó el test de neutralidad de Tajima (D-Tajima), el test exacto de Ewens-Watterson-Slatkin (EWS) y se estimó el coeficiente de coancestría, mediante el programa Arlequin versión 3.5 (Excoffier y Lischer 2010). La relación entre los haplotipos se diagramó mediante una red con el programa Network 5.0 usando el algoritmo median-joining. Mediante el programa Mega 6 se determinó que el modelo evolutivo Tamura con 3 parámetros se ajustaba mejor a los datos; con este modelo evolutivo se estimaron las distancias utilizadas en los análisis de varianza molecular (Amova) con el fin de estimar la estructura y la diferenciación genética (FST) entre las poblaciones usando el programa Genalex versión 6.5 (Peakall y Smouse 2012).
RESULTADOS
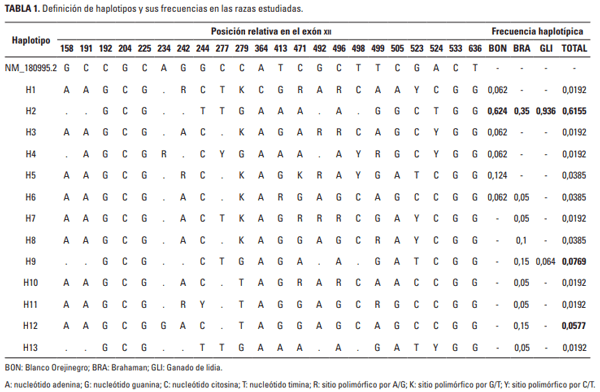
En la comparación con la secuencia consenso se identificaron 13 haplotipos diferentes en toda la población analizada (Tabla 1) definidos por 22 sitios variables de un amplificado con un tamaño de 702 pares de bases, con 10 transiciones y una transversión observada. Los haplotipos más frecuentes fueron el H2 (0,6155), el H9 (0,0769) y el H12 (0,0577), mientras los restantes 10 haplotipos tuvieron una frecuencia acumulada de 0,2499.
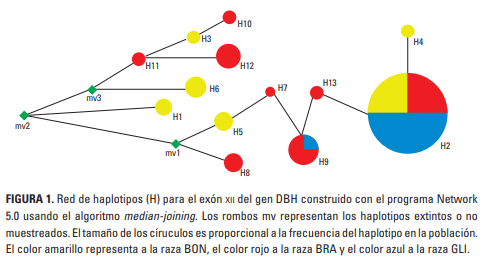
En la raza BON se encontraron seis haplotipos (H1 al H6), de los cuales el H2 fue el más frecuente (0,624), seguido por el H5 (0,124); los restantes cuatro haplotipos presentaron frecuencias de 0,062. En la raza BRA se encontraron nueve haplotipos (H2, H6 al H13) y los más frecuentes en esta raza fueron H2, H9 y H13 con frecuencias de 0,350 y 0,150 para los dos últimos, respectivamente. De otro lado, en el GLI solo se encontraron los haplotipos H2 y H9 con frecuencias de 0,936 y 0,064. En las razas BON y BRA se encontraron cuatro y seis haplotipos únicos y compartieron los haplotipos H2 y H6; la raza GLI no presentó haplotipos únicos y el H2 en esta raza se compartió con las razas BON y BRA, mientras que el H9 solo lo compartió con la raza BRA. El algoritmo median-joining determinó que el dorso de la red de haplotipos está conformada por los haplotipos H3, H10 y H11 (Figura 1) todos con frecuencia de 0,0192, y que es necesario la aparición de tres haplotipos extintos (mv) para explicar las relaciones observadas.
Otros parámetros de diversidad en el exón XII del gen DBH se presentan en la Tabla 2. La diversidad genética promedio fue de 0,728 ± 0,06, el mayor valor se encontró en la raza BRA seguido por BON y GLI. La diversidad nucleotídica total fue de 0,009 ± 0,004, por razas, el GLI presentó el menor valor y la BRA el mayor. El número promedio de diferencias por pares de secuencias presentó los valores más altos en las razas BRA y GLI, el valor MNPD en el BON fue de 4,291 ± 2,24 y en total fue de 4,742 ± 2,35. El test de neutralidad (D-Tajima) presentó valores positivos en las razas BON (1,58) y BRA (3,55) aunque no fueron significativos (P > 0,05), en GLI, este valor fue negativo y significativo (−1,92, P < 0,05). El valor de D-Tajima para toda la población no fue significativo. De otro lado el test de EWS no fue significativo en ninguna de las razas, ni para toda la población
El análisis de varianza molecular mostró mayor variación entre los individuos dentro de raza (76,07%) que entre razas para este gen (23,93%), sin embargo, el valor del FST fue de 0,239 (P < 0,001) (Tabla 3). Los valores de FST y los valores del coeficiente de coancestría por pares de razas se presentan en la Tabla 4. En ambos estadísticos los mayores valores se presentaron entre el par de razas GLI y BRA, seguido por GLI y BON y, el menor valor estuvo entre BON y BRA.
DISCUSIÓN
Existen pocos trabajos en bovinos dirigidos a describir los polimorfismos del gen DBH. En un estudio realizado en Colombia, Arredono y Saavedra (2016) reportan cuatro haplotipos en bovinos de lidia, ocho en el ganado Blanco Orejinegro y 13 en el ganado Brahaman. En México, Lourenco-Jaramillo et al. (2012) en las razas Charolais (n = 50), Brahman (n = 50), Holstein (n = 21) y de lidia (n = 19), reportan tres, siete, tres y un haplotipos, respectivamente. El presente trabajo, con un número menor de animales para BRA y GLI, reporta dos y un haplotipo más, respectivamente. De otro lado, Lourenco (2013) en 330 animales de ganaderías mexicanas, reporta un total de 21 haplotipos, 13 en Charolais y 16 en Brahman, con ocho haplotipos compartidos entre las dos razas. Las diferencias encontradas en el número de haplotipos entre los reportes citados y el presente trabajo se deben seguramente al tamaño de muestra utilizado.
De los siete haplotipos que LourencoJaramillo et al. (2012) reportan en Brahman, dos tienen una frecuencia acumulada del 87,5%. En un estudio posterior, de 16 haplotipos descritos en este mismo grupo racial, los dos más frecuentes suman 73,1% (Lourenco 2013). Por su parte, Arredono y Saavedra (2016), también en Brahman, hallaron tres haplotipos que suman el 50% de las frecuencias, mientras que los tres haplotipos más frecuentes en el presente trabajo para la raza BRA tienen una frecuencia acumulada del 65%. Lo anterior evidencia una mayor diversidad genética para este locus en la raza colombiana, lo cual se fundamenta en que fue el grupo racial, de los tres evaluados, con mayor valor de diversidad genética y nucleotídica.
La red de haplotipos revelada con el método de median-joining determinó que los haplotipos H3, H10 y H11 son los ancestrales (ver Figura 1): el primero de ellos solo se encontró en el BON y los últimos dos en el BRA; este hecho, junto con los haplotipos exclusivos de raza, las frecuencias haplotípicas, valores de diversidad genética y nucleotídica, permiten proponer un basamento genético para explicar las diferencias de temperamento entre las razas y justifican la realización de investigaciones encaminadas a determinar la asociación entre dichos polimorfismos y el temperamento de las razas con fondo genético B. taurus y B. indicus, y de esta manera ampliar los marcadores genéticos para esta característica (Mormède 2005; Core et al. 2009)
Con respecto a los índices de diversidad genética y nucleotídica, los mayores valores se presentaron en los ganados BRA y BON, y el menor en la raza GLI. Arredono y Saavedra (2016) reportan valores de DN similares en los tres grupos genéticos (0,012 en Brahaman, 0,008 en Blanco Orejinegro y 0,001 en el ganado de lidia). Las variaciones en estos parámetros pueden explicarse por varias razones; una de ellas es el origen y la formación de las razas. Para el caso del BON, que es una raza criolla colombiana, se dio a partir de los ganados traídos por los conquistadores españoles y es producto de aproximadamente 500 años selección natural en nuestro medio (Martínez 1992); por su parte, la raza BRA fue introducida al país en 1915 a través de ejemplares traídos desde Estados Unidos y en 1927 con animales importados de Brasil (Asocebú 2016); de otro lado, el GLI que fue introducido a Colombia en 1923 y son animales que provienen de las razas bravas criadas en España (Fedegan 2016).
Otra razón que puede explicar la baja diversidad en la raza GLI es la consanguinidad; Calero (2008) analizó la genealogía en dos ganaderías de lidia en Colombia y reporta que en dos de ellas hay valores de consanguinidad del 45,98% y 63,82%. Por su parte, Domínguez et al. (2010) reportan 43,2% de animales consanguíneos en cuatro ganaderías de lidia en México, mientras que, usando marcadores moleculares tipo microsatélites en 77 ganaderías de lidia mexicanas (n = 1.640 animales), se reporta un valor de endogamia de 47% (Cañón et al. 2007). Por su parte en la raza BON, Pedraza (2010) estimó un valor promedio de endogamia de 15% mediante 14 marcadores microsatélites y en la raza BRA, Álvarez (2008) usó 15 marcadores microsatélites y halló un valor promedio de endogamia del 22%. Por último, el número observado de transiciones nucleotídicas encontradas en BRA (10), BON (9) y GLI (5), junto con el alto valor de parecido entre los pares de secuencias (MNPD) en la última raza, explicarían la baja diversidad para el locus en estudio.
Los valores de diversidad genética y nucleotídica determinados en el gen DBH, pueden ser inferiores a aquellos medidos en regiones hipervariables del genoma, en las cuales haya mayor tasa de mutación (Sharma et al. 2015).
Con el fin de determinar la existencia de efectos de la selección neutral para el gen DBH en la población estudiada, se realizó el test de D-Tajima; éste mostró valores mayores que cero, aunque no significativos (P > 0,05) en las razas BON y BRA, indicando que en estas dos razas este locus ha evolucionado por equilibrio entre mutación y deriva genética, indicando selección neutral. Por otro lado, en la raza GLI el valor fue negativo y significativo (P < 0,05) lo que sugiere un barrido selectivo reciente después de un cuello de botella (Tajima 1989). De igual manera, el test de EWS, que permite evidenciar los efectos de la selección balanceadora, no fue significativo, lo que indica que en el locus en mención no han ocurrido eventos de selección positiva o negativa contra algún alelo (Hernández et al. 2015); ello sugiere que el temperamento o comportamiento de los animales ha sido tenido en cuenta en los planes de mejoramiento genético o de selección de reproductores de forma empírica y no con bases genéticas. Lo anterior lo corroboran Arredono y Saavedra (2016), quienes no encontraron asociaciones significativas entre entre SNPs y/o haplotipos en este gen con el temperamento de los animales evaluado como comportamiento en brete y velocidad de salida del brete.
El coeficiente de diferenciación genética (FST) para el locus y la población analizada fue más alto que el reportado por Arredono y Saavedra (2016), ya sea en la población total o al analizar por razas. Sin embargo, en ambos reportes el valor de FST indica una separación genética de las razas y un flujo genético nulo entre ellas (Wright 1950), valor que se sustenta en la gran variación encontrada entre razas con el análisis de varianza molecular (ver Tabla 3). Así mismo, no se encontraron similitudes genéticas entre las razas BON y GLI a pesar que comparten el mismo fondo genético (B. taurus). Sin embargo, los valores de coancestría, indican que la probabilidad de que dos alelos tomados al azar en dos individuos sean idénticos por descendencia es alta, sobre todo cuando se analiza la raza GLI, lo que puede ser interpretado como una huella de selección para el gen al igual que un aumento de la homocigosis en la población (Qanbari et al. 2010).
CONCLUSIONES
Se encontraron haplotipos compartidos entre las razas, así como haplotipos únicos en BON y BRA, con mayor diversidad en BRA, seguido por BON y GLI, lo que se explica por el origen y formación de las razas, así como posibles valores diferenciales de endogamia. Las secuencias analizadas sugieren que no se ha realizado selección a favor del temperamento en función del gen DBH, aunque las razas están bien diferenciadas.
AGRADECIMIENTOS
A Fernando Morales Vallecilla (Zootecnista, Especialista, candidato a Ph.D.) y a los productores Darío Sanint, Jaime Escobar y Juan Camilo Palomino. Al laboratorio de biología molecular de la Corporación Universitaria Santa Rosa de Cabal. A los estudiantes Yency Neira, Guillermo Castro, Nathaly Valencia, Marcela Santamaría y Claudia Restrepo.