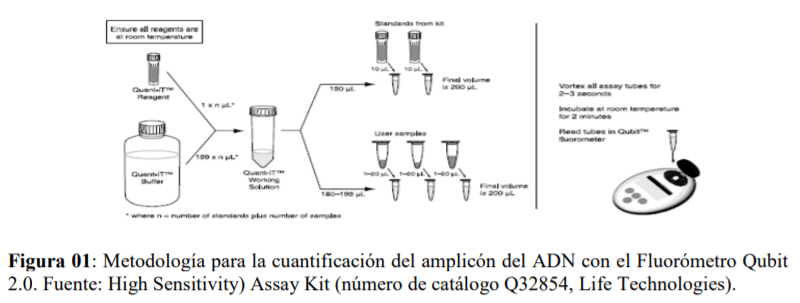
Estandarización de una PCR para detectar Fowl Adenovirus tipo 4 (FADV-4) en Farvet S.A.C.
Las enfermedades producidas por adenovirus aviar tipo 4 (FAdV-4) se trasmiten en forma vertical y horizontal causando grandes pérdidas económicas en el sector avícola, es por ello que existe la necesidad de llevar a cabo un proceso de estandarización de una reacción en cadena de la polimerasa (PCR) para detectar nuestro virus de interés. Para ello en la presente investigación se procedió a realizarlo en dos (02) fases: La primera consistió en la preparación del material de referencia interna en donde se extrajo el ADN, a partir de un cultivo células EB66, midiendo su cantidad del amplicón a través del Fluorómetro Qubit 2.0. La segunda fase fue estandarizar la PCR convencional, en esta fase se procedió a la selección de los cebadores (Primer´s) específicos, éstos fueron HHS*-2, HHS*-3 y HHS*-4; se realizó la prueba de su gradiente de temperatura basándose en el kit de PCR Q5® High-Fidelity 2X Master, seguido se realizó la prueba de concentración de cebadores, la prueba de sensibilidad siendo el 100% para los tres sets de cebadores y la prueba de especificidad obteniendo como resultado el 46.15%, 100% y 91.67% respectivamente.
Palabras claves: PCR, cebadores, estandarización, sensibilidad, especificidad.
i. Estandarizar la técnica de PCR para detectar Adenovirus aviar tipo 4 (FadV-4).
2.2. OBJETIVOS ESPECÍFICOS
i. Estandarizar la técnica de PCR convencional para detectar FAdV- 4
ii. Seleccionar los sets de cebadores específicos para detectar FAdV- 4 en PCR convencional
iii. Determinar el porcentaje de sensibilidad y especificidad del set de cebador seleccionado y su concentración de amplicón en la fase de estandarización de PCR convencional.
3. METODOLOGÍA
3.1. FASE I: PREPARACIÓN DEL MATERIAL DE REFERENCIA INTERNA (M.R.i.)
En los ambientes del Laboratorio de Cultivo Celular y Virología, se realizó el cultivo celular en células EB66 (células madre de embrión de pato) en suspensión, se utilizó el medio GRO I (marca SIGMA) por un periodo de 48 horas a 37°C con 5% de CO2. Una vez que el cultivo en el spinner alcanzó una cantidad de 6 x 106 células/ml, se procedió a infectarlo con Fowl adenovirus tipo 4 (FAdV-4) a una multiplicidad óptima de infección (M.O.I) de 0.02. Posteriormente, el cultivo infectado se incubó a 37°C con 5% de CO2 por un período de 6 días.
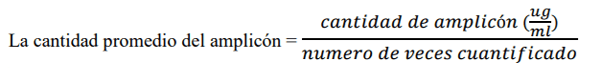
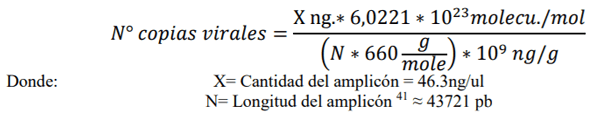
3.2. FASE II: ESTANDARIZACIÓN DE LA P.C.R CONVENCIONAL
En esta fase se realizaron las siguientes actividades:
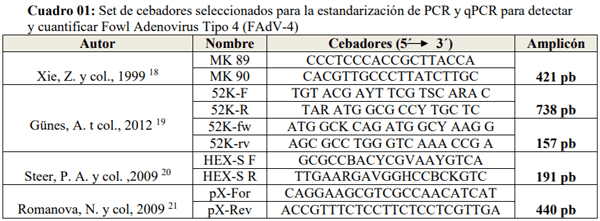
Para la elección de los cebadores, se realizó la búsqueda bibliográfica a partir de publicaciones en revistas científicas indexadas en la base de datos de National Center for Biotechnology Information (NCBI), las cuales fueron específicos para FAdV-4. La especificidad de dichos cebadores se evaluó con respecto a la secuencia genómica de FAdV-4 obtenidas de la base de datos del NCBI utilizando el programa bioinformático de libre acceso llamado SnapGene Viewer 4.0.3.
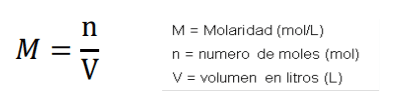
Se calculó la gradiente de temperatura de hibridación de los cebadores seleccionados, tomándose en cuenta su temperatura de fusión (Tm) se obtuvo en base a la siguiente fórmula Tm = 2(A+T) + 4(G+C).


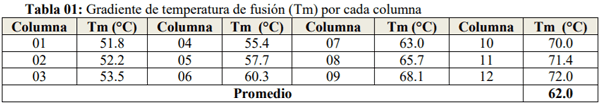
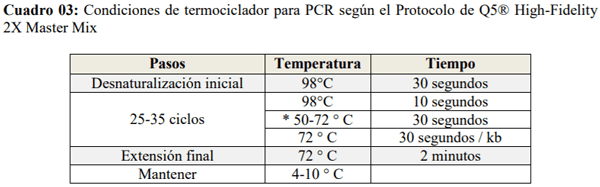
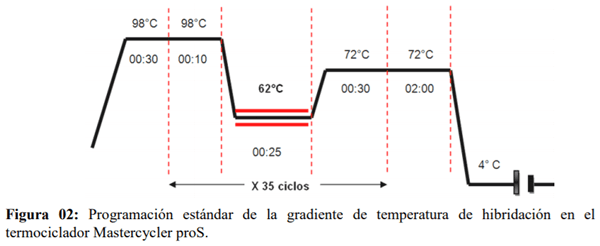
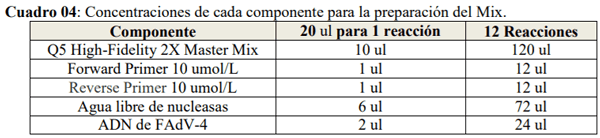
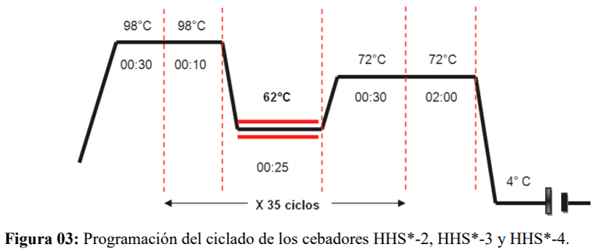
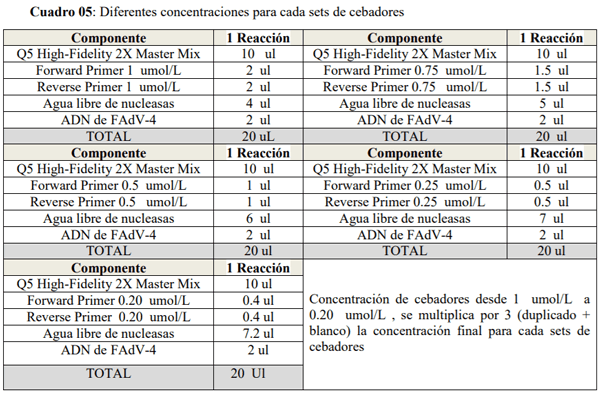
Una vez visualizado las bandas de los productos de PCR obtenidos (amplicones) a través de la electroforesis se eligieron los sets de cebadores que pasaron la prueba de gradiente de temperatura presentando bandas sin inespecificidades (sets de cebadores HHS*-2, HHS*-3 y HHS*-4) siendo la temperatura de hibridación seleccionada de 60°C. Luego se procedió a la preparación del Mix de reacción a diferentes concentraciones de cada sets de cebadores seleccionados partiendo desde 1 umol/L, 0.75 umol/L, 0.5 umol/L, 0.25 umol/L hasta 0.20 umol/L, por duplicado y un control negativo para cada mix (Cuadro 05). La programación estándar para los sets de cebadores seleccionados se basó en el Protocolo Q5® High-Fidelity 2X Master Mix, fue la siguiente: un paso de desnaturalización inicial una temperatura de 98°C por 30 segundos. Seguido por 30 ciclos de una etapa desnaturalización a 98°C por 10 segundos, una etapa de hibridación a una temperatura de 60°C por 30 segundos y una etapa de extensión fue de 72°C por 25 segundos. Posteriormente continuó un paso de extensión final de 72°C por 2 minutos, y culminó con un paso de enfriamiento a 4°C continuo una vez terminado el procedimiento.
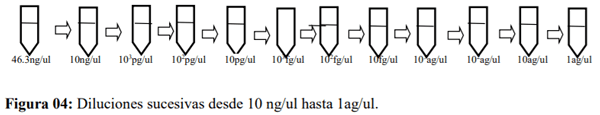
Una vez cuantificado el número de copias virales del M.R.i. se procedió a tomar un vial y se realizaron diluciones sucesivas, con el fin de saber hasta qué concentración llega la sensibilidad de cada sets de cebadores partiendo desde 10 ng/uL hasta 1ag/uL (Figura 04), este procedimiento se hizo en base a la fórmula de concentración – volumen ya mencionada:
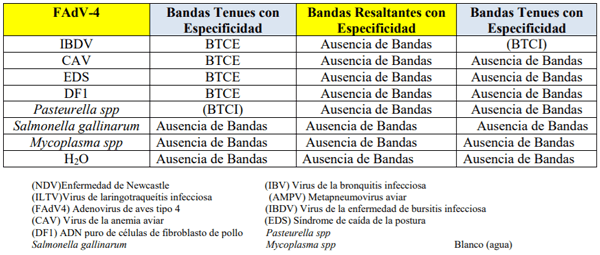
Esta prueba nos permitió saber si los sets de cebadores seleccionados son específicos únicamente para detectar FAdV-4, para ello se utilizaron varias muestras positivas para diferentes virus y bacterias aviares. Los patógenos utilizados fueron: el virus de la enfermedad de Newcastle (NDV), virus de la bronquitis infecciosa (IBV), virus de laringotraqueítis infecciosa (ILTV), metapneumovirus aviar (AMPV), Adenovirus de aves tipo 4 (FAdV4), virus de la enfermedad de bursitis infecciosa (IBDV), virus de la anemia aviar (CAV), síndrome de caída de la postura (EDS, por sus siglas en inglés), ADN puro de células de fibroblasto de pollo (DF1), Pasteurella spp, Salmonella gallinarum, Mycoplasma spp y como blanco (agua).

La cantidad promedio de amplicón del ADN extraído fue de 46.3 ng/ul, siendo éste el resultado del promedio entre la cantidad de amplicón en el ADN extraído y el número de veces (03) cuantificado. El ratio de la muestra de ADN fue de 1.91 teniendo un 60% de ácido Nucleico y 40% de proteínas. La cantidad del número de copias virales fue de 9.662* 107 copias virales.


ii. Para el sets de cebadores HHS* -3, su sensibilidad fue de 100% mientras que su especificidad fue de 100%, hasta una concentración del amplicón ≥1pg/ul.
iii. Para el sets de cebadores HHS* -4, su sensibilidad fue de 100% mientras que su especificidad fue de 91.67%, hasta una concentración del amplicón ≥1ng/ul.
ii. Los sets de cebadores que se seleccionaron para la detección de Adenovirus aviar tipo 4 fueron: 52K-F/52K-R (HHS* -2), 52K-fw/52K-rv (HHS* -3) y HEX-S F/HEX-S R (HHS* - 4),
iv. El sets de cebador que se seleccionó para la detección de Adenovirus aviar tipo 4 fue 52Kfw/52K-rv (HHS* -3), con una sensibilidad y especificidad de 100%, hasta una concentración del amplicón ≥1pg/ul.
1. King, A. M., Lefkowitz, E., Adams, M. J., & Carstens, E. B. (Eds.). Virus taxonomy: ninth report of the International Committee on Taxonomy of Viruses. Elsevier. 2011.
2. Wells, R. J., & Harrigan, K. A fatal adenovirus infection of broiler chickens: inclusion body hepatitis. Veterinary Record, 94(21). 1974;481-482.
3. Mazaheri, A., Prusas, C., Voss, M., & Hess, M. Some strains of serotype 4 fowl adenoviruses cause inclusion body hepatitis and hydropericardium syndrome in chickens. Avian Pathology.1998; 27(3), 269-276
4. Asthana, M .; Chandra, R .; Kumar, R. Síndrome de hidropericardio: estado actual y desarrollos futuros. Arco. Virol. 2013; 158 , 921–931.
5. McFerran JB, Adair BM. Infecciones por adenovirus del grupo I En: Saif YM, editor. Enfermedades de las aves de corral. Ames, Iowa: Iowa State Pr; 2003. pp. 214–227.
6. Zavala G. [homepaje on the Internet]. Epidemiología, Patogenia, Diagnóstico y Control de Adenovirus Aviares. AviNews.02 de Noviembre 2016. Disponible en: https://avicultura.info/epidemiologia-patogenia-diagnostico-control-adenovirusaviares/
7. Hess, M. (2000). Detection and differentiation of avian adenoviruses: a review. Avian Pathology. 2000; 29(3), 195-206.
8. Roldán Castañeda, O. J. Caracterización serológica de adenovirus tipo I y de la enfermedad de hepatitis con cuerpos de inclusión en los departamentos de Cundinamarca y Santander.2013
9. Instituto Nacional de Salud. Serie de Normas Técnicas N°38- Manual de procedimientos de electroforesis para proteínas y ADN. Lima, Perú:2003 El Ministerio de Salud. Recuperado el 11 de Diciembre de 2017, de http://www.ins.gob.pe/insvirtual/images/otrpubs/pdf/Manual%20Electroforesis%2038.pdf
10. Sharma, N., Verma, R., Kumawat, K. L., Basu, A., & Singh, S. K. miR-146a suppresses cellular immune response during Japanese encephalitis virus JaOArS982 strain infection in human microglial cells. Journal of neuroinflammation. 2015; 12(1), 30
11. Sah, S., Chen, L., Houghton, J., Kemppainen, J., Marko, A. C., Zeigler, R., & Latham, G. J. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome medicine. 2013; 5(8), 77.
12. Li, L., Luo, L., Luo, Q., Zhang, T., Zhao, K., Wang, H., & Zhang, W. Genome sequence of a fowl adenovirus serotype 4 strain lethal to chickens, isolated from China. Genome announcements. 2016; 4(2), e00140-16.
13. Xie, Z., Fadl, A. A., Girshick, T., & Khan, M. I. Detection of avian adenovirus by polymerase chain reaction. Avian diseases. 1999; 98-105
14. Günes, A., Marek, A., Grafl, B., Berger, E., & Hess, M. Real-time PCR assay for universal detection and quantitation of all five species of fowl adenoviruses (FAdV-A to FAdV-E). Journal of virological methods. 2012; 183(2), 147-153.
15. Steer, P. A., Kirkpatrick, N. C., O'Rourke, D., & Noormohammadi, A. H. Classification of fowl adenovirus serotypes by use of high-resolution melting-curve analysis of the hexon gene region. Journal of clinical microbiology. 2009; 47(2), 311-321
16. Romanova, N., Corredor, J. C., & Nagy, É. Detection and quantitation of fowl adenovirus genome by a real-time PCR assay. Journal of virological methods. 2009; 159(1), 58-63.
17. Snap Gene (Viewer 4.0.3) [software]. Obtenido de https://www.snapgene.com/
18. Xie, Z., Fadl, A. A., Girshick, T., & Khan, M. I. Detection of avian adenovirus by polymerase chain reaction. Avian diseases. 1999; 98-105
19. Günes, A., Marek, A., Grafl, B., Berger, E., & Hess, M. Real-time PCR assay for universal detection and quantitation of all five species of fowl adenoviruses (FAdV-A to FAdV-E). Journal of virological methods. 2012; 183(2), 147-153.
20. Steer, P. A., Kirkpatrick, N. C., O'Rourke, D., & Noormohammadi, A. H. Classification of fowl adenovirus serotypes by use of high-resolution melting-curve analysis of the hexon gene region. Journal of clinical microbiology. 2009; 47(2), 311-321
21. Romanova, N., Corredor, J. C., & Nagy, É.Detection and quantitation of fowl adenovirus genome by a real-time PCR assay. Journal of virological methods. 2009; 159(1), 58-63.
22. De Arruda Júnior, R. G. Temperatura de Melting: um estudo comparativo. Campo Grande. 2010
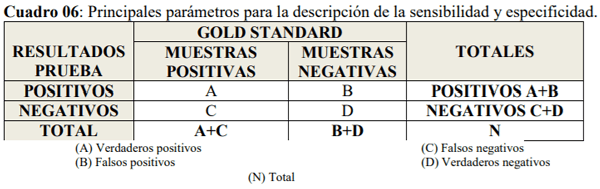
23. Altman DG, Bland JM. Diagnostic tests 1:Sensitivity and specificity. BMJ 1994; 308 (6943): 1552.