Los marcadores moleculares en la mejora genética
Publicado: 31 de mayo de 2023
Por: José Miguel Soriano y Rubén Rufo (programa de Cultivos Extensivos, IRTA) y Agata Gadaleta (Università degli Studi di Bari Aldo Moro, Italia).
Resumen
En este artículo dedicado a la mejora genética y a la biotecnología queremos hacer un repaso a los distintos tipos de marcadores que se utilizan en los programas de mejora genética, desde aquellos visuales y/o morfológicos usados en la mejora clásica, hasta los marcadores moleculares utilizados para realizar una selección precoz. También se trata su uso en la construcción de mapas genéticos y las distintas aproximaciones para el mapeo de caracteres de interés.
La mejora genética y sus inicios
La mejora genética comenzó hace más de 8000 años con el inicio de la agricultura sedentaria y la domesticación de los primeros cultivos, pero no fue hasta principios del siglo XX cuando se produjo su despegue con el redescubrimiento de los trabajos de Mendel de finales del siglo XIX que determinaron las leyes de la herencia. En la actualidad es una disciplina muy amplia gracias a los progresos de la biología y la genética molecular, habiéndose desarrollado nuevas técnicas basadas en la secuenciación del ADN.
La mejora genética vegetal se define como la elección hecha por el hombre de las mejores plantas escogidas dentro de una población en la que existe variabilidad. Por tanto, las premisas más importantes para el planteamiento de un programa de mejora son: la existencia de variabilidad, la capacidad de detectarla y de manipularla para producir una variedad más estable.
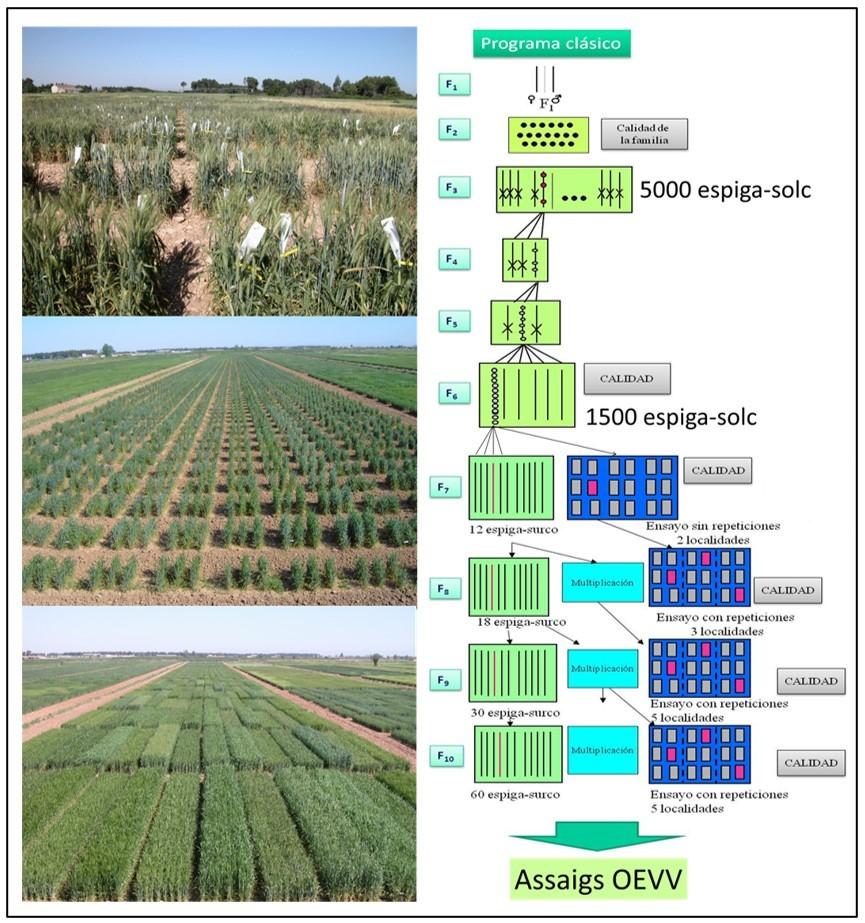
La mejora genética clásica se basa en el cruce de las mejores variedades y la selección de la descendencia mediante caracteres morfológicos o visuales tales como la resistencia a enfermedades y el rendimiento, los denominados marcadores morfológicos. Este proceso puede tardar entre diez y once años en dar resultados en el caso de cereales o hasta medio siglo en el caso de frutales. La figura 1 muestra de forma esquemática un programa de mejora de trigo, donde durante los primeros años la selección se realiza en espiga-surco. En estos ensayos se tienen en cuenta caracteres relacionados con la calidad, resistencia a enfermedades y componentes del rendimiento (número de espigas, número de granos por espiga, peso del grano). A partir del sexto año se empieza a realizar una selección en micro-parcelas para seleccionar las líneas más productivas.
La mejora genética basada en el ADN
El principal problema del uso de marcadores morfológicos es su alta influencia ambiental y su baja repetibilidad. Durante la década de los años 50 aparecieron los primeros marcadores bioquímicos, pero no fue hasta la década de los 90 cuando se dio un gran impulso a la mejora genética vegetal con la aparición de la biología molecular, en particular de la PCR (por las siglas en inglés de 'reacción en cadena de la polimerasa) y los marcadores basados en ADN (Figura 2). La ventaja de este tipo de marcadores es que pueden detectarse en cualquier estadio de desarrollo de la planta, no están influenciados por las condiciones ambientales y pueden encontrarse en un número prácticamente ilimitado. A partir de ellos, se han podido desarrollar mapas genéticos y localizar los genes responsables de algunos caracteres.
Los primeros tipos de marcadores de ADN que surgieron fueron los RFLP, basados en hibridación con sondas radiactivas que hacía muy laborioso su manejo. Para solucionar esta situación se desarrollaron marcadores basados en la PCR como RAPD, AFLP o SSR. Los dos primeros tipos se abandonaron progresivamente debido a su aleatoriedad y baja repetibilidad. Por el contrario, los SSR se han utilizado durante muchos años por su robustez, distribución a lo largo del genoma y su gran variabilidad. No ha sido hasta los últimos años cuando han empezado a sustituirse por los SNP. Estos marcadores se caracterizan por presentar polimorfismo en una sola base del ADN, lo que hace que estén muy representados en los genomas. El rápido avance en la tecnología de genotipado de alto rendimiento en el que se pueden obtener miles de marcadores ha rebajado mucho el precio para genotipar una variedad, haciendo que hoy los SNP sean los marcadores elegidos por la mayoría de grupos de investigación.

Figura 2. Amplificación mediante la PCR. A: aparato de PCR; B: cubeta de electroforesis en gel de agarosa; C: resultados de la PCR en gel de agarosa visualizado mediante rayos UV.
Esta nueva tecnología permite la construcción de mapas genéticos altamente saturados y, junto con la secuenciación completa del genoma, una identificación de genes candidatos muy rápida y económica.
Mapeo genético
Los marcadores moleculares se han usado ampliamente para la construcción de mapas genéticos. Éstos representan la posición de los marcadores moleculares a lo largo de los cromosomas y se basan en los eventos de recombinación entre los individuos de la población de mapeo (Figura 3). Las distintas aplicaciones de los mapas genéticos en la mejora vegetal pasan por la identificación de las asociaciones entre los marcadores moleculares y los caracteres de interés, y por el descubrimiento de los genes responsables de los caracteres. También se utilizan para la comparación de genomas entre diferentes especies y el mapeo físico para facilitar el ensamblaje de los genomas.
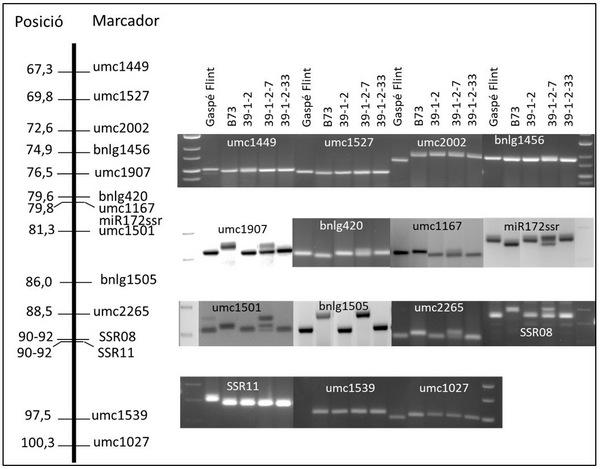
Figura 3. Representación de la parte central del cromosoma 3 del maíz y polimorfismo de los marcadores en la población resultante del cruce B73 x Gaspé Flint.
Los caracteres a mapear pueden dividirse en simples y complejos. Un carácter simple es aquel controlado por un gen, mientras que los caracteres complejos están controlados por dos o más genes, los denominados QTL, por sus siglas en inglés (quantitative trait loci). Los caracteres simples presentan una herencia mendeliana y son mapeados como un marcador más, haciendo muy fácil su localización cromosómica. Por el contrario, los caracteres complejos presentan distintos tipos de herencia o efectos (dominante, aditiva, epistasis, pleiotropía) y para su mapeo es necesario el uso de programas específicos.
Clásicamente, el mapeo de QTL se ha realizado en poblaciones derivadas de un cruce entre dos parentales que presentan diversas características fenotípicas, el denominado mapeo biparental o mapeo de ligamiento. El éxito del mapeo depende de la densidad de marcadores que existen en el mapa genético, el número de individuos de la población y la heredabilidad del carácter. Para solucionar las limitaciones de este mapeo biparental surgió otro tipo de mapeo, el llamado mapeo por asociación o GWAS (por sus siglas en inglés, genome wide association study). Éste es una aproximación complementaria que proporciona una cobertura de alelos más amplia y una mayor resolución del mapeo. Las diferencias entre ambas metodologías se encuentran en la Tabla 1. El GWAS se basa, al contrario que el mapeo bi-parental clásico, en el desequilibrio de ligamiento (LD), y se define como la asociación no hecha al azar de alelos de distintos genes. Para el análisis GWAS, en lugar de poblaciones derivadas de dos parentales, se utilizan colecciones de variedades con muchas y desconocidas relaciones entre ellas, por lo que es necesario realizar un análisis previo de su diversidad genética.
Tabla 1. Comparación del mapeo de QTL bi-parental y GWAS
Mapeo bi-parental | Mapeo de asociación |
Dos parentales conocidos | Muchos parentales desconocidos |
Pocas y conocidas recombinaciones entre alelos | Muchas y desconocidas recombinaciones entre alelos |
Estructura poblacional simple | Estructura poblacional compleja |
LD basado en la recombinación | LD basado en distintos sucesos: recombinación, mutación, deriva, selección. |
Es necesario desarrollar mapas genéticos | No requiere la construcción de mapas |
La selección asistida por marcadores
Los métodos de selección de los programas de mejora han evolucionado a lo largo del tiempo. La mejora clásica se basaba en la selección fenotípica, que implica la selección de las variedades con caracteres de interés usando características visuales o morfológicas como el rendimiento, fecha de floración, enfermedades, etc. El principal inconveniente de este tipo de selección es el tiempo invertido y el número de personas necesarias para realizarla.
Los marcadores moleculares son una alternativa para incrementar la eficacia de los programas de mejora mediante la selección asistida por marcadores o MAS (Figura 4). Ésta permite hacer una selección temprana de los individuos y, por tanto, reducir el tamaño de la población a evaluar simplificando el proceso de selección. La MAS depende del lincaje genético entre el carácter y el marcador molecular y, por tanto, es necesario conocer su posición en el mapa genético para escoger aquellos marcadores más cercanos al carácter. Esta selección se puede realizar únicamente con caracteres controlados por uno o muy pocos genes. La selección asistida se ha empleado principalmente en la mejora de la resistencia a enfermedades.
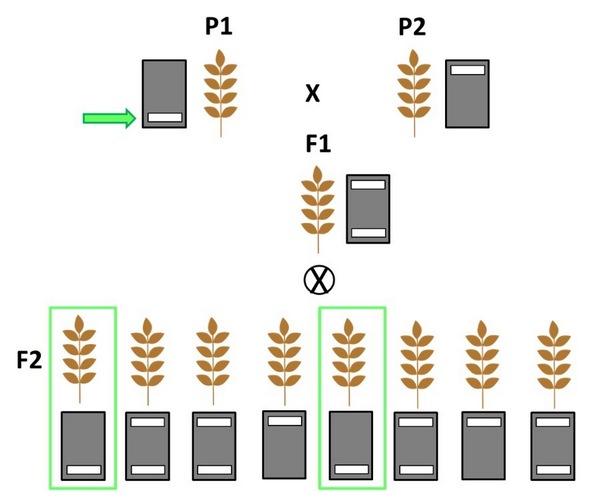
Figura 4. Esquema de selección asistida en un programa de mejora con líneas puras. El cruce entre dos líneas parentales con dotación alélica diferente origina una F1 con todos los individuos heterocigotos. La autopolinización de esta F1 genera una F2 con segregación. Solamente los individuos con la dotación alélica de interés en homocigosis son seleccionados para los últimos cruces, eliminando el 75% de la plantas.
Nuevas poblaciones de mapeo
Tal y como ya se ha comentado, las poblaciones biparentales tienen la limitación de poseer un bajo número de recombinaciones entre los dos parentales. Para solucionar esta situación se introdujo el mapeo de asociación como herramienta complementaria, que consiste en el uso de colecciones de germoplasma con individuos de distintos orígenes
En los últimos años, otros tipos de poblaciones de mapeo basadas en estas dos principales han ido desarrollándose para incrementar la significación estadística en la relación marcador-carácter. Dos ejemplos son las poblaciones MAGIC y NAM (Figura 1).
Las poblaciones MAGIC (Multiparent Advanced Generation InterCross) se generan por el cruce de varios parentales y la posterior autopolinización para generar líneas recombinantes. Comparado con las poblaciones biparentales, el uso de varios parentales en los cruces iniciales permite incrementar el número de recombinaciones y, por tanto, la resolución del mapeo y la diversidad alélica. En las poblaciones NAM (Nested Association Mapping) varios genotipos se cruzan con la misma línea de referencia para obtener familias segregantes “interconectadas”. La principal ventaja de estas poblaciones es la posibilidad de incorporar un alto número de alelos en un mismo fondo genético, permitiendo un mapeo más esmerado al combinar el mapeo biparental y el análisis de asociación.
Las variedades tradicionales presentan un gran interés para la generación de este tipo de poblaciones, sobre todo por su adaptación a diferentes ambientes, diversidad genética y resiliencia a estreses bióticos y abióticos.
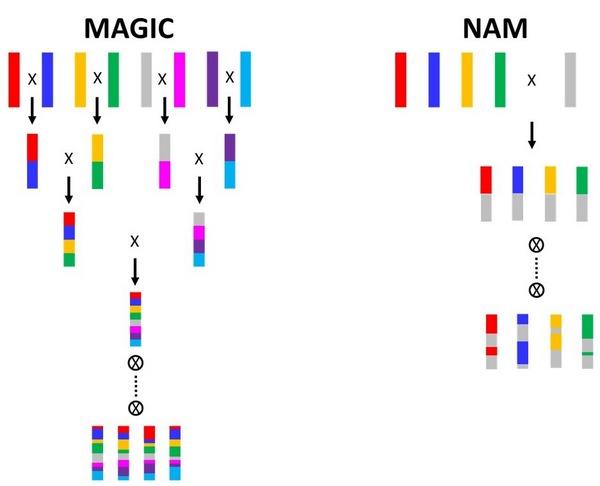
Figura 5. Esquema de la obtención de poblaciones MAGIC y NAM
Genes candidatos: una nueva estrategia para el estudio de caracteres complejos
Uno de los principales objetivos de la genética molecular es la identificación y aislamiento de los genes que controlan caracteres de interés. Existen tres estrategias para la clonación de estos genes: la clonación posicional (técnica para localizar la posición de un gen en un cromosoma), la mutagénesis insercional (introducción de secuencias de ADN extraño en un gen), y los genes candidatos (GC). En este apartado abordaremos esta última estrategia por ser la más sencilla y no requerir experimentos previos de mapeo o transformación genética.
Para la identificación de genes candidatos se parte de la secuencia de genes de función conocida que podría corresponder con el gen de interés. Los genes candidatos pueden ser genes estructurales o implicados en la regulación de vías metabólicas. La hipótesis es que una variación en estos genes puede explicar la variación fenotípica. La estrategia de los genes candidatos se basa en tres pasos:
1) Elección de los genes candidatos basada en estudios previos de la función de los genes o en su posición en mapas genéticos.
2) Estudio de variaciones en la secuencia de los genes candidatos para asociarla al carácter de interés en un conjunto de variedades.
3) Experimentos complementarios para confirmar la asociación real entre la variación en la secuencia y la expresión fenotípica.
2) Estudio de variaciones en la secuencia de los genes candidatos para asociarla al carácter de interés en un conjunto de variedades.
3) Experimentos complementarios para confirmar la asociación real entre la variación en la secuencia y la expresión fenotípica.
La estrategia de genes candidatos se adapta mejor al estudio de caracteres complejos que la clonación posicional y la mutagénesis insercional por el número de genes implicados en la expresión del carácter, su efecto parcial y por no ser necesario conocer su posición genética. La secuenciación de genomas y la creación de bases de datos de expresión génica han permitido grandes avances en la identificación de genes candidatos gracias a la aplicación de nuevos métodos de computación para priorizar la elección de estos genes.
La edición génica
La edición génica permite la manipulación específica de una secuencia diana para potenciar el rendimiento de un carácter concreto sustituyendo a los métodos de la mejora genética clásica. Al contrario que con el uso de plantas transgénicas, que no permiten conocer el lugar donde se integra el transgén, la edición génica modifica el gen in situ y, por tanto, no implica el uso de genes foráneos.
El sistema más conocido es CRISPR/Cas9, basado en un complejo sistema inmunitario que protege a las bacterias de infecciones víricas. CRISPR, del inglés 'clustered regularly interspaced short palindromic repeats' es una secuencia de ADN que contiene pequeños fragmentos de secuencia de virus que previamente han infectado la bacteria con el objetivo de reconocer el virus en una próxima infección. Las proteínas Cas son endonucleasas con la función de cortar el ADN en lugares concretos. Cuando se produce una nueva infección, las proteínas Cas reconocen estas secuencias y cortan el material genético del virus.
Esta propiedad de reconocer una determinada secuencia, cortarla y repararla siguiendo un patrón específico se ha adoptado para la edición génica (Figura 2). En primer lugar, se diseña una molécula de ARN (ARN guía) que es específica para una secuencia del ADN y que será insertada en una célula con la proteína Cas9. Una vez dentro de la célula, mediante el mecanismo de complementariedad del ADN, la molécula de ARN guía reconoce el lugar exacto del genoma donde Cas9 debe cortar. En una segunda etapa se activan los mecanismos de reparación del ADN cortado y se permite el reemplazamiento de una secuencia concreta que debe incorporarse a la célula.
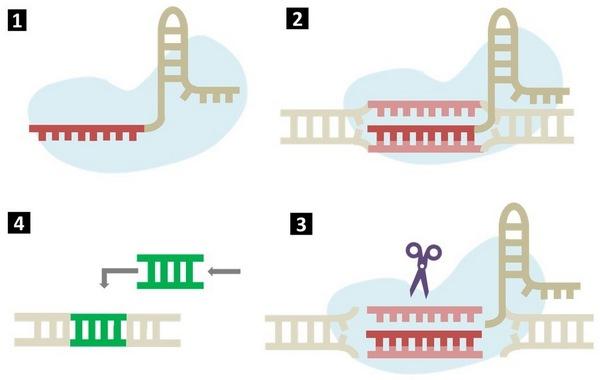
Figura 6. Técnica CRISPR/Cas9. 1) Se diseña una molécula de ARN guía con la secuencia del ADN que se desea modificar y se incorpora a la célula junto con la proteína Cas9. 2) El ARN guía se acopla a la secuencia diana. 3) Se produce el corte de la secuencia diana por parte de la proteína Cas9. 4) Se incorpora el fragmento de ADN a reemplazar y la maquinaria celular repara los cortes.
La selección genómica
Los métodos de selección usados en la mejora de cereales han evolucionado rápidamente a lo largo del tiempo. La mejora clásica basada en la selección fenotípica de los caracteres, principalmente morfológicos o visuales, fue la principal metodología utilizada por los mejoradores durante el siglo XX. El principal inconveniente de este proceso se presenta cuando los caracteres a evaluar dependen mucho de las condiciones ambientales. El desarrollo de la biología molecular permitió el uso de marcadores basados en la secuencia o polimorfismos del ADN, naciendo así la selección asistida por marcadores. El inconveniente de este método es que depende del ligamento genético del carácter con el marcador y sólo es válido para caracteres controlados por uno o muy pocos genes con gran efecto. Desgraciadamente, la mayoría de los caracteres agronómicos son cuantitativos y están controlados por muchos genes, por lo que son necesarias nuevas metodologías de selección. La disponibilidad de plataformas de genotipado de alta densidad ha revolucionado la mejora vegetal con nuevas herramientas para la predicción y selección genómica. La selección genómica se refiere a la selección de genotipos utilizando la información genómica a gran escala para estimar los efectos de todos los genes que controlan un carácter simultáneamente y predecir los valores en la descendencia (Figura 3). A diferencia de la selección asistida, la selección genómica lleva una importante base estadística y requiere cálculos más complejos.
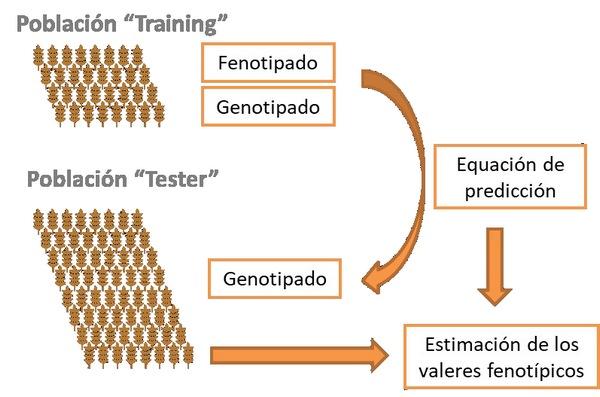
Figura 7. Selección genómica.
Para realizar un programa de selección genómica se parte de dos poblaciones relacionadas. La población “training” (TR) y la población “tester” (TS). La TR debe presentar por un lado variabilidad genética, pero por otro debe estar relacionada con la población en la que se aplicará la selección y presentar individuos de esa misma generación (si la selección se aplica en la generación F6, la TR deberá presentar individuos en F6). Esta población se fenotipa y genotipa masivamente para establecer una ecuación de predicción que asocie el carácter estudiado con los marcadores. Por el contrario, la TS sólo se genotipa de la misma forma que la TR y se aplica la ecuación establecida en la TR para estimar el valor del carácter en cada individuo de la población.
Speed Breeding
El término Speed Breeding se refiere al avance rápido de generaciones en condiciones controladas. Estos protocolos son considerados una buena aproximación para el desarrollo de nuevas poblaciones de mapeo y para avanzar las primeras generaciones de los programas de mejora, asistidas por marcadores moleculares, y así ahorrar tiempo a los mejoradores, reduciendo la longitud de los ciclos de mejora y seleccionando a los mejores individuos. Esta técnica se basa en la extensión del fotoperiodo usando potentes luces LED a una temperatura adecuada para el crecimiento. Algunas investigaciones en cereales han logrado llegar hasta 6 generaciones en un año.
Perspectivas futuras
Para llegar a cubrir las necesidades alimentarias del futuro, los productores tendrán que incrementar los rendimientos teniendo en cuenta el cambio climático. La información obtenida a partir de la secuenciación de genomas en cereales, junto a los estudios de mapeo de precisión y clonación de genes, permitirá la identificación de un gran número de marcadores moleculares y genes ligados a caracteres complejos que liderarán el aumento de rendimiento en los cultivos.
La reducción del coste de las nuevas tecnologías de secuenciación y la gran disponibilidad de datos de secuencias en bases de datos online permitirá la reducción del tiempo necesario para la identificación de estos genes candidatos. Por todo ello, proyectos que anteriormente requerían más de 10 años podrán lograrse en 1-2 años.
Temas relacionados:
Autores:
Referentes que Recomendaron :
Carlos C. De La Hoz C.Recomendar
Comentar
Compartir
Recomendar
Responder
Recomendar
Responder

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.