Introducción
El arroz (Oryza sativa L.) es uno de los cereales más importante cultivado en Venezuela. Entre los factores bióticos que afectan al cultivo del arroz se encuentra el añublo de la vaina o pudrición de la vaina (PDV), enfermedad causada por el hongo habitante del suelo Thanatephorus cucumeris (Frank) Donk (Anam: Rhizoctonia solani Kühn) (Ogoshi, 1987) e identificada por primera vez en Venezuela por Malaguti (1951). El agente causal del añublo de la vaina del arroz en Venezuela se ubica en el grupo de anastomosis AG-1 IA (Cedeño et al., 1996), caracterizado también usando microsatélites por González-Vera et al. (2010).
Las plantas de arroz manifiestan los síntomas de la enfermedad en la fase de máximo macollamiento, aproximadamente 50-60 días después de la germinación. Las infecciones comienzan al entrar en contacto los tallos con los esclerocios que sobrenadan en el agua de riego, estos germinan, desarrollan hifas que crecen en los tejidos de la planta y de estas hifas se desarrollan otras lobadas conocidas como cojín infeccioso de donde emergen las hifas de penetración (Rodríguez et al., 1999).
La mayoría de los síntomas en las plantas se observan en la base de los tallos a nivel del agua de riego, comenzando con una mancha acuosa, circular, oblonga o elipsoidal, de color verde oliva de aproximadamente 1 cm de longitud. Luego se desarrollan hasta alcanzar de 2-4 x 1-1,5 cm de longitud, presentándose un centro de color marrón claro o blanquecino, rodeada de un halo de color marrón o morado con un borde irregular (Correa-Victoria, 1993). En condiciones favorables, la enfermedad puede afectar las hojas superiores, produciendo síntomas similares a los que ocurren en las vainas y si estas condiciones persisten las lesiones pueden unirse y provocar el secamiento de las hojas y tallos, las perdidas en rendimiento pueden llegar de un 5% hasta un 50% (Nass et al., 1995).
Para el control de la enfermedad, se han usado prácticas culturales, entre ellas el uso de productos químicos (Nass et al., 1995), pero en los últimos años, por su impacto ambiental negativo, ha generado preocupación por el abuso del uso de agroquímicos en campos comerciales de arroz. Por ello, una alternativa duradera, económica y amigable con el ambiente es la obtención de cultivares de arroz resistentes a R. solani (Graterol et al., 1996; Delgado et al., 2004), siendo necesaria la identificación de genes que confieren resistencia a la enfermedad.
Aun cuando, R. solani afecta a un amplio rango de hospedantes, no se han identificado cultivares comerciales de arroz o especies silvestres relacionadas que presenten resistencia completa. Sin embargo, se han observado diferencias importantes en la susceptibilidad a R. solani entre los cultivares de arroz. Por lo anterior, se cree que la resistencia en el arroz está controlada por múltiples genes o loci de rasgos cuantitativos [QTL] (Jia et al., 2012; Zeng et al., 2014).
Una técnica para detectar genes de resistencia es la Selección Asistida por Marcadores Moleculares (SAM) que ayuda ahorrar tiempo, recursos y esfuerzo en los programas de mejoramiento de plantas. Esta técnica se puede aplicar en etapa de plántula, siendo útil cuando las características de interés se manifiestan en etapas posteriores de desarrollo de la planta, eliminando los genotipos no deseados en etapas tempranas, reduciendo el número de líneas a evaluarse en las etapas avanzadas del programa y aumenta la eficiencia del mejoramiento por realizarse la selección cuando existe mayor probabilidad de ligamiento entre el marcador y el QTL seleccionado. La SAM es una vía rápida por seleccionar plantas según su genotipo, al ser difícil distinguir por fenotipo las plantas homocigotas de las heterocigotas para el carácter deseado (Akhtar et al., 2010).
Para el uso de la SAM se requiere que los marcadores sean co-segregarse o mapearse lo más cerca posible del gen objetivo. Para mayor exactitud con el uso de la SAM deben usarse dos marcadores que flanquean el gen objetivo (Peng et al., 2000), los marcadores deben expresar polimorfismo entre los genotipos que presentan o no el gen objetivo. Se necesita que los marcadores PCR sean simples y económicos garantizando el poder de genotipiado para el cribado de grandes poblaciones.
Entre los marcadores moleculares están los denominados microsatélites, también llamados marcadores de secuencias simples repetidas (SSR), son actualmente la herramienta más completa para el SAM. Existen grandes colecciones de marcadores SSR ubicados tanto en la porción no codificada como expresada del genoma del arroz (Mohler y Singrun, 2004; Srinivasachary et al., 2011).
Para lograr el objetivo con la SAM, es importante comprender los mecanismos de resistencia a la PDV, tener un mapa detallado de los genes de interés y la clonación de los mismos. Existen dos problemas comunes para el uso de Selección Asistida por Marcadores Moleculares (SAM) con QTL de resistencia al PDV, (i) los marcadores de ADN identificados pueden no estar asociados a la resistencia a PDV en un germoplasma diferente al evaluado, y (ii) los métodos de evaluación fenotípica deben ser precisos para verificar la resistencia genética (Pan et al., 2005). De allí que el objetivo de esta investigación fue determinar en dos poblaciones segregantes de arroz, los marcadores moleculares polimórficos y su asociación con los genes de resistencia a R. solani.
Materiales y métodos
El estudio se localizó en las instalaciones del INIA-Portuguesa, Araure, estado Portuguesa (9º36’ N y 69º13’ O; altitud de 200 msnm), donde se conformaron las poblaciones experimentales a partir de los cultivares de arroz Palmar, Jefferson, Fonaiap 1 y Fonaiap 2000, se utilizaron como progenitores resistentes los cultivares Palmar y Jefferson y como susceptibles los cultivares Fonaiap 1 y Fonaiap 2000, estos parentales fueron elegidos de acuerdo con los resultados obtenidos por Delgado y Rodríguez (2005), realizándose los cruces entre Fonaiap 1 x Palmar y Fonaiap 2000 x Jefferson. Mientras que la extracción del ADN de los progenitores y su descendencia, así como también la amplificación y evaluación de los diferentes SSR seleccionados, se efectuó en el Laboratorio de Biotecnología del INIA-CENIAP.
Las plantas segregantes y sus progenitores se mantuvieron en potes cada una por separado, realizándose la fertilización comercial, desmalezado a mano y control de insectos plagas con insecticidas, los controles de enfermedades, de ser necesarios, se realizaron con fungicidas específicos no recomendados para R. solani.
De la descendencia F2 de cada cruce se tomaron 100 plantas, evaluándose la reacción a R. solani, tanto en los progenitores como en sus descendencias, mediante inoculaciones artificiales realizadas en 3 macollos.planta-1 en condiciones de umbráculo y cultivadas en potes de 11.775 cm3 contentivos de suelo estéril. Se usó como inoculante de R. solani el aislamiento 101-18, recolectado de plantas enfermas en campos de producción comercial, registrado, identificado y preservado en el laboratorio de Fitopatología del INIA-Portuguesa.
Las hojas de las. plantas de arroz se enumeraron del 1 al 5, siendo la número 1 la hoja bandera, evaluándose las lesiones en la segunda, tercera, cuarta y quinta hoja por ser estas las más indicadas para determinar la severidad de la infección (Eizenga et al., 2002). Las plantas se inocularon en el estado de crecimiento R4 (inicio de la exerción de la panícula), colocando un disco de agar contentivo de micelio del R. solani en el nudo dentro de la vaina de la hoja, después de la inoculación las plantas se llevaron a una cámara húmeda y se dejaron incubadas por una semana (Eizenga et al., 2002).
Luego de este tiempo, se realizaron las evaluaciones en los 3 macollos, planta-1 inoculados y se promedió su valor, utilizando la escala del sistema estándar de evaluación del Instituto Internacional de Investigación del Arroz (IRRI, 2002), midiendo el avance de las lesiones (Avance (%) = (AM/AP)*100), donde AM= es la suma de la longitud del área afectada por la PDV en el macollo inoculado y AP= altura del macollo, medido desde el suelo hasta el nudo de la segunda hoja, determinándose así el porcentaje de área afectada de las plantas por la enfermedad PDV (Yadav et al., 2015). Posteriormente las plantas se agruparon según la escala de 0 a 9 donde 0 = sin lesión y 9 = lesiones cubriendo del 90 al 100% del área. La escala se basa en la altura relativa de la lesión, pero por realizarse en el presente trabajo inoculaciones artificiales y proveer mejores condiciones para el desarrollo de la enfermedad, la escala se modificó en 10 valores con intervalos de 10%, es decir 0, 10 y 20% y así sucesivamente
Las plantas inoculadas se clasificaron según la escala de evaluación en resistentes (R) y susceptibles (S), representándose en una matriz. El criterio para dicha clasificación fue el siguiente, todas las plantas con valores entre 0 y 4 fueron consideradas resistentes, mientras que las que tuvieron valores entre 5 y 9 se consideraron susceptibles, se consideran resistentes hasta un valor de 45% de área afectada, superior a este valor se consideró susceptible (Yadav et al., 2015). Para el análisis de la segregacion de la resistencia a R. solani, se usó la prueba de bondad de ajuste Chi-cuadrado χ2 .
Antes de la inoculación de las plantas para su evaluación, estas se colocaron en umbráculo y cuando alcanzaron el máximo macollamiento (45-60 dds), se recolectaron 7 hojas. planta-1 para la extracción del ADN, realizado según el método de Zambrano et al. (2002). La calidad y cantidad del ADN fueron observadas en gel de agarosa 0,8% teñido con bromuro de etidio, bajo luz UV, comparando con estándares comerciales de concentración conocida.
Para determinar la asociación entre los microsatélites y su asociación con el gen o los genes de resistencia presentes en los cultivares progenitores y sus segregantes, se seleccionaron las secuencias y posición de 100 marcadores SSR polimórficos en los cuatro progenitores y con una separación de 10 cM aprox de la base de datos GRAMENE y fueron sintetizados en IDT, Inc. (Coralville, IA, EE.UU).
Los extractos de ADN obtenidos, se sometieron a PCR usando los iniciadores de los microsatélites o de secuencias simples repetidas (SSR) seleccionados, bajo las siguientes condiciones de reacción: volumen final 15 µL, extracto de ADN 1,5 µL 10 ng.mL-1, cebadores (primers) 0,6 µL (10 µM, sentido y contrasentido), mezcla de dNTPs 0,3 µL (10 mM), Tampón 10X PCR MgCl2-11.51,5 µL, Taq polimerasa 0,15 µL y agua desionizada estéril 10,95 µL (Pérez-Almeida et al., 2011)
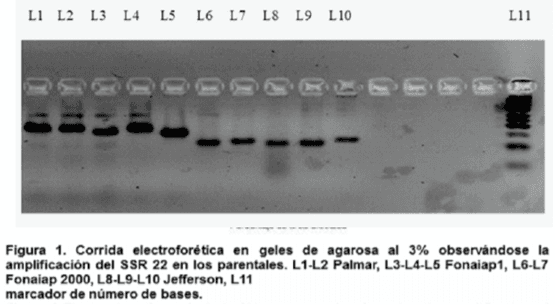
El programa de amplificación se realizó en un termociclador PTC-100 Programmable Termal Controller MJ Research, Inc., durante 34 ciclos a una temperatura de alineación de 55 °C (Pérez-Almeida et al., 2011). Una vez amplificado el ADN, las muestras se sometieron a corridas electroforéticas en geles de agarosa a una concentración del 3% usando el tampón de corrida TBE 1X en una cámara de electroforesis horizontal, con el fin de observar el polimorfismo de los microsatélites evaluados. Se observaron los productos de PCR amplificado que fueron mayores de 50 pb (figura 1). Los productos de la PCR fueron visualizados en un transiluminador UV, (marca BIORAD modelo CHEMIDOC, utilizando el programa Quantity One v. 4.2®, California USA) previa electroforesis en geles de agarosa al 3% en TBE y tinción con bromuro de etidio.
Para detectar la asociación entre los genes de resistencia a R. solani en arroz y los 100 marcadores SSR evaluados, se usó el programa Windows QTL Cartographer®, versión 2.5 (Wang et al., 2007), utilizando los productos de PCR visualizados en los geles de agarosa se preparó una matriz para asignarle el alelo correspondiente a cada SSR seleccionado a cada progenitor y progenie F2. El promedio del porcentaje de área afectada de cada individuo de la población F2 y la función de Kosambi fueron utilizados para determinar la distancia de mapeo por intervalo compuesto (CIM) (Díaz, 2005).
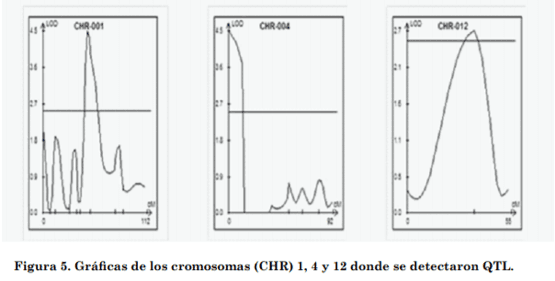
El programa Windows QTL Cartografer®, se usó con los parámetros predeterminados del modelo 6, modelo estándar, con cinco marcadores fijados previamente asociados al carácter a través del método de regresión paso a paso y una longitud de ventana de 10 cM a los lados del intervalo. Para declarar la asociación entre el fenotipo y el marcador se usó el umbral de LOD ≥2,4 (P= 0,05).
Resultados y discusión
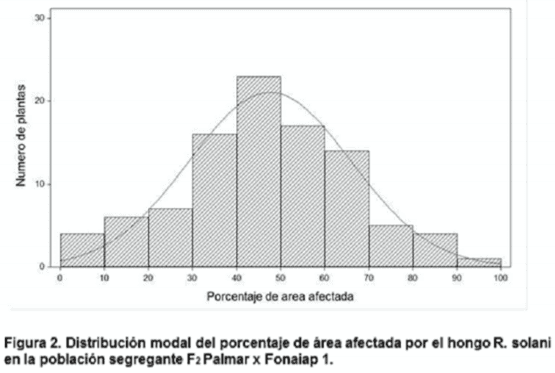
De acuerdo con la escala de evaluación los cultivares Palmar (11%) y Jefferson (32%), resultaron resistentes, mientras que los cultivares Fonaiap 1 (60%) y Fonaiap 2000 (67%) resultaron susceptibles, resultados que concordaron con los señalados por Delgado y Rodríguez (2005). La variación fenotípica a la reacción PDV entre los progenitores, medida por la diferencia del área afectada, fue de 49% entre los cultivares Palmar y Fonaiap 1 y de 35% entre los progenitores Jefferson y Fonaiap 2000. La población segregante F2 del cruce entre Palmar x Fonaiap 1 mostró una variación fenotípica a la reacción PDV de 83%, con un valor máximo de 91% y un valor mínimo de 8%. Mientras que en la población segregante F2 del cruce Jefferson x Fonaiap 2000 la variación fenotípica a la reacción PDV de 68%, siendo el valor mínimo 12%, y el máximo de 80% (cuadro 1).
En las progenies (F2 ) evaluadas se observó una variación continua en la reacción fenotípica producto de la inoculación artificial del hongo R. solani, además, ambas poblaciones mostraron segregación transgresiva, determinada en la descendencia por la reacción fenotípica que mostró individuos más resistentes o más susceptibles que los respectivos parentales; la herencia transgresiva es considerada el efecto principal, producto de la acción complementaria de genes presentes en ambos progenitores, siendo esta una característica cuantitativa condicionada por varios genes.

En este mismo sentido, se ha determinado la ausencia de resistencia completa en arroz a R. solani (Pan et al., 1999; Mew et al., 2004; Jia et al., 2012), considerándose que la resistencia en arroz al hongo es multigénica, siendo difícil evaluar el efecto individual de los genes que confieren esta resistencia (Che et al., 2003). Además, Srinivasachary et al. (2011) señalaron como hipótesis que existen dos mecanismos principales y distintos que contribuyen a la resistencia a la PDV, una es la resistencia fisiológica, que es independiente de los rasgos morfológicos y la segunda es el escape de la enfermedad atribuida a caracteres morfológicos, especialmente la altura de la planta, o la fecha de emisión de panícula. En el presente trabajo se establece la resistencia fisiológica por haberse hecho inoculaciones artificiales a las plantas en la misma etapa de crecimiento (R4).
Además, la existencia de herencia transgresiva en ambas progenies segregantes, explicó por qué en estas poblaciones se observó una segregación no ajustada a las leyes de herencia de Mendel, siendo la proporción determinada 1 resistente:1 susceptible (1:1); por esto la resistencia a PDV estuvo condicionada por varios genes.
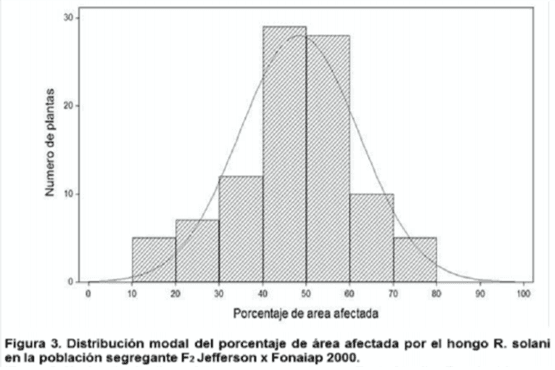
La reacción fenotípica de las dos poblaciones segregantes F2 al hongo R. solani se ajustaron a una distribución normal (figura 2 y 3). La población F2 derivada del cruce Palmar x Fonaiap 1 mostró, de acuerdo a la fórmula de probabilidad para normalidad de Shapiro-Wilk, una variación continua con un valor de W= 0,9862 y P(W)= 0,4084, mientras que la población F2 obtenida del cruce Jefferson x Fonaiap 2000 presentó un valor de W= 0,9757 y un valor de P(W)= 0,1052, siendo para ambas poblaciones el valor W mayor que el valor P(W), por lo tanto se ajustaron a una distribución normal. Por lo anterior, el análisis para la asignación de QTL se realizó manteniendo para cada planta su valor en porcentaje de área afectada (severidad).
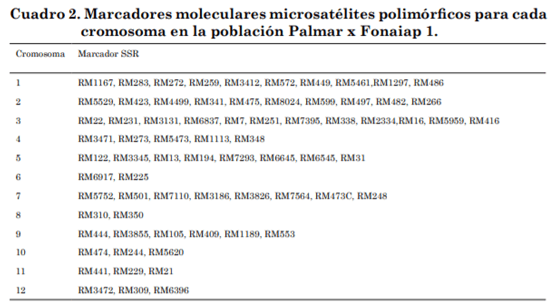
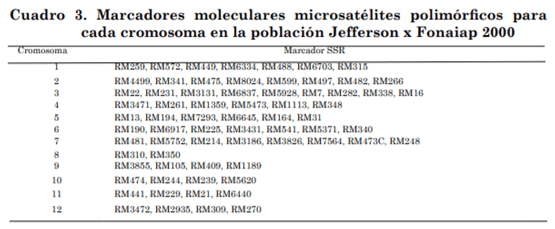
Las dos poblaciones segregantes evaluadas mostraron polimorfismo para los SSR analizados, resultando polimórficos en la descendencia de Palmar x Fonaiap1 70 SSR y pára Jefferson x Fonaiap 2000 69 SSR de los 100 SSR seleccionados. Se muestra la distribución en los cuadros 2 y 3, respectivamente.
En todos los marcadores SSR polimórficos evaluados, la segregación para ambas poblaciones fue 1:2:1 siguiendo una herencia de acuerdo con las leyes de Mendel para codominancia, pudiéndose diferenciar las plantas homocigotas de los heterocigotas para cada marcador SRR.
En la descendencia segregante del cruce Jefferson x Fonaiap 2000 no se detectó la presencia de QTL, por ubicarse la asociación entre los marcadores SSR y la resistencia a PDV por debajo del límite LOD= 2,4 (P= 0,05). Mientras que en la población segregante producto del cruce Palmar x Fonaiap 1 se detectó asociación entre los marcadores SSR y la resistencia a R. solani.
Lo anterior, puede explicarse por la divergencia fenotípica que presentó la población segregante Jefferson x Fonaiap 2000 la cual fue menor a la mostrada por la población segregante Palmar x Fonaiap 1 (cuadro 1). Además, al comparase los resultados estadísticos con los obtenidos en la población segregante Palmar x Fonaiap 1, mostró una desviación estándar menor, lo que indicó que los datos estuvieron agrupados cerca de la media, una varianza menor, por lo que se necesita aumentar el tamaño de la población experimental para poder detectar los QTL con LOD > 2,4, lo que concordó con lo señalado por Semagn et al. (2006).
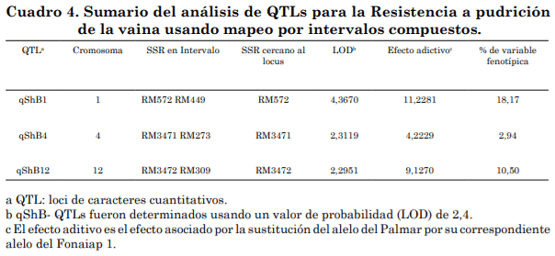
En la descendencia del cruce Palmar x Fonaiap 1, se detectó asociación entre marcadores SSR y la resistencia a R. solani t se identificaron tres QTL para la resistencia a ShR, qShB1, qShB4 y qShB12, localizados en los cromosomas 1, 4 y 12, respectivamente.
Los alelos resistentes provenientes del cultivar Palmar, mostraron los siguientes resultados, el qShB1 explicó 18,17% de la variación fenotípica total, con un efecto aditivo de 11, 2281; el qShB4 explicó el 2,94% de la variación fenotípica total, con un efecto aditivo de 4,2229, mientras que el qShB12 explicó el 10,5% de la variación fenotípica total, con un efecto aditivo de 9,1270 (cuadro 4). Los marcadores microsatélites RM572 (36,2 cM) y RM449 (50,8 cM) flanquearon el QTL qShB1, los microsatélites RM3471 (0,0 cM) y RM273 (53,4 cM) flanquearon el QTL qShB4 y los microsatélites RM3472 (0,0 cM) y RM309 (35,3 cM) flanquearon el QTL qShB12 (figura 4 y 5). Los alelos qShB1 y qShB12 explicaron el 28,67% de la variación fenotípica total, con un efecto aditivo de 20,36%; siendo importante detectar estos dos qShB en la descendencia de los cruces Palmar x Fonaiap 1.
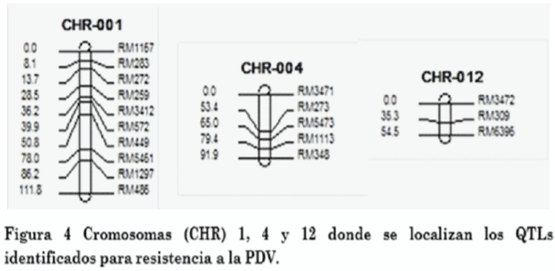
La detección de tres QTL en la población Palmar X Fonaiap1 ubicados en diferentes cromosomas, también constituyeron un factor que explicó el no haberse observado una segregación ajustada a las leyes de la herencia de Mendel.
Srinivasachary et al. (2011) señalaron dos mecanismos que contribuyeron a la resistencia a PDV, una fue el escape de la enfermedad, atribuida a caracteres morfológicos y la segunda fue la resistencia que fue independiente de estos rasgos. La inoculación artificial de R. solani bajo condiciones controladas en R4, evitó que las respuestas de resistencia se asociaran con otras variables como las ambientales (Lu et al., 1996), la altura de la planta y la emisión de la panícula, que confundieron la asociación entre la respuesta de resistencia y los genes cuantitativos (Zou et al., 2000; Sato et al., 2004; Jia et al., 2009; Zeng et al., 2014). Así mismo, se señaló que los QTL para resistencia a la PDV y varios QTL para caracteres morfológicos en arroz se localizaron en el mismo intervalo (Pan et al., 2005); siendo, probablemente, la inadecuada forma de evaluar la respuesta de la planta a la PDV, la razón principal de los resultados inconsistentes entre los reportes (Han et al., 2003).
Zuo et al. (2008) demostraron el potencial que tienen los marcadores moleculares para la clonación e implementación del uso de genes de resistencia QTL en el mejoramiento del germoplasma en arroz entre subespecies. Además, se ha reportado que cultivares de arroz susceptibles poseen genes que confirieron resistencia a la PDV y los padres susceptibles portadores de estos genes fueron una fuente valiosa para el desarrollo de nuevas líneas con resistencia a la PDV (Zuo et al., 2011). Por otro lado, han sido identificados 11 genes de quitinasa de clase III en el QTL mapeado qSBR11-1 en el cultivar de arroz Tetep que se expresaron después de la inoculación con R. solani, lo que ayudó en el proceso de mejoramiento del arroz (Richa et al., 2016; Richa et al., 2017)
Con el uso de la técnica SAM, se seleccionaron genotipos deseados manifestados al final del proceso de mejoramiento, obteniéndose líneas con resistencia durable y de amplio espectro (Pastor-Corrales et al., 1998). Por último, los tres QTL detectados en el presente trabajo explicaron en total el 31,81% de la variación fenotípica total y constituyen un valioso aporte al estudio de la resistencia a la PDV.
Conclusión
Las inoculaciones artificiales en la etapa de crecimiento del arroz R4, es una técnica muy útil para identificar la asociación de las respuestas fenotípicas de la reacción a la PDV en arroz con genes de resistencia (QTL) presentes en los diferentes cultivares, encontrándose en este estudio tres regiones del ADN del arroz ubicados en los cromosomas 1, 4 y 12 que contribuyen en la consecución de cultivares de arroz con resistencia genética que permita junto con el control químico la disminución de las pérdidas ocasionadas por el hongo R solani y tendrá un gran impacto en la estabilidad del rendimiento, la sustentabilidad y la productividad del cultivo del arroz.