Parámetros poblacionales de la raza Brangus
Estimación de parámetros poblacionales de la raza Brangus argentina mediante un análisis de genealogía
Publicado: 22 de febrero de 2023
Por: Fassa V.B.1* y Munilla S.2,3
1 Universidad de Buenos Aires, Facultad de Ciencias Veterinarias, Cátedra de Genética; 2 Universidad de Buenos Aires, Facultad de Ciencias Agrarias, Departamento de Producción Animal; 3 INPA. CONICET-FAUBA.
Introducción
Conocer la estructura, la consanguinidad y el tamaño efectivo de una población, entre otros parámetros, es importante para caracterizarla y así prevenir pérdidas en la diversidad genética de una población bajo selección. El objetivo de este trabajo fue estimar estos parámetros mediante un análisis de genealogía a través del programa Endog (Gutiérrez y Goyache, 2005). El análisis se llevó a cabo para un grupo representativo de individuos de la raza Brangus. Estos constituirán la base de una población simulada a través de la cual se ensayarán diferentes estrategias de genotipificación en el contexto de un programa de selección genómica.
Materiales y Métodos
Se utilizó la base de registros fenotípicos del programa de Evaluación de Reproductores Brangus (ERBra) a su estado luego de la evaluación 2021. Si bien esta tabla de datos constaba de 382.159 registros, no se trabajó con todos ellos, sino que se definió un subconjunto de 29.207 individuos que representa nuestra población objetivo o de referencia, obtenidos al aplicar los siguientes criterios:
a. Que procedan de cabañas argentinas que hayan reportado datos fenotípicos entre 2011 y 2020 ininterrumpidamente.
b. Que ambos padres estuvieran registrados.
c. Que hayan nacido entre 2016 y 2020.
Luego, se extrajo la genealogía completa de los individuos asociados a estos registros aplicando un recorte o “pruning” con el programa relaX2 de Ismo Strandén sobre los 559.583 individuos que integran el pedigree de la raza. El criterio para realizar el recorte fue retener a todos los individuos que contribuyeron con un registro fenotípico y los ancestros que los conectaban. En total, se obtuvo una genealogía de 56.073 individuos. La idea detrás de estos filtros fue la de obtener una genealogía representativa de la raza con un número mucho más acotado de individuos.
Con este grupo se llevó a cabo el análisis de la genealogía a través del programa Endog v.4.8 y se obtuvieron estimaciones de los parámetros poblacionales.
Resultados y Discusión
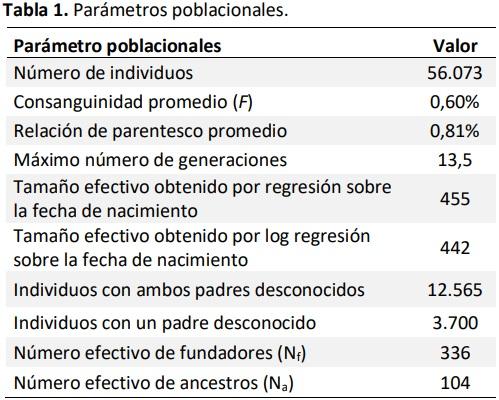
De los 56.073 individuos hay 17.038 machos y 39.035 hembras. Este número refleja el desbalance en los tamaños de familia de toros y vacas, dado que en la población objetivo la distribución de los sexos es pareja.
En la Tabla 1 se presentan las estimaciones de los parámetros poblacionales obtenidos. El 22% de los animales (12.565) tienen ambos padres desconocidos y, en consecuencia, forman la población fundadora. Sin embargo, la contribución de estos progenitores a la población objetivo es desbalanceada y una medida más apropiada es el número efectivo de fundadores (Nf), que representa el número teórico de animales con padres desconocidos que, contribuyendo balanceadamente, generarían la diversidad genética observada en la población bajo estudio.
Un parámetro similar, aunque no idéntico es el número efectivo de ancestros (Na), que representa el número mínimo de ancestros, sean fundadores o no, necesarios para explicar diversidad genética en la población objetivo. En este caso, los valores de Nf y Na fueron, respectivamente de 336 y 104 individuos. Ambos fueron calculados con la contribución de los individuos de la población de referencia. Estos valores son comparables a los reportados por Ron Garrido et al. (2008) en la misma población hace más de una década (Nf = 766 y Na = 388). Estos valores son altos si se comparan con los reportados para otras razas de bovinos de carne y esto se puede deber a la política abierta de registración de la raza Ron Garrido et al. (2008).
En ese mismo trabajo, se reporta una consanguinidad promedio de 0,24%. Aquí obtuvimos un valor de 0,60%, apenas superior. Como es de esperar, la consanguinidad promedio por pedigree en una población como la del Brangus argentino está subestimada en gran medida. En un trabajo reciente, Forneris et al. (2021) desagregaron la consanguinidad por año de nacimiento y reportaron un aumento anual, entre 1950 y 2019, de ΔF = 0,039%. En los animales nacidos en 2019, la consanguinidad acumulada estimada por pedigree fue del 4%. Por genómica, según reportan, esta cifra ascendió al 13%.
Referencias
Forneris NS (2021). J. Anim. Sci. 1; 99 (11)
Gutierrez y Goyache. 2005. J. Anim. Breed. Genet. 122, 172- 176.
Ron Garrido (2008). Livestock Science 117, 43-51.
Contenido del evento:
Temas relacionados:
Autores:
Universidad de Buenos Aires
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.




.jpg&w=3840&q=75)