Secuenciacion de cepas virales de diarrea epidemica porcina aisladas en diversas regiones de México
Publicado: 21 de septiembre de 2015
Por: Abraham Massa Peniche, Marta Macías, Flores A, Chávez A, Ramírez G, Raya R, Alfredo Franco. Laboratorio de Diagnóstico. Lapisa SA de CV
Introducción
Desde inicios del año 2104 en que en el Laboratorio de Diagnóstico de Lapisa se comenzaron a realizar los primeros aislamientos exitsos del virus de la Diarrea Epidémica Porcina (DEP), los títulos obtenidos en pases en cultivo celular marcaron el camino natural siguiente: la secuenciación de las cepas aisladas. Los análisis de las secuencias de las cepas proveerían información sobre la presencia de virus INDEL, cepa colorado y/o posible presencia de cepas chinas.
Material y métodos
Se tomaron 8 muestras de cepas del virus DEP aisladas y replicadas exitosamente en el laboratorio de Diagnóstico de Lapisa y se procesaron para su secuenciación. El material genético se extrajo mediante el kit QIAamp® viral RNA minikit, posteriormente se purificó mediante el kit QIAquick® PCR purification kit, se cuantificó en un espectrofotómetro NanoDrop 2000 Thermo Scientific, y se procedió a la secuenciación.
Para la secuenciación se utilizaron primers forward y reverse marcados con IRdye en 700 y 800 nm y ésta se realizó en un equipo Li-cor 4300 DNA Analizer. Los fragmentos analizados fueron de 651-bp, 833-bp y 808 bp respectivamente para el gen Spike (S), membrana (M) y ORF3.
Resultados y discusión
Los resultados de secuenciación de los fragmentos anteriormente mencionados se analizaron y se compararon entre si. Las secuencias del gen Spike (S) considerada la parte variable de la estructura del virus fue la que se comparó entre los diversos aislados.
Para el análisis de las secuencias obtenidas se utilizó el programa BioEdit Sequence Alignement Editor 7.2.5.
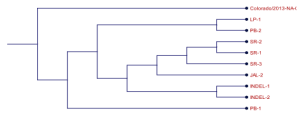
Se elaboró el árbol filogenético correspondiente.
Figura1: Árbol filogenético secuencias gen Spike (s)
Conclusiones
Como se puede observar en el árbol, las secuencias parciales del gen Spike (s) nos muestran ciertas diferencias entre los aislamientos nacionales vs la cepa INDEL y la cepa colorado.
Entre los aislamientos replicados exitosamente en cultivo celular se ha detectado una cepa similar a la cepa INDEL.
Se continúan las secuenciaciones de las cepas detectados en el laboratorio de Diagnóstico para tener un mapa más amplio de la distribución de estas en el país.
Referencias
1. Ruiging Sun, Zhangming Leng, Shao-Lun Zhai, Dekum Chen, Changxu Song. 2014. Genetic Variability and Phylogeny of Current Chinese Porcine Epidemic Diarrhea Virus Strains Base don Spike, ORF3 and Membrane Genes. Scientific World Journal. 2014; 2014.
2. Vlasova A, Marthaler Douglas, Wang Qiuhjong, Culhane Marie, Rossow Kurt, Rovira Albert, Collins James and Saif Linda. 2014. Distinct Characteristics and Complex Evolution of PED Strains, North America, May-2013-February 2014. Emerging Infectious Deseases. Vol 20, Nº 10
Temas relacionados
Autores:
Únete para poder comentar.
Una vez que te unas a Engormix, podrás participar en todos los contenidos y foros.
* Dato obligatorio
¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Crear una publicación




