Introducción
El síndrome reproductivo y respiratorio porcino (PRRS) es una enfermedad de distribución mundial que genera pérdidas económicas en la industria porcina estimadas aproximadamente en $3.08 dólares americanos por cerdo al mercado(1).
El agente etiológico del PRRS es un virus ARN de cadena sencilla que pertenece al género Arterivirus, cuyas principales características son su elevada tasa de mutación, que le confiere una alta variabilidad antigénica, y su capacidad para inducir infecciones persistentes(2,3) . El reporte inicial de la enfermedad del PRRS en México describió que las cerdas infectadas mostraban un cuadro de problema reproductivo y mortalidad en la línea de producción(4). La primer descripción clínica, epidemiológica y productiva de la enfermedad, así como el primer aislamiento del virus del PRRS, fueron reportados en los estados de México, Guanajuato, Veracruz y Puebla(5) .
La infección por el virus del PRRS se caracteriza por pobre conversión alimenticia que se traduce en bajos pesos de los cerdos en general, así como alteraciones en la fertilidad en hembras reproductoras tales como repetición del estro, mortalidad fetal, momificación, inducción de abortos y baja viabilidad de lechones al nacimiento(1) .
La vacunación es el método más común para el control del PRRS y a la fecha ha logrado prevenir en cierta medida la infección por PRRS. Sin embargo, la eficacia de las vacunas aún se encuentra lejos de ser universal, ya que el virus posee la habilidad para evadir la respuesta inmune del hospedador, y además, entre hospedadores existen diferencias de origen genético en su respuesta ante la presencia del virus(6,7) . La vacunación puede tener cierta eficacia cuando se trata de cepas homólogas, pero al tratarse de cepas heterólogas su efectividad se reduce drásticamente. Por lo tanto, actualmente vacunar contra el virus del PRRS sólo garantiza una reducción en la duración de la viremia y del período de eliminación del virus, así como una disminución en el grado de presentación de signos y síntomas clínicos, pero no suprime la enfermedad(8).
La existencia de variantes génicas asociadas a la interacción entre el virus del PRRS y su huésped, así como la evidencia de una variabilidad natural en la tolerancia y/o susceptibilidad al PRRS en las líneas porcinas comerciales, han abierto la puerta al uso de tecnologías moleculares como una valiosa herramienta para combatir la enfermedad del PRRS(9). Al respecto, la selección asistida por marcadores moleculares (SAM) puede emplearse para identificar, a través del estudio de genes candidatos, a aquellos animales que poseen una habilidad genética superior para la expresión de caracteres de importancia económica, entre los cuales se incluye la resistencia o tolerancia a enfermedades(10). Los primeros ejemplos de la aplicación de estas tecnologías en cerdos fueron la selección en contra del gen del halothano, y la identificación de una asociación significativa entre el gen del receptor del estrógeno y el número de lechones nacidos vivos(11). Sin embargo, el desarrollo actual de sistemas de cómputo más robustos ha facilitado la selección genómica completa, la cual involucra el uso extensivo de marcadores moleculares que cubren la totalidad del genoma, de tal manera que cientos de miles de variantes moleculares pueden ser estudiadas al mismo tiempo a fin de explicar la variación genética de una característica fenotípica(12). Este método permite realizar estudios asociativos que incluyen el análisis de una gran cantidad de marcadores en forma simultánea a través del uso de dispositivos de baja (10k=10,000 SNP) o alta densidad (50k=50,000 a 60k=60,000 SNP).
Diversas investigaciones se han realizado en cerdos con el fin de identificar regiones en el ADN relacionadas con rasgos de interés económico, entre los cuales se encuentra la resistencia al virus del PRRS(13,14,15). Reportes iniciales sugirieron la existencia de base genética asociada a la enfermedad del PRRS. Al respecto, cerdos de la raza Hampshire infectados con PRRS mostraron lesiones pulmonares más graves que cerdos de las razas Duroc y Meishan(16). Por otra parte, cerdos de la línea sintética Large White-Landrace mostraron una menor temperatura rectal y una reducción en la viremia después de ser infectados con el virus del PRRS, en comparación con cerdos de la línea sintética HampshireDuroc(17). Recientemente, se reportó una región génica dentro del cromosoma 4 asociada a la resistencia al virus del PRRS, lo cual evidenció la existencia de un fuerte componente genético asociado a esta habilidad(3) .
Sin embargo, actualmente es escasa la información reportada sobre genes y variantes génicas relacionadas a las diferencias fenotípicas observadas en la eficiencia reproductiva de hembras infectadas con el virus del PRRS. Por lo tanto, el análisis de la base genética de la respuesta reproductiva de dichas hembras, podría conducir a la identificación de marcadores genéticos asociados a un adecuado comportamiento reproductivo, lo cual sería de gran utilidad para la implementación de programas más eficientes de selección, que incluyan cerdas con habilidad genética superior para tolerar y/o resistir la infección del virus del PRRS.
En base a lo anterior, el objetivo del presente estudio fue identificar polimorfismos de nucleótido simple asociados al número de lechones nacidos vivos al primer (LNV1) y segundo parto (LNV2) en cerdas reproductoras infectadas con el virus del PRRS.
Material y métodos
Sitio y unidades experimentales
El presente estudio se realizó en una granja porcina comercial de ciclo completo ubicada en el valle del Yaqui, Sonora, México (LN: 27°17’, LO: 109°56’). Se utilizaron 100 cerdas reproductoras de la línea comercial Landrace(¾)/Yorkshire(/¼), de 12 meses de edad y libres de la enfermedad del PRRS.
Manejo sanitario y reproductivo
A los 15 días posteriores a su ingreso al área de pie de cría, 75 cerdas resultaron infectadas en forma natural por el virus del PRRS (positivas; n= 75), debido a que, aunque la granja era seronegativa a PRRS, ésta se encuentra ubicada en una región endémica afectada por diversas cepas de linaje norteamericano (PRRSV NA). Por otra parte, un grupo control negativo integrado por 25 hembras se mantuvo libre de la infección del PRRS (control; n= 25). Lo anterior fue confirmado por pruebas serológicas y moleculares realizadas desde el inicio y hasta el final del experimento. Dentro del área de pie de cría, las hembras iniciaron su manejo reproductivo que consistió en proporcionarles dos servicios con verracos de fertilidad probada después de ser observadas en estro. Una vez confirmadas como gestantes, las cerdas fueron trasladadas al área de gestación y permanecieron ahí hasta el día previo a su parto, momento en el cual fueron movilizadas al área de maternidad. Inmediatamente después del parto se contabilizó el número de lechones nacidos totales, nacidos vivos y nacidos muertos; los datos resultantes fueron registrados dentro del programa de cómputo PigWIN®. El mismo manejo reproductivo y registro de información antes descrito se repitió para la segunda cubrición de cada hembra y su parto correspondiente.
Análisis de laboratorio
Muestras de sangre fueron colectadas en forma individual por punción de la vena auricular a los días 7, 30, 120 y 240 posteriores al ingreso de las hembras al área de pie de cría, para la determinación sérica de los títulos de anticuerpos específicos contra el virus del PRRS usando la herramienta diagnóstica “ELISA-IDEXX” (Enzyme Linked Immunoassay, Lab Inc.). El aislamiento de ARN viral se realizó a partir del suero sanguíneo mediante un sistema de extracción automática de ácidos nucleicos por separación magnética (TACO System, Gene Reach Biotechnology Corporation). Con las muestras de ARN purificadas se realizó el análisis de PCR en tiempo real utilizando un Kit comercial (Tetracore Nextgen Real-Time QT-PCR), el cual reconoce un segmento del ORF 7 del virus del PRRS y cuyos resultados se reportan como número de copias de ARN del virus del PRRS por mL de muestra (Cepheid Smart Cycler V2.0d).
Estudio asociativo del genoma completo
Se obtuvo una muestra adicional de sangre (0.5 ml) de cada hembra la cual fue colocada en tarjetas FTA para recolección de ácidos nucleicos. Las tarjetas se almacenaron a 25°C y posteriormente fueron enviadas al Laboratorio Neogen donde se realizó la extracción, purificación y cuantificación del ADN para cada una de las hembras muestreadas. El ADN fue sometido a un proceso de genotipado utilizando un dispositivo de perfil genómico de baja densidad (Low Density Porcine BeadChip, Neogen®, Lincoln, NE) con capacidad para análisis de 10,000 polimorfismos de nucleótido simple (SNP). Una vez obtenidos los resultados, se utilizó el software PLINK (V1.07)(18) para el análisis de calidad y depuración de genotipos que consistió en la remoción de los SNP con tasa de genotipado por debajo del 90 %, frecuencia del alelo menor inferior al 5 % y tasa de error Mendeliano superior a 0.1. Al finalizar el estudio de calidad, un total de 8,826 SNP resultaron útiles e informativos para el estudio asociativo de genoma completo. Para ello, un modelo mixto multi-locus fue construido con el fin de identificar SNP asociados a los caracteres reproductivos de LNV1 y LNV2 utilizando el programa Golden Helix SVS 7 (Golden Helix Inc., Bozeman, Montana, USA). Al procesar el modelo asociativo antes mencionado, se utilizó el procedimiento “stepwise” para identificar los SNP significativos como covariables de efectos fijos. Además, el modelo permitió la utilización de una matriz de relaciones genómicas estimada a partir de los genotipos disponibles (SNP) para cada animal. Se consideró a los SNP como asociados a los fenotipos de interés a un α=0.001, y todos aquellos que resultaron significativos (P<0.001) fueron retenidos para su análisis de validación.
Análisis estadístico
Las estadísticas descriptivas para las variables de lechones nacidos totales, nacidos vivos y nacidos muertos se calcularon con el procedimiento MEANS del paquete estadístico SAS versión 9.4 (SAS Inst. Inc., Cary, NC). Un análisis de varianza fue utilizado para determinar si las variables antes mencionadas diferían entre hembras positivas y hembras negativas al PRRS (P<0.05), usando el procedimiento PROC GLM. Las pruebas de normalidad e igualdad de varianza fueron realizadas usando el procedimiento UNIVARIATE(19) .
Validación de marcadores genéticos asociados a LNV1 y LNV2
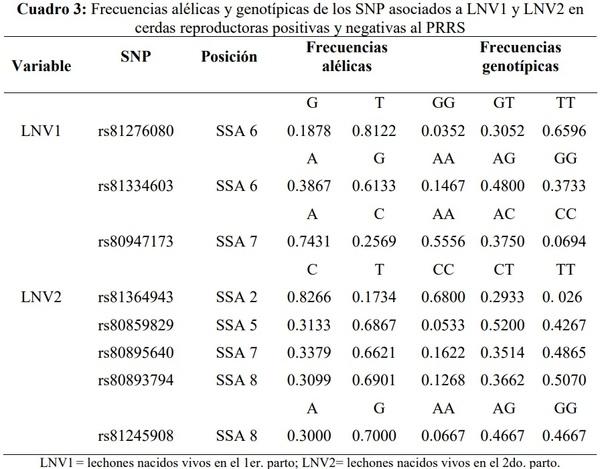
El cálculo de las frecuencias alélicas y genotípicas, así como la prueba Ji-cuadrada (X 2 ) para verificar posibles desviaciones del equilibrio Hardy-Weinberg, fueron desarrollados usando el procedimiento ALLELE(20). Los SNP que resultaron asociados (P<0.001) a los caracteres de LNV1 y LNV2, y que cumplieron con los criterios de frecuencia del alelo menor superior al 10 % (FAM>0.10) y no-desviación del equilibrio Hardy-Weinberg (X 2>0.05), fueron sometidos a un análisis de validación a través de un estudio asociativo entre genotipo y fenotipo, empleando el procedimiento MIXED para variables de distribución continua. Dicho análisis de validación individual para cada SNP se efectuó a través de un modelo de efectos mixtos, el cual incluyó los efectos fijos del genotipo del polimorfismo y la edad de la madre, el efecto aleatorio del semental (ej. usando la estadística Z para probar si Ho:δw 2=0), y el efecto residual (media=cero, varianza= δe 2 ).
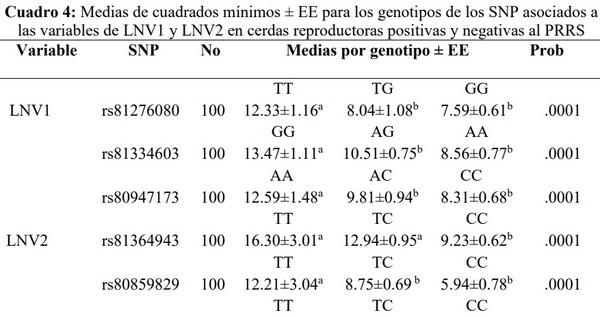
Las comparaciones entre medias para los genotipos de los SNP asociados con las variables de LNV1 y LNV2 fueron obtenidas utilizando la opción PDIFF del procedimiento LSMEANS, incluyendo el ajuste Bonferroni, siempre y cuando el término genotipo del SNP resultó ser fuente de variación (P<0.05) en el análisis asociativo. Los efectos de sustitución alélica fueron estimados a través del modelo de efectos mixtos descrito previamente, el cual para este análisis incluyó el término genotipo como covariable(21) .
Resultados
Variables asociadas al PRRS
En el Cuadro 1 se muestran las estadísticas descriptivas de los caracteres reproductivos en estudio, así como de variables referentes a la viabilidad de los lechones al nacimiento y al destete. Los valores promedio para las variables de lechones nacidos totales y nacidos vivos al primer y segundo partos fueron significativamente (P<0.01) menores para hembras positivas a PRRS, en comparación con hembras negativas (testigo), mientras que el efecto opuesto fue observado para la variable de lechones nacidos muertos, lo cual sugiere que dichas variables están asociadas a la infección del virus del PRRS en el presente estudio.
Estudio asociativo del genoma
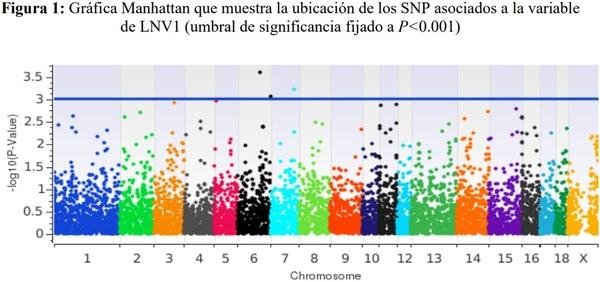
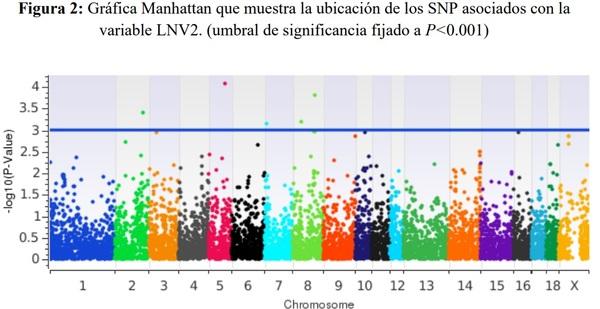
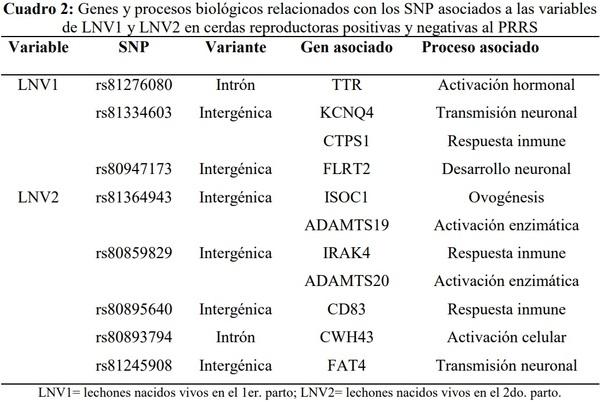
completo Un total de 8,856 SNP cumplieron con los criterios de calidad para ser incluidos en el análisis genómico asociativo, el cual detectó regiones genómicas asociadas a LNV1 (cromosomas 6 y 7; Figura 1) y a LNV2 (cromosomas 2, 5, 7 y 8; Figura 2), las cuales explican el 3.6 y el 4.1% de la varianza asociada a los caracteres de LNV1 y LNV2, respectivamente. El análisis genómico individual detectó tres SNP asociados a la variable de LNV1 (P<0.001) y cinco SNP asociados a la variable de LNV2 (P<0.001). Sólo dos de los ocho SNP antes mencionados se encuentran ubicados dentro de regiones génicas funcionales (intrones), el rs81276080 dentro del gen TTR (Transthyretin) y el rs80893794 dentro del gen CWH43 (Cell wall biogenesis 43 C-terminal homolog). El registro de los ocho SNP detectados como asociados a los caracteres de LNV1 y LNV2, así como los genes y procesos biológicos relacionados con dichos SNP, se muestran en el Cuadro 2.
Validación de marcadores genéticos
Los ocho SNP identificados por su asociación con las variables reproductivas de LNV1 y LNV2, cumplieron con los criterios de no-desviación del equilibrio Hardy-Weinberg (X 2=1.0, P>0.28) y de frecuencia del alelo menor superior al 10% (FMA>0.10; Cuadro 3), por lo que fueron considerados como genes candidatos en el presente estudio. Tres de estos SNP fueron validados como predictores de LNV1 (P<0.001) y cinco como predictores de LNV2 (P<0.001). El Cuadro 4 muestra las medias de cuadrados mínimos para los genotipos de cada uno de los SNP asociados a las variables de LNV1 y LNV2. Los SNP más favorables fueron rs81334603 y rs81364943 ya que mostraron los valores más altos para LNV1 (GG= 13.47 ± 1.11) y LNV2 (TT= 16.30 ± 3.01), respectivamente; mientras que los SNP menos favorables fueron rs81276080 y rs80859829 ya que mostraron los valores más bajos para LNV1 (GG= 7.59 ± 0.61) y LNV2 (CC= 5.94 ± 0.78), respectivamente. Sin embargo, cada uno de los ocho SNP validados en el presente estudio mostró un genotipo asociado favorablemente a los caracteres reproductivos analizados.

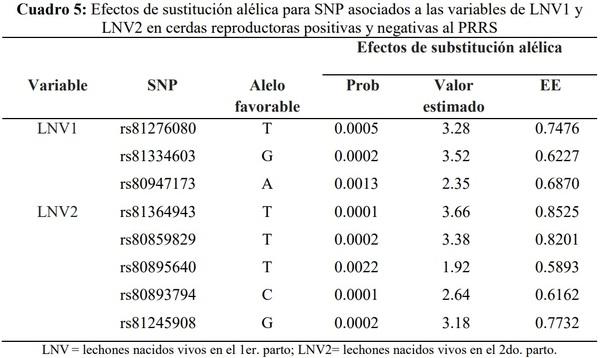
Los efectos de substitución alélica para cada SNP se muestran en el Cuadro 5. Los alelos favorables de los SNP rs81276080, rs81334603 y rs80947173 asociados a LNV1, fueron T, G y A, ya que elevan en 3.28 ± 0.74, 3.52 ± 0.62 y 2.35 ± 0.68 el número de LNV1, respectivamente (P<0.005). Por otra parte, para los SNP rs81364943, rs80859829, rs80895640, rs80893794 y rs81245908 asociados a LNV2, los alelos favorables fueron T, T, T, C y G, ya que incrementan en 3.66 ± 0.85, 3.38 ± 0.82, 1.92 ± 0.58, 2.64 ± 0.61 y 3.18 ± 0.77 el número de LNV2, respectivamente (P<0.005). Los valores antes descritos indican una contribución favorable de los ocho SNP sobre los caracteres reproductivos evaluados en las cerdas incluidas en el estudio.
Discusión
El efecto negativo de la infección por el virus del PRRS sobre el número de lechones nacidos vivos observado en el presente estudio ha sido previamente reportado en cerdas de diferentes paridades(22,23). En un estudio similar realizado en cerdas infectadas con el virus del PRRS que fueron comparadas con cerdas sanas, se observó un incremento en los valores promedio de cerdos momificados y no-nacidos de 0.04 a 1.13 y 0.63 a 1.02, respectivamente, así como una reducción en el número de cerdos nacidos vivos de 10.3 a 9.8(24) .
Por otra parte, la existencia de variabilidad genética asociada al desempeño reproductivo de cerdas infectadas con el virus del PRRS también ha sido descrita en previas investigaciones. Al respecto, Rashidi et al(15) reportaron una variación de 3.83 ± 0.31 en el número de lechones nacidos vivos en cerdas infectadas con el virus del PRRS, en comparación con una variación de 1.96 ± 0.06 en el número de lechones nacidos vivos en cerdas sanas. La variabilidad antes mencionada, principalmente en las cerdas infectadas con PRRS, sugiere la existencia de una base genética asociada a la respuesta reproductiva ante la enfermedad. Por lo anterior, se ha señalado que una estrategia interesante para reducir el impacto negativo del PRRS en hembras reproductoras, es la identificación de marcadores moleculares que permitan un mejor entendimiento del control genético de la respuesta al virus, para eventualmente utilizarlos en la implementación de programas de selección asistida por marcadores (SAM)(25). .
En el presente estudio, el análisis del genoma completo identificó regiones genómicas y un total de ocho SNP asociados a las variables de LNV1 y LNV2 (P<0.001) en cerdas reproductoras infectadas y no infectadas con el virus del PRRS. Dichos SNP mostraron una frecuencia del alelo menor superior al 10%, lo cual en términos generales es considerado como un requisito para evitar resultados falsos en estudios asociativos entre genotipo y fenotipo(26) .
En el análisis estadístico individual, los ocho SNP identificados fueron validados como predictores de las variables LNV1 y LNV2, y de los cuales sólo dos de ellos se encuentran dentro de regiones funcionales. Por una parte, el SNP rs81276080 (asociado a LNV1) se localiza en la región 5’ del gen TTR (Transthyretin), el cual codifica la síntesis de una proteína transportadora de hormonas tiroideas en plasma y fluido cerebroespinal. El TTR ha sido propuesto como un potencial gen candidato asociado a la respuesta fisiológica de cerdos sometidos a estrés por calor, el cual limita seriamente la capacidad reproductiva(27), ya que disminuye la calidad de los ovocitos y la viabilidad embrionaria, intensificando los efectos negativos del PRRS sobre la fertilidad de cerdas infectadas. Por otra parte, el SNP rs80893794 (asociado a LNV2) se localiza en una región intrónica del gen CWH43 (Cell wall biogenesis 43 C-terminal homolog). El gen CWH43 está involucrado en la remodelación de lípidos de la pared celular de las levaduras(28) y se ha reportado que dicho gen es homólogo al PGAP2 (Post-GPI attachment to proteins), el cual está implicado en los ciclos de deacetilación y re-acetilación de las proteínas que sintetizan Phosphatidyl Inositol (PI) en las células mamíferas(29). El PI es una familia de lípidos que participan en el mecanismo de segundo mensajero en la membrana celular, y que es empleado por diversas hormonas, entre ellas la PGF2α, la cual juega un papel importante en la función ovárica y actividad uterina, y por lo tanto influye directamente en las variables de LNV1 y LNV2.
Los restantes seis SNP se localizan en regiones intergénicas (posicionales). Al respecto, es importante puntualizar que, al explorar el genoma completo, resulta complicado detectar una variante causal o directamente responsable de los cambios fenotípicos en las poblaciones; sin embargo, debido a la propiedad del desequilibrio de ligamiento en el genoma, es posible detectar asociaciones indirectas entre diversos SNP y los fenotipos de interés. Lo anterior sustenta la importancia de tomar en cuenta genes con ubicación cromosómica cercana a aquellos SNP que resulten significativos y que tienen posiciones intergénicas (cuando menos en un rango de 100 mil pares de bases; 100 kbp)(30). Uno de estos SNP es el rs81334603 asociado a LNV1, mismo que se ubica a una distancia de 40.26 kbp del gen KCNQ4 (Potassium voltage-gated channel subfamily Q member 4). La expresión de este gen se encuentra disminuida en ganglios linfáticos traqueobronquiales de cerdos infectados con la cepa norteamericana VR-2332 del virus del PRRS(31). Además, aproximadamente a 72.77 kbp del SNP rs81334603 se localiza el gen CTPS1 (CTP Synthase 1), el cual codifica para la producción de la enzima CTP sintetasa encargada de la biosíntesis de nucleótidos de pirimidinas (UTP y CTP), así como también para la síntesis de ciclopentenilcitosina, un agente antiviral de amplio espectro(32) .
En lo que respecta al SNP rs80947173, también asociado a LNV1, el gen más cercano a su ubicación es FLRT2 (fibronectin leucine rich transmembrane protein 2) localizado a 889.05 kbp de distancia. Si bien es cierto que este gen se encuentra considerablemente alejado del SNP antes mencionado, niveles útiles de desequilibrio de ligamiento (>0.3) parecen extenderse a distancias mayores en cerdos que en ganado Holstein, lo que implica que paneles de SNP menos densos aún pueden brindar resultados confiables en estudios asociativos de genoma completo(33). Por su parte, se ha reportado que el gen FLRT2 se encuentra asociado al número de lechones nacidos vivos en poblaciones de cerdos Large White y Landrance(34) . En el caso de los SNP asociados a LNV2, uno de ellos es el rs81364943, ubicado a 88.72 kbp de distancia del gen ISOC1 (isochorismatase domain containing 1) y a 178.67 kbp del gen ADAMTS19 (ADAM metallopeptidase with thrombospondin type 1 motif 19). El gen ISOC1 ha sido vinculado a procesos de actividad catalítica en ovocitos porcinos mediante un estudio de co-expresión genética(35), mientras que polimorfismos del gen ADAMTS19 en mujeres, se han relacionado con la presencia del síndrome de ovarios poliquísticos(36) .
Otro de los SNP asociados a LNV2 fue rs80859829, el cual se posiciona a 31.75 y 177.73 kbp de los genes ADAMTS20 (ADAM metallopeptidase with thrombospondin type 1 motif 20) e IRAK4 (interleukin 1 receptor associated kinase 4), respectivamente. La posible explicación de la asociación de este polimorfismo con el desempeño reproductivo al parto de cerdas reproductoras infectadas con PRRS, se atribuye a que el gen ADAMTS20 se expresa en alto grado en órganos como el cerebro y las gónadas(37), mismos que resultan afectados tras la infección por el virus del PRRS(38). Del mismo modo, la quinasa asociada al receptor de interleucina-1 (producto proteico del gen IRAK4), se ha visto involucrada en los mecanismos de replicación viral del PRRS, puesto que su producción se limita o disminuye a causa de la acción de un conocido micro RNA (miRNA-373) con efectos provirales(39) .
El SNP rs80895640 también asociado a LNV2, se encuentra a 34.3 kbp del gen CD83 (CD83 molecule); este gen, ha sido previamente vinculado con el número de lechones nacidos totales y el número de lechones nacidos vivos en cerdos híbridos Ibérico X Meishan(40,41). De manera interesante, dentro del contexto inmunológico, la molécula CD83 ha sido recientemente señalada como una pieza clave del mecanismo de escape ante el sistema inmune que utiliza el virus del PRRS, ya que éste es capaz de regular positivamente la expresión en forma soluble de la molécula CD83 (sCD83), la cual se encuentra asociada a la inmunosupresión de la proliferación de células T en el hospedador(42). Similarmente, a 54.05 kbp del SNP rs80895640, se ubica el gen RNF182 (Ring finger protein 182), mismo que está involucrado en procesos de apoptosis neuronal(43) que ocurren comúnmente tras la infección de cepas altamente infecciosas del virus del PRRS(44) .
Finalmente, el SNP rs81245908 (asociado a LNV2) se encuentra a una distancia aproximada de 53.52 kbp del gen FAT4 (FAT atypical cadherin 4). La asociación de este polimorfismo podría explicarse a partir de un estudio que brinda evidencia de que la expresión del gen FAT4 en humanos ha sido identificada en el tejido cerebral fetal y en el cerebro de infantes(45), mientras que por otra parte, se ha comprobado que la infección materna por PRRS puede afectar el desarrollo neuronal en los lechones, reduciendo el número de neuronas del hipocampo y aumentando el número de glías(46) .
Conclusiones e implicaciones
La detección de regiones genómicas que explican el 3.6 y 4.1% de la variación asociada a las variables de lechones nacidos vivos al primer y segundo parto, así como la validación de ocho SNP localizados dentro de dichas regiones, sugieren la existencia de una base genética que sustenta la respuesta reproductiva de cerdas reproductoras infectadas con el virus del PRRS. Por lo tanto, se propone que los ocho SNP asociados con las variables de LNV1 y LNV2, dos funcionales y seis posicionales, sean considerados como marcadores genéticos en programas de selección enfocados a mejorar la eficiencia reproductiva de cerdas infectadas con el virus del PRRS. Se sugiere realizar estudios adicionales para evaluar la funcionalidad de los SNP detectados; asimismo, se considera importante validar las regiones genómicas y los genes reportados en el presente estudio, en poblaciones de cerdas infectadas con el virus del PRRS pero de diferente raza.












.jpg&w=3840&q=75)