Parvovirus porcino: un antiguo pero todavia importante patógeno
Publicado: 9 de julio de 2021
Por: Andrés Felipe Streck; Diezza Biondo; y Jessica Gomes Maciel. Diagnostic Laboratory in Veterinary Medicine. University of Caxias do Sul. Brazil
AGENTE ETIOLÓGICO, EPIDEMIOLOGÍA Y PATOGÉNESIS
El Parvovirus Porcino (PVP o parvovirus Ungulado 1) es un virus de ADN, reconocido por primera vez a finales de la década de 1960, como miembro de la familia Parvoviridae (Streck; Truyen, 2020). Los parvovirus pertenecen a la familia Parvovirídae, que consta de dos subfamilias, Parvovirinae y Densovirinae. Son virus pequeños (parvus significa pequeño en latín) que tienen un genoma viral monocatenario de ADN de 5 kb, con cuatro genes que codifican dos proteínas estructurales y dos proteínas de replicación, generalmente actuando por empalme alternativo (alternative splicing).
Los Parvovirus tienen una estructura compleja de pinza palindrómica de aproximadamente 120-200 bases, en ambas secuencias terminales del genoma, que son un requisito para la replicación del ADN. La replicación viral es altamente dependiente de las fases celulares S o G1, las cuales corresponden al momento de la división celular, afectando por tanto órganos que presentan proliferación celular, como la médula ósea, las células embrionarias y los precursores intestinales (Zádori; Szelei; Tijssen, 2005).

Los animales susceptibles pueden infectarse por vía oronasal o venérea, lo que puede resultar en infección transplacentaria, muerte del embrión y daño a los tejidos reproductivos maternos (Van Leengoed et al., 1983). Se distribuye mundialmente en la población porcina y se puede encontrar en todas las categorías de cerdos. La simplicidad estructural del PVP permite una elevada resistencia al calor, las modificaciones del pH y los compuestos químicos (Paul et al., 2003). Por lo general, el virus puede permanecer activo durante meses en instrumentos, establos, ropa, botas y equipos, que también pueden servir como transporte viral de una granja a otra.
El mecanismo de entrada de PVP en la célula no está claro, pero puede ocurrir por endocitosis o micropinocitosis mediada por clatrina a través de la vía endosomática, que permite que el virus alcance el núcleo y se replique mediante el mecanismo celular, lo que causa daño oxidativo e induce apoptosis. Inicialmente, se replica en el tejido linfoide y se disemina sistémicamente a través de la viremia. Cuando ocurre una infección fetal, el virus tiene un ambiente susceptible de infección y replicación debido a las altas actividades mitóticas presentes en los tejidos fetales (Boisvert; Fernandes; Tijssen, 2010; Paul; Mengeling, 1980; zhao, x. 2016).
Todavía no se sabe cómo el PVP atraviesa la barrera placentaria, ya que está formada por capas, que ni siquiera permiten el paso de anticuerpos. Sin embargo, la mejor teoría es que el virus usa macrófagos para atravesar la placenta epiteliocorial e infectar al feto, debido a su potencial de permanecer infeccioso después de ser fagocitado (Paul; Mengeling, 1980). Las propiedades de virulencia del PVP parecen estar relacionadas con el gen de la proteína viral, como se pudo observar in vitro, utilizando virus recombinantes derivados de PVP patógenos y no patógenos, se observó que el cambio de un solo aminoácido en la proteína de la cápside afectaba la capacidad de replicación en algunas líneas celulares y afectaba drásticamente a los efectos patogénicos en los fetos (Boisvert; Fernandes; Tijssen, 2010; Streck; Truyen, 2020).
Figura 1: Representación 3D de la cápside del parvovirus. Disponible en DOI: 10.2210/pdb1K3V/pdf.
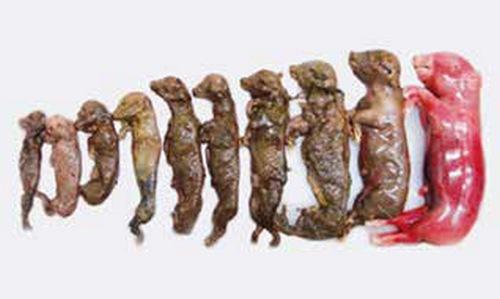
Figura 2: Imagen de fetos momificados diagnosticados con PVP. Fuente: HIPRA.
En la mayoría de los casos, la infección por PVP no presenta signos clínicos en cerdas no gestantes. La manifestación clínica más común son los fallos reproductivos en cerdas, especialmente nulíparas o primíparas (Truyen y Streck, 2019). El fallo reproductivo inducida por PVP suele conllevar la aparición de un número inusualmente grande de fetos momificados. La infección en las primeras etapas de la gestación provoca la muerte embrionaria y la reabsorción con camadas más pequeñas (Mengelin et al., 2000).
Los signos clínicos reproductivos están estrechamente relacionados con el momento de la gestación. Al comienzo temprano de la gestación, el embrión está protegido por la zona pelúcida y es resistente a la infección. Durante la etapa de embrión, la infección provoca su muerte y reabsorción. Después del día 35 de gestación, la infección del feto puede provocar la muerte y la posterior momificación. A partir de entonces, aproximadamente el día 70 de gestación, el feto puede desarrollar una respuesta inmune eficaz y eliminar el virus (Mengeling et al. 2000).
Las lesiones son específicas y se limitan al útero y al feto. Macroscópicamente, se puede observar en el feto una pérdida de condición corporal, congestión, edema, hemorragia, acumulación de líquido sanguinolento en las cavidades corporales y deshidratación de los tejidos que conduce a la momificación fetal (Mengeling, 1999). Microscópicamente, la necrosis celular en varios tejidos y la infiltración de células mononucleares son las principales observaciones (Joo; Johnson, 1977; Hogg et al., 1977). En las cerdas gestantes, las lesiones microscópicas se definen por inflamación, hipertrofia endotelial e infiltración de células mononucleares en endometrio, mucosas y miometrio (Hogg et al., 1977).
El diagnóstico se realiza mediante diversas técnicas de laboratorio, con el fin de detectar anticuerpos o virus. Entre ellas se encuentran las técnicas serológicas, útiles para verificar el estado inmunológico y la condición hereditaria del animal cuando se evalúan juntas, lo que permite la observación de los picos de circulación viral en los animales (Mengeling et al., 1999). La técnica estándar utilizada para la detección de anticuerpos contra el PVP es el ensayo de inhibición de la hemaglutinación (IH), que tiene un bajo coste y la posibilidad de titulación de anticuerpos. Enzyme Like Immuno Sorbent Assay (ELISA) también tiene ventajas, como la lectura automatizada, la alta reproducibilidad y el uso de kits, siendo considerado tan efectivo como el IH (Truyen y Streck, 2019).
El aislamiento viral en cultivo de células porcinas también se puede utilizar en el diagnóstico, debido a la replicación viral de alta eficiencia en células renales o testiculares porcinas, principalmente en PK-15 (riñón porcino), SK-6 (riñón porcino), STE (testículo porcino), ESK (riñón embrionario de cerdo) y SPEV (riñón embrionario de cerdo) (Truyen y Streck, 2019). Generalmente, la replicación de este virus da como resultado efectos citopáticos como granulaciones, forma irregular, replicación lenta, inclusiones intranucleares, núcleo picnótico y, en vista de eso, muerte celular (Cartwright et al., 1969; Mengeling, 1972). Debido a posibles efectos citopáticos o enzimáticos similares a otros virus, el aislamiento y la titulación del virus a menudo se asocian con la reacción en cadena de la polimerasa (PCR) (Truyen y Streck, 2019).
Con el reciente avance de la biología molecular, la identificación de PVP se puede realizar mediante técnicas basadas en la detección de secuencias virales específicas. La PCR es la técnica más utilizada, por su especificidad y sensibilidad, pudiendo detectar el virus en tejidos fetales, semen y otras muestras (Truyen y Streck, 2019). La PCR en tiempo real se ha considerado una herramienta de diagnóstico eficaz para el PVP, que vincula la sensibilidad y la especificidad de la PCR convencional con el beneficio de permitir la cuantificación y los resultados de la lectura automatizados (Mckillen et al., 2007). Recientemente, la técnica de amplificación isotérmica mediada por bucle (LAMP) se ha descrito como una opción ventajosa para el diagnóstico molecular porque es un método de extrema especificidad, sensibilidad y velocidad, además de ser aún más accesible en comparación con otras técnicas (Zhao, K. et al.,2020).
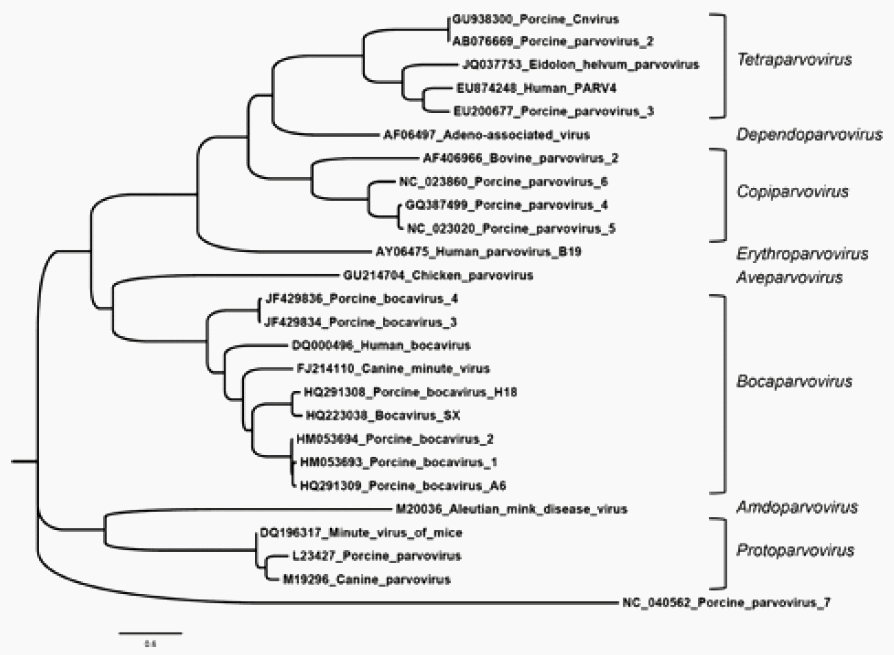
En las dos últimas décadas se describieron seis nuevos tipos de Parvovirus Porcino (Figura 3).

Figura 3: Equipamiento de laboratorio de diagnóstico. Fuente: HIPRA Diagnos.
El Comité Internacional de Taxonomía de Virus (ICTV) clasificó en una nueva propuesta los nuevos virus como el Parvovirus Porcino 2 (PVP2) y el Parvovirus Porcino 3 (PVP3), especies de tetraparvovirus ungulado 3 y Ungulate tetraparvovirus 2, respectivamente, en el género Tetraparvovirus y el Parvovirus Porcino 4 (PVP4), especie Ungular el copiparvovirus 2 en el género Copiparvovirus. El Parvovirus Porcino 5 (PVP5), aún no está categorizado en la propuesta de taxonomía de ICTV, pero el análisis genético mostró que este virus está altamente relacionado con PVP4. El ICTV informa el parvovirus 6 porcino (PVP6) como un virus asociado en el género Copiparvovirus (Cotmore et al., 2019; MIŁEK et al., 2019). El Parvovirus Porcino 7 (PVP7) es el más recientemente descubierto, en el que se propuso el nuevo género Chappaparvovirus (Palinski; Mitra; Hause, 2016). Varios estudios han demostrado una distribución geográfica muy amplia de nuevos Parvovirus Porcinos en todo el mundo. Todas las especies de PVP se identificaron en China, EE.UU. y Polonia. Se detectaron informes de PVP2, PVP3 y PVP4 en Hungría, Rumania, Tailandia, Japón y Sudáfrica. El PVP3, PVP4 y PVP7 se verificaron en Reino Unido, Brasil y Corea, respectivamente (Miłek et al., 2019).
Estos nuevos virus se detectaron en cerdos con diferentes manifestaciones clínicas, así como en animales sanos. Se encontraron en varios tipos de muestras de fluidos corporales, órganos como hígado, pulmones, corazón, bazo, riñón, ganglios linfáticos, amígdalas y fetos abortados. La relevancia de los nuevos Parvovirus Porcinos con respecto a la salud animal aún no se comprende bien. Es difícil afirmar la importancia real de los nuevos parvovirus para la salud de los cerdos porque no se han realizado estudios de aislamiento en cultivos celulares e infecciones experimentales (Miłek et al., 2019).
| Figura 4. La historia evolutiva se infirió utilizando el método de máxima verosimilitud y el modelo LG. El árbol inicial para la búsqueda heurística se obtuvo automáticamente aplicando los algoritmos Neighbour-Join y BioNJ a una matriz de distancias por pares estimadas utilizando el modelo JTT, y luego seleccionando la topología con un valor de probabilidad logarítmico superior. Se utilizó una distribución gamma discreta para modelar las diferencias de tasa de evolución entre los sitios (6 categorías). El árbol está dibujado a escala, con las longitudes de las ramas medidas en el número de sustituciones por sitio. Los análisis evolutivos se realizaron en MEGA X (Kumar et al., 2018). |
También se estudió la coinfección de PVP con Circovirus Porcino Tipo 2 (PCV2) y suscitó preocupación. Los informes muestran que los cerdos infectados con PVP y PCV2 tienen una disminución significativa en el peso corporal en comparación con los sanos. Además, en los ganglios linfáticos de los cerdos infectados por ambos virus se observan reacciones inflamatorias granulomatosas y depleción linfoide, lesiones típicas en cerdos con Síndrome de desmedro multisistémico posdestete (PMWS), hoy conocido como enfermedad sistémica por PCV-2. Por lo general, las camadas coinfectadas tienen lesiones patológicas más graves, lo que indica una acción sinérgica entre los dos virus, lo que indica que la infección por PVP proporciona un mejor entorno para la infección por PCV2 (Kennedy et al., 2000; OUYANG et al., 2019; Sharma; Saikumar , 2010). La coinfección por PCV2 y PVP está relacionada con la falta de activación temprana de IFN-γ y TNF-α (factores del sistema inmunológico), una respuesta inmunitaria humoral baja y retardada y viremia persistente (Opriessnig et al., 2017).
No existe un tratamiento específico para el Parvovirus Porcino, por lo que se deben implementar medidas de higiene y manejo para mantener el status sanitario de la granja. Para estandarizar la inmunidad de la granja, se pueden utilizar dos medidas: infección intencionada o vacunación. La infección intencional, muy utilizada en el pasado, se basa en el contacto directo de las nulíparas con las cerdas de la granja o material supuestamente infectado, como heces, restos placentarios y fetos momificados, generalmente un mes antes de la cubrición. No se recomienda esta estrategia, debido al riesgo de propagar otras enfermedades en los animales. Además, el material expuesto puede no contener suficiente virus para causar infección y no inducir la seroconversión de todos los animales (Abreu et al., 2018; Truyen y Streck, 2019).
La vacunación es el método más utilizado para controlar el PVP, con el principal objetivo de estimular la inmunidad del animal, especialmente en las cerdas nulíparas, evitando la posterior infección intrauterina. Las vacunas disponibles están elaboradas con virus inactivados, considerándose seguras y eficaces frente al PVP. Estas vacunas tienen un amplio margen de seguridad y confieren protección contra PVP incluso si se administran junto con otros agentes en vacunas polivalentes. El uso de estas vacunas induce buenos títulos de anticuerpos que pueden reducir las manifestaciones clínicas, aunque todavía no son capaces de prevenir la infección o diseminación (ZEEUW et al., 2007). Las vacunas de virus vivo modificado (MLV) pueden ser una alternativa al PVP. Se están desarrollando otras vacunas recombinantes pero la inactivada sigue siendo la más utilizada, debido a su amplio margen de seguridad (Truyen y Streck, 2019).
Referencias
- ABREU, Muriel Becker et al. O que há de novo sobre a parvovirose em suínos. In: BORTOLOZZO, Fernando Pandolfo et al. Avanços em sanidade, produção e reprodução de suínos III. Porto Alegre: UFRGS, 2018. Cap. 10. p. 69-76.
- BOISVERT, Maude; FERNANDES, Sandra; TIJSSEN, Peter. Multiple pathways involved in porcine parvovirus cellular entry and trafficking toward the nucleus. Journal of virology, [s. l.], v. 84, n. 15, p. 7782–7792, 2010.
- CARTWRIGHT, S F; LUCAS, M; HUCK, R A. A small haemagglutinating porcine DNA virus. I. Isolation and properties. Journal of comparative pathology, England, v. 79, n. 3, p. 371–377, 1969.
- COTMORE, Susan F et al. ICTV Virus Taxonomy Profile: Parvoviridae. The Journal of general virology, [s. l.], v. 100, n. 3, p. 367–368, 2019.
- HOGG, G.G.; LENGHAUS, C.; FORMAN, A.J. Experimental porcine parvovirus infection of foetal pigs resulting in abortion, histological lesions and antibody formation. Journal of comparative pathology, 87(4), 0–549. 1977.
- JOO, H. S.; JOHNSON, R. H. Serological Responses in Pigs Vaccinated With Inactivated Porcine Parvovirus. Australian Veterinary Journal, [s. l.], v. 53, n. 11, p. 550–552, 1977.
- KENNEDY, S et al. Reproduction of lesions of postweaning multisystemic wasting syndrome by infection of conventional pigs with porcine circovirus type 2 alone or in combination with porcine parvovirus. Journal of comparative pathology,
- England, v. 122, n. 1, p. 9–24, 2000.
- KUMAR S., STECHER G., LI M., KNYAZ C., AND TAMURA K. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms. Molecular Biology and Evolution, 35, p. 1547-1549, 2018.
- MCKILLEN, John et al. Molecular beacon real-time PCR detection of swine viruses. Journal of Virological Methods, [s. l.], v. 140, n. 1–2, p. 155–165, 2007.
- ABREUs: Properties and prevalence of a strain isolated in the United States. Am. J. Vet. Res. 33, 2239–2248.
- MENGELING, W.L. Porcine Parvovirus. In STRAW, B.E.; D’ALLAIRE, S.; MENGELING, W.L.; TAYLOR, D.J. (Org.). Diseases of Swine. 8.ed. Iowa State University Press: Ames, Cap.8, p. 119- 124, 1999.
- MENGELING, W.L.; LARGER, K.M.; VORWALD, A.C. The effect of porcine parvovirus and porcine reproductive and respiratory syndrome virus on porcine reproductive performance. Animal Reproductive Science, v. 60-61, p. 199-210, 2000.
- MIŁEK, Dagmara et al. Detection Patterns of Porcine Parvovirus (PPV) and Novel Porcine Parvoviruses 2 through 6 (PPV2-PPV6) in Polish Swine Farms. Viruses, [s. l.], v. 11, n. 5, 2019.
- OPRIESSNIG, Tanja et al. Markedly different immune responses and virus kinetics in littermates infected with porcine circovirus type 2 or porcine parvovirus type 1. Veterinary immunology and immunopathology, Netherlands, v. 191, p. 51–59, 2017.
- OUYANG, Ting et al. Co-Infection of Swine with Porcine Circovirus Type 2 and Other Swine Viruses. Viruses, [s. l.], v. 11, n. 2, 2019.
- PALINSKI, Rachel M; MITRA, Namita; HAUSE, Ben M. Discovery of a novel Parvovirinae virus, porcine parvovirus 7, by metagenomic sequencing of porcine rectal swabs. Virus Genes, [s. l.], v. 52, n. 4, p. 564–567, 2016.
- PAUL, P. S. et al. Exogenous porcine viruses. Current Topics in Microbiology and Immunology, [s. l.], v. 278, p. 125–183, 2003.
- PAUL, P S; MENGELING, W L. Evaluation of a modified live-virus vaccine for the prevention of porcine parvovirus-induced reproductive disease in swine. American journal of veterinary research, United States, v. 41, n. 12, p. 2007–2011, 1980.
- SHARMA, Rinku; SAIKUMAR, G. Porcine parvovirus- and porcine circovirus 2-associated reproductive failure and neonatal mortality in crossbred Indian pigs. Tropical animal health and production, United States, v. 42, n. 3, p. 515–522, 2010.
- Current issues in molecular biology, England, v. 37, p. 33–46, 2020.
- TRUYEN, Uwe; STRECK, André Felipe. Parvoviruses. In: ZIMMERMAN, Jeffrey J. et al. Diseases of Swine. 11. ed. Hoboken: John Wiley & Sons, Inc., 2019. Cap. 38. p. 611-621.
- VAN LEENGOED, L. A. et al. Porcine Parvovirus infection: review and diagnosis in a sow herd with reproductive failure. The Veterinary quarterly, [s. l.], v. 5, n. 3, p. 131–141, 1983.
- ZÁDORI, Zoltán; SZELEI, József; TIJSSEN, Peter. SAT: a late NS protein of porcine parvovirus. Journal of virology, [s. l.], v. 79, n. 20, p. 13129–13138, 2005.
- ZEEUW, E J L et al. Study of the virulence and cross-neutralization capability of recent porcine parvovirus field isolates and vaccine viruses in experimentally infected pregnant gilts. The Journal of general virology, England, v. 88, n. Pt 2, p. 420–427, 2007.
- ZHAO, Kai et al. Establishment of a porcine parvovirus (PPV)
- LAMP visual rapid detection method. Journal of virological methods, [s. l.], v. 284, p. 113924, 2020.
- ZHAO, Xiaomin et al. Porcine parvovirus infection activates mitochondria-mediated apoptotic signaling pathway by inducing ROS accumulation. Virology journal, [s. l.], v. 13, p. 26, 2016.
Temas relacionados:
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.




.jpg&w=3840&q=75)