Efecto de la grasa de la Dieta sobre la expresión de genes relacionados con el metabolismo Lipídico del cerdo
Publicado: 7 de julio de 2008
Por: Duran-Montgé, P.(IRTA) , Theil, P. K. (Department oh Animal Health, Welfare, and Nutrition, Danish Institute), Lauridsen, C. (Department oh Animal Health, Welfare, and Nutrition, Danish Institute), Esteve-Garcia, E. (IRTA)
La regulación del metabolismo de los ácidos grasos (AG) se da, entre otros, a través de cambios a nivel de trascripción, procesado del mRNA, estabilidad del mRNA o actividad de varios factores de transcripción. En este último caso, como la familia peroxisome proliferator activated receptor (PPAR) (Lee et al., 1995) y la sterol regulatory element binding proteins (SREBP1) (Kim y Spiegelman, 1996). Estos factores de transcripción regulan la expresión de diversos enzimas clave dentro las rutas metabólicas de los AG. En cerdos, el tejido adiposo es el principal órgano de síntesis de los AG (O’Hea y Leveille, 1969), pero hasta el momento sobre los efectos de los distintos AG de la dieta sobre la transcripción únicamente se han realizado estudios con cultivos celulares y cerdos destetados (Liu et al., 2005; Hsu et al., 2004). En especies donde el hígado es órgano primario de lipogénesis, los AG insaturados son más inhibitorios que los saturados, como es el caso de aves y roedores; en especies donde el tejido adiposo es el órgano principal, los AG saturados podrían ser tan potentes (o más) que los insaturados (Azain, 2004). Este estudio fue diseñado para estudiar el efecto de la composición de AG de la dieta sobre la transcripción de los genes involucrados en el metabolismo de la grasa en el hígado y tejido adiposo.
MATERIAL Y MÉTODOS
Animales y dietas. 61 cerdas (Duroc ♂x Landrace ♀) (62 ±5 kg pv) fueron asignadas a uno de los 7 tratamientos, con 8 animales por tratamiento, en 3 series de 3, 3 y 2 cerdas por serie. Las siete dietas fueron formuladas de acuerdo a los requerimientos nutricionales de la NRC (1998). Los tratamientos se asignaron de forma aleatoria por peso y camada. Las seis dietas con un 10% de grasa fueron a base de cebada y soja, y la dieta semisintética se formuló para contener un nivel muy bajo de grasa (Tabla1).
Las grasas utilizadas fueron: sebo, aceite de girasol alto-oleico, aceite de girasol, aceite de linaza, mezcla (55% sebo, 35% aceite girasol, 10% aceite de linaza) y una mezcla con aceite de pescado (40% aceite de pescado, 60% aceite de linaza). Para el estudio se tomaron muestras durante el sacrificio del hígado y grasa subcutánea del cuello que se congelaron inmediatamente en nitrógeno líquido y posteriormente a -75ºC.
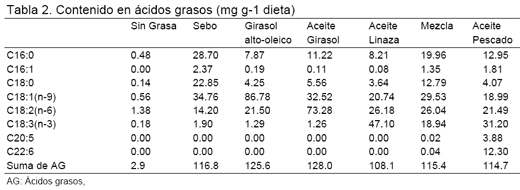
Análisis. Las dietas se analizaron para determinar el contenido en materia seca (AOAC, 1990), proteína cruda (Dumas), energía (AOAC, 1990) y AG. Los AG se extrajeron según Folch et al. (1957) y se transmetilaron con BF3 i KOH en metanol, posteriormente se cuantificaron por cromatografía de gases utilizando C19:0 como patrón interno (Tabla 2).
El mRNA se extrajo con TriReagent, y luego se adicionó BCP (Mol. Res. Cent., Cincinnati, Ohio 45212, USA) para separar el RNA y posteriormente se precipitó con isopropanol y purificó con etanol. La síntesis del cDNA se realizó mediante la transcriptasa inversa (Invitrogen, Taastrup, Denmark) siguiendo el protocolo del proveedor. El cDNA fue amplificado con TaqMan Universal PCR Master Mix (Applied Biosystems, Stockholm, Sweden) utilizando primers específicos para cada gen, siendo el marcaje del producto amplificado realizado mediante SYBR Green (SREBP-1 y PPAR-α) o sondas marcadas con carboxufluoresceína en el extremo 5’ (ACACA, FASN, SCD, D6D y HPRT1). La HPRT1 se utilizó como control endógeno (housekeeping gene). Para evaluar las cantidades de mRNA se calculó el número de ciclos (Ct) por el que la curva logarítmica cruzaba un valor umbral calculado, y se utilizó para determinar un ΔCt (ΔCt=Ct gen – Ct housekeeping gene). Los valores de ΔCt se analizaron por cada gen utilizando el procedimiento MIXED del SAS (tratamiento y series de animals, efectos fijos; camada x series efecto aleatorio).
RESULTADOS Y DISCUSSIÓN
Varios autores (Brown y Goldstein, 1997; Xu et al., 2001) sugieren que la SREBP1c regula la expresión de diferentes genes lipogénicos. Experimentos in vivo con ratas (Xu et al.,2002) y también en cerdos (Hsu et al., 2004; Lui et al., 2005) han demostrado que el aceite de pescado, rico en PUFAs de cadena larga, disminuye el contenido en mRNA de SREBP1. Al igual que estos experimentos, los animales alimentados con aceite de pescado mostraron los niveles más bajos de SREBP1 mRNA (Tabla 3). Los contenidos en ACACA y SCD mRNA (niveles bajos en la dieta que contiene aceite de pescado) muestran un comportamiento parecido al de la SREBP1, sugiriendo una regulación de estos SREBP1.
La regulación de los contenidos en mRNA de los genes relacionados con la síntesis de AG siguió una pauta diferente al hígado. A diferencia del hígado, los cerdos alimentados con la dieta sin grasa presentaron una expresión más alta de los genes involucrados en la síntesis de esteárico: acetil CoA carboxilasa y ácido graso sintasa (ACACA y FASN) y también de un gen involucrado en la desaturación (Stearoil CoA desaturasa) (SCD). Estos resultados se ajustan al incremento de la lipogénesis observado por Smith et al. (1996) en cerdos alimentados con una dieta sin grasa comparado con cerdos alimentados con 10% de grasa. A diferencia de lo observado en el hígado, el contenido en mRNA de la SREBP no mostró diferencias entre tratamientos, lo cual se corresponde con lo observado por Liu et al. (2005). Los cerdos alimentados con sebo, dieta rica en AG saturados, presentó los niveles más bajos en ACACA y FASN mRNA, mientras que los cerdos alimentados con aceite de girasol presentó los valores más altos. Otros autores (Smith et al., 1996; Allee et al., 1971) también han observado una reducción en la lipogénesis en cerdos alimentados con dietas enriquecidas con grasas saturadas comparando con dietas ricas en grasas insaturadas lo cual podría ser relacionado con una menor digestibilidad de las grasas saturadas; en nuestro caso esta posible interferencia se resolvió fijando la cantidad de grasa de las la dietas en función de su digestibilidad (Duran-Montgé et al., 2007). PPAR-α no mostró ningún cambio en los contenidos de mRNA por el tipo de dieta, tanto en tejido adiposo como en hígado.
Los resultados demuestran que la composición de la grasa de la dieta modifica la expresión de genes relacionados con la lipogénesis, tanto en hígado como en tejido adiposo. El tejido adiposo es el principal órgano de síntesis de AG en el cerdo y nuestros resultados sugieren que los efectos del tipo de grasa de la dieta en el hígado son distintos. Los AG saturados, comparados con otras fuentes de grasa, reducen la expresión de la FASN, ACACA y la SCD en el tejido adiposo.
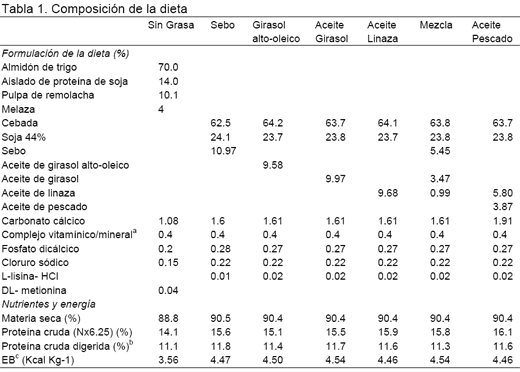
a Corrector vitamínico-mineral por kg de pienso: Vitamina A: 5000 UI; Vitamina D3: 1000 UI; Vitamina E: 15 mg; Vitamina B1: 1,3 mg; Vitamina B2: 3,5 mg; Vitamina B12: 0.025 mg; Vitamina B6: 1,5 mg; Pantotenato cálcico: 10 mg; Ácido nicotínico: 15 mg; Biotina: 0,1 mg; Ácido fólico: 0,6 mg; Vitamina K3: 2 mg; Fe: 80 mg; Cu: 6 mg; Co: 0,75 mg; Zn: 60 mg; Mn: 30 mg; I: 0,75 mg; Se: 0,10 mg; Etoxiquín: 150 mg.
bProteína bruta digerida fué obtenida de un experimento de digestibilidad fecal de proteína (Duran-Montgé et al. in press 2007)
c EB=Energía bruta
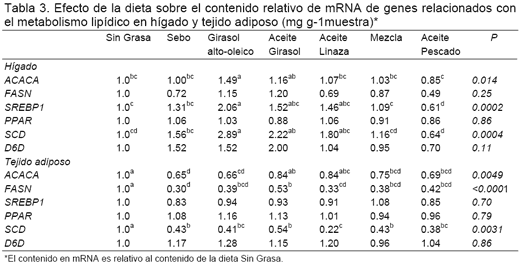
*El contenido en mRNA es relativo al contenido de la dieta Sin Grasa.
Acetil CoA carboxilasa α (ACACA), Ácido Graso sintasa (FASN), sterol regulatory element binding protein-1 (SREBP1), peroxisome proliferator activated receptor α (PPAR-α), estearoil CoA desaturasa (SCD), Δ6-desaturasa (D6D), HPRT1: Hipoxantina fosforibosiltransferasa
REFERENCIAS BIBLIOGRÀFICAS
MATERIAL Y MÉTODOS
Animales y dietas. 61 cerdas (Duroc ♂x Landrace ♀) (62 ±5 kg pv) fueron asignadas a uno de los 7 tratamientos, con 8 animales por tratamiento, en 3 series de 3, 3 y 2 cerdas por serie. Las siete dietas fueron formuladas de acuerdo a los requerimientos nutricionales de la NRC (1998). Los tratamientos se asignaron de forma aleatoria por peso y camada. Las seis dietas con un 10% de grasa fueron a base de cebada y soja, y la dieta semisintética se formuló para contener un nivel muy bajo de grasa (Tabla1).
Las grasas utilizadas fueron: sebo, aceite de girasol alto-oleico, aceite de girasol, aceite de linaza, mezcla (55% sebo, 35% aceite girasol, 10% aceite de linaza) y una mezcla con aceite de pescado (40% aceite de pescado, 60% aceite de linaza). Para el estudio se tomaron muestras durante el sacrificio del hígado y grasa subcutánea del cuello que se congelaron inmediatamente en nitrógeno líquido y posteriormente a -75ºC.
Análisis. Las dietas se analizaron para determinar el contenido en materia seca (AOAC, 1990), proteína cruda (Dumas), energía (AOAC, 1990) y AG. Los AG se extrajeron según Folch et al. (1957) y se transmetilaron con BF3 i KOH en metanol, posteriormente se cuantificaron por cromatografía de gases utilizando C19:0 como patrón interno (Tabla 2).
El mRNA se extrajo con TriReagent, y luego se adicionó BCP (Mol. Res. Cent., Cincinnati, Ohio 45212, USA) para separar el RNA y posteriormente se precipitó con isopropanol y purificó con etanol. La síntesis del cDNA se realizó mediante la transcriptasa inversa (Invitrogen, Taastrup, Denmark) siguiendo el protocolo del proveedor. El cDNA fue amplificado con TaqMan Universal PCR Master Mix (Applied Biosystems, Stockholm, Sweden) utilizando primers específicos para cada gen, siendo el marcaje del producto amplificado realizado mediante SYBR Green (SREBP-1 y PPAR-α) o sondas marcadas con carboxufluoresceína en el extremo 5’ (ACACA, FASN, SCD, D6D y HPRT1). La HPRT1 se utilizó como control endógeno (housekeeping gene). Para evaluar las cantidades de mRNA se calculó el número de ciclos (Ct) por el que la curva logarítmica cruzaba un valor umbral calculado, y se utilizó para determinar un ΔCt (ΔCt=Ct gen – Ct housekeeping gene). Los valores de ΔCt se analizaron por cada gen utilizando el procedimiento MIXED del SAS (tratamiento y series de animals, efectos fijos; camada x series efecto aleatorio).
RESULTADOS Y DISCUSSIÓN
Varios autores (Brown y Goldstein, 1997; Xu et al., 2001) sugieren que la SREBP1c regula la expresión de diferentes genes lipogénicos. Experimentos in vivo con ratas (Xu et al.,2002) y también en cerdos (Hsu et al., 2004; Lui et al., 2005) han demostrado que el aceite de pescado, rico en PUFAs de cadena larga, disminuye el contenido en mRNA de SREBP1. Al igual que estos experimentos, los animales alimentados con aceite de pescado mostraron los niveles más bajos de SREBP1 mRNA (Tabla 3). Los contenidos en ACACA y SCD mRNA (niveles bajos en la dieta que contiene aceite de pescado) muestran un comportamiento parecido al de la SREBP1, sugiriendo una regulación de estos SREBP1.
La regulación de los contenidos en mRNA de los genes relacionados con la síntesis de AG siguió una pauta diferente al hígado. A diferencia del hígado, los cerdos alimentados con la dieta sin grasa presentaron una expresión más alta de los genes involucrados en la síntesis de esteárico: acetil CoA carboxilasa y ácido graso sintasa (ACACA y FASN) y también de un gen involucrado en la desaturación (Stearoil CoA desaturasa) (SCD). Estos resultados se ajustan al incremento de la lipogénesis observado por Smith et al. (1996) en cerdos alimentados con una dieta sin grasa comparado con cerdos alimentados con 10% de grasa. A diferencia de lo observado en el hígado, el contenido en mRNA de la SREBP no mostró diferencias entre tratamientos, lo cual se corresponde con lo observado por Liu et al. (2005). Los cerdos alimentados con sebo, dieta rica en AG saturados, presentó los niveles más bajos en ACACA y FASN mRNA, mientras que los cerdos alimentados con aceite de girasol presentó los valores más altos. Otros autores (Smith et al., 1996; Allee et al., 1971) también han observado una reducción en la lipogénesis en cerdos alimentados con dietas enriquecidas con grasas saturadas comparando con dietas ricas en grasas insaturadas lo cual podría ser relacionado con una menor digestibilidad de las grasas saturadas; en nuestro caso esta posible interferencia se resolvió fijando la cantidad de grasa de las la dietas en función de su digestibilidad (Duran-Montgé et al., 2007). PPAR-α no mostró ningún cambio en los contenidos de mRNA por el tipo de dieta, tanto en tejido adiposo como en hígado.
Los resultados demuestran que la composición de la grasa de la dieta modifica la expresión de genes relacionados con la lipogénesis, tanto en hígado como en tejido adiposo. El tejido adiposo es el principal órgano de síntesis de AG en el cerdo y nuestros resultados sugieren que los efectos del tipo de grasa de la dieta en el hígado son distintos. Los AG saturados, comparados con otras fuentes de grasa, reducen la expresión de la FASN, ACACA y la SCD en el tejido adiposo.

a Corrector vitamínico-mineral por kg de pienso: Vitamina A: 5000 UI; Vitamina D3: 1000 UI; Vitamina E: 15 mg; Vitamina B1: 1,3 mg; Vitamina B2: 3,5 mg; Vitamina B12: 0.025 mg; Vitamina B6: 1,5 mg; Pantotenato cálcico: 10 mg; Ácido nicotínico: 15 mg; Biotina: 0,1 mg; Ácido fólico: 0,6 mg; Vitamina K3: 2 mg; Fe: 80 mg; Cu: 6 mg; Co: 0,75 mg; Zn: 60 mg; Mn: 30 mg; I: 0,75 mg; Se: 0,10 mg; Etoxiquín: 150 mg.
bProteína bruta digerida fué obtenida de un experimento de digestibilidad fecal de proteína (Duran-Montgé et al. in press 2007)
c EB=Energía bruta


*El contenido en mRNA es relativo al contenido de la dieta Sin Grasa.
Acetil CoA carboxilasa α (ACACA), Ácido Graso sintasa (FASN), sterol regulatory element binding protein-1 (SREBP1), peroxisome proliferator activated receptor α (PPAR-α), estearoil CoA desaturasa (SCD), Δ6-desaturasa (D6D), HPRT1: Hipoxantina fosforibosiltransferasa
REFERENCIAS BIBLIOGRÀFICAS
• Allee, G.L. et al. 1971Journal of Animal Science 33(6): p. 1248.
• Azain, M.J. 2004. Journal of Animal Science 82: p. 916-924.
• Brown, M.S. y J.L. Goldstein. 1997. Cell 89(3): p. 331-340.
• Duran-Montgé, P. et al. 2007.Livestock Science.
• Folch, J. et al. 1957. Journal of biological chemistry 226(1): p. 497-509.
• Hsu, J.M. et al. 2004. Journal of Animal Science 82(3): p. 683-689.
• Kim, J.B. y B.M. Spiegelman. 1996. Genes & Development10(9): p. 1096-1107.
• Lee, S.S.T. et al. 1995. Molecular and Cellular Biology 15(6): p. 3012-3022.
• Liu, B.H. et al. 2005. Journal of Animal Science 83: p. 1516-1525.
• Liu, B.H. et al. 2005. Asian-Australasian Journal of Animal Sciences 18(10): p. 1451-1456.
• O'Hea, E.K. y G.A. Leveille. 1969. Journal of Nutrition 99(3): p. 338.
• Smith, D.R. et al. 1996. Journal of Animal Science 74: p. 975-983.
• Xu, J. et al. 2001. Journal of Biological Chemistry 276(13): p. 9800-9807.
• Xu, J. et al. 2002. Journal of Nutrition 132(11): p. 3333-3339.
Temas relacionados
Autores:
Únete para poder comentar.
Una vez que te unas a Engormix, podrás participar en todos los contenidos y foros.
* Dato obligatorio
¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Crear una publicación




.jpg&w=3840&q=75)