Circulación de Rotavirus grupo A en cerdos de Argentina
Publicado: 20 de enero de 2015
Por: Celina Vega; Marina Bok; Javier Cappuccio; Marina Dibarbora; Andrés Wigdorovitz; Viviana Parreño (Lab. de Virus Gastroentéricos, Inst. de Virología, CICVyA, INTA); Fernanda Jabif y Nicolas Fermentini (Vetanco S.A.), Argentina
INTRODUCCIÓN
En todo el mundo, la diarrea neonatal es endémica y constituye un importante problema sanitario de las explotaciones porcinas. Las diarreas neonatales resultan en pérdidas económicas directas e indirectas que se deben principalmente a los altos costos de tratamiento de los animales afectados, el aumento de la susceptibilidad a otras infecciones (las infecciones respiratorias resultan especialmente relevantes), la pérdida de uniformidad en la ganancia de peso y la muerte de los animales. Los lechones que sobreviven son destetados con menor peso al deseado, lo cual significa mayores costos de alimentos de recepción al destete, de gran incidencia en el costo de producción. Las causas de las diarreas son multifactoriales y multietiológicas.
Existen múltiples agentes infecciosos involucrados en los cuadros de diarrea neonatal de los cerdos, que pueden encontrarse por separado aunque es aún más frecuente la ocurrencia de coinfecciones. Si bien la etiología de las diarreas es compleja, los virus podrían ser el factor clave ya sea por su rol de agente primario de generación de enteritis o como factor predisponente de infecciones secundarias por otros agentes infecciosos, como bacterias y parásitos (Zhang y col., 2013). Rotavirus es responsable de cuadros de diarrea en neonatos de múltiples especies animales. Este virus se ha detectado en todos los países donde existe producción porcina, independientemente del tipo de explotaciones. Es ubicuo en el ambiente de las granjas y se considera una infección hiperendémica.
En muchos casos, la patología de Rotavirus se manifiesta al no poder cumplir con las condiciones óptimas de ambiente, temperatura, desinfección o descanso de las instalaciones previas a ser ocupadas por hembras a parir.La transmisión es directa por vía fecal -oral e indirecta mediada por alimentos, agua, vehículos o vectores mecánicos. En general, sólo afecta a lechones (<40 días) y en el caso de haber diarrea, la misma es de 1 -2 días de duración como máximo (Dhama y col, 2009).
Los Rotavirus son virus desnudos y altamente resistentes a la inactivación física y química. Su genoma está compuesto por 11 segmentos de ARN de doble cadena que codifican 11 ó 12 proteínas, 6 estructurales (VP) y 5 ó 6 no estructurales (NSP). Se los clasifica en grupos, de los cuales el Grupo A (RVA) es el más importante epidemiológicamente. A su vez, este grupo se subclasifica en G y P genotipos según la variación genética de las dos proteínas de la cápside externa, VP7 (glicoproteína, G) y VP4 (proteasa sensible, P). En el caso particular del ganado porcino, los G tipos más frecuentemente encontrados son G3, G4, G5, G9 y G11 combinados con P[6], P[7], P[13] y P[19] para RVA en el mundo (Midgley y col., 2012).
En estudios previos se ha reportado la circulación de los genotipos G5P[7], G4P[6], G8P[6] y G6P[1] en cerdos con y sin sintomatología en Argentina (Parra y col., 2008). Dado el movimiento de animales y el fuerte crecimiento experimentado por el sector, es fundamental conocer la epidemiología de las diarreas neonatales en nuestro país, donde particularmente no se vacuna a los animales contra RVA.
OBJETIVOS
Caracterizar las cepas de RVA circulantes granjas porcinas de nuestro país durante el año 2013 y su relación con cepas encontradas en otras regiones.
OBJETIVOS
Caracterizar las cepas de RVA circulantes granjas porcinas de nuestro país durante el año 2013 y su relación con cepas encontradas en otras regiones.
RESULTADOS


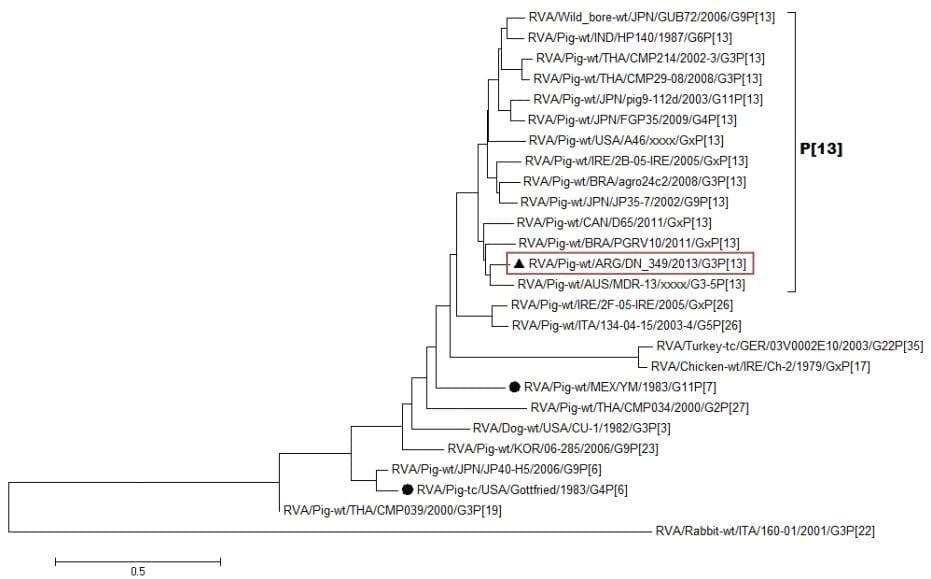
Análisis filogenético de la cepa P[13] encontrada
MATERIAL Y MÉTODOS
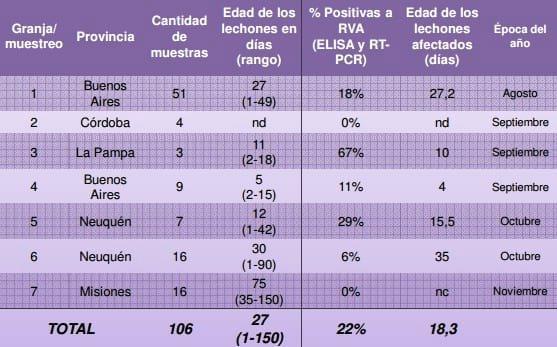
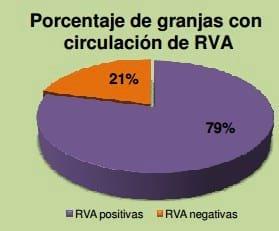
Se colectaron un total de 118 muestras de material fecal de lechones con diarrea y 20 no diarreicas. Las mismas procedían de 7 establecimientos y se tomaron entre agosto y diciembre de 2013. Todas las muestras fueron analizadas por ensayo de ELISA para la detección de RVA. Aquellas muestras positivas fueron analizadas por RT-PCR y se secuenciaron los genes codificantes de VP4 y VP7. RESULTADOS Se detectó la presencia de RVA por ensayo de ELISA en 10 muestras de material fecal (índice de detección de 7%) procedentes de lechones de entre 3 y 35 días de vida con y sin síntomas de diarrea. Sin embargo, RVA fue encontrado en el 57% de las granjas incluidas en este estudio.
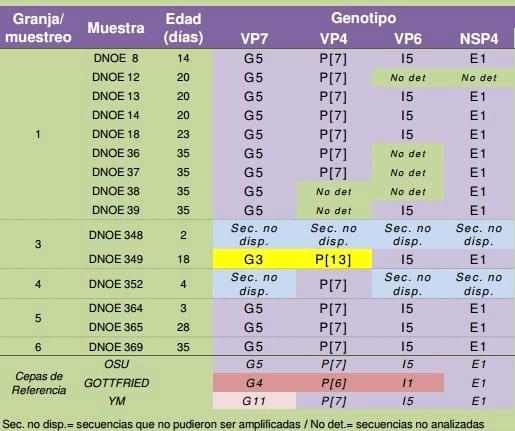
Las muestras positivas correspondían a granjas de las provincias de Buenos Aires, Córdoba, La Pampa and Neuquén. El análisis molecular de las mismas reveló la presencia del genotipo G5P[7] como el predominante. Una muestra proveniente de un lechón de 18 días fue genotipificada como G3P[13].
DISCUSIÓN
La combinación genotípica G3P[13] es rara y ha sido reportada en cerdos de Asia, Italia y más recientemente en Brasil (Okitsu y col, 2013). Este estudio constituye el primer reporte de este genotipo en cerdos de nuestro país. El análisis filogenético del P[13] encontrado revela que se encuentra fuertemente relacionado con cepas de Canadá, Brasil y Australia (distancias genéticas de 15.7%, 14% y 12.1%, respectivamente).
La combinación G5P[7], encontrada en mayor proporción en nuestro país, está incluida en las vacunas que se comercializan en otros países (México, Estados Unidos), y que no están disponibles actualmente en Argentina. Si bien se desconoce el impacto que RVA tiene en la producción porcina nacional, conocer la epidemiología de las cepas circulantes permitirá implementar rápidamente estrategias de control apropiadas en casos donde sea necesario.
CONCLUSION
El origen y diseminación de nuevos genotipos virales de RVA en cerdos estaría asociado al movimiento de animales dentro de la región. El 80% de las granjas relevadas presentan circulación de RVA en lechones de distintas edades. Si bien la combinación G5P[7] fue encontrada en mayor proporción en nuestro país, se debe profundizar el estudio filogenético de la misma y determinar su rol en las diarreas neonatales porcinas en Argentina. Este es el primer reporte de la circulación del nuevo genotipo de RVA porcino G3P13 en nuestro país. Dado que se desconoce el impacto que RVA tiene en la producción porcina nacional, conocer la epidemiología de las cepas circulantes permitirá implementar rápidamente estrategias de control apropiadas y racionalmente diseñadas.
BIBLIOGRAFÍA
- Dhama, K., R. S. Chauhan, et al. (2009). "Rotavirus diarrhea in bovines and other domestic animals." Vet Res Commun33(1): 1-23.
- Midgley, S. E., K. Banyai, et al. (2012). "Diversity and zoonotic potential of rotaviruses in swine and cattle across Europe." Vet Microbiol 156(3-4): 238-245.
- Okitsu, S., et al. (2013). “Whole-genomic analysis of G3P[23], G9P[23] and G3P[13] rotavirus strains isolated from piglets with diarrhea in Thailand, 2006–2008”. Inf, Gen and Evol. 18:74-86.
- Parra, G. I., G. Vidales, et al. (2008). "Phylogenetic analysis of porcine rotavirus in Argentina: increasing diversity of G4 strains and evidence of interspecies transmission." Vet Microbiol 126(1-3): 243-250.
- Zhang, Q., Hu, R., et al. (2013). “Occurrence and investigation of enteric viral infections in pigs with diarrhea in China”.Arch Virol158(8):1631-6.
Temas relacionados:
Autores:
Vetanco
GITEP
Instituto Nacional de Tecnología Agropecuaria - INTA
Instituto Nacional de Tecnología Agropecuaria - INTA
Instituto Nacional de Tecnología Agropecuaria - INTA
Instituto Nacional de Tecnología Agropecuaria - INTA
Mostrar más
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.