Análisis filogenético del virus de la diarrea epidémica porcina detectado en los Estados de Michoacán y Guanajuato
Publicado: 6 de noviembre de 2015
Por: Fernando Diosdado, Socci G, Amalia Martinez, Carrera E, Victor Quintero Ramirez, Gómez NL, Rivera BJF. CENID-Microbiología, INIFAP. México
Introducción
La diarrea epidémica porcina (DEP) es una enfermedad infecciosa de los cerdos, altamente contagiosa de origen viral. En los Estados Unidos de Norteamérica el virus de la diarrea epidémica porcina (VDEP) fue identificado a fines de abril del 2013, teniendo una identidad nucleotídica del 99.5 % con la cepa AH2012 que ocasionó problemas en China (1). En México, a principios de julio del 2013, productores y veterinarios de los estados de Michoacán y Guanajuato, dedicados a la clínica porcina reportaron cuadros de diarrea que concordaban con signos clínicos sugerentes de la infección por el VDEP. El reporte oficial se registró el 22 de mayo de 2014, sin embargo pese a los numerosos casos que se han reportado no se ha analizado la filogenia de los virus. Por lo que el objetivo de este estudio fue detectar al VDEP por medio de la técnica de RT-PCR, realizar la secuenciación parcial del gen S y analizar su filogenia para identificar el origen del VDEP que ocasionó los brotes es los estados ya mencionados.
Material y métodos
Se obtuvieron muestras de intestino delgado, intestino grueso y heces de dos lechones con cuadros agudos de diarrea sugerente al VDEP, provenientes de una granja de ciclo completo del estado de Michoacán y de tres lechones de dos granjas de Guanajuato. Para la detección del VDEP se empleó la técnica de RT-PCR utilizando un par de iniciadores que amplifican un fragmento de 503 pb del gene S del VDEP (2). Los productos de amplificación del VDEP obtenidos del RT-PCR, se purificaron con el kit (Wizard SV Gel and PCR Clean-Up System) y fueron secuenciados. El análisis de las secuencias se llevó a cabo con el programa Blast y Mega 5.
Resultados
El VDEP se logró detectar en tres de los cinco lechones a partir de las muestras de intestino delgado, grueso y heces de los lechones provenientes de los dos estados. El análisis de las secuencias de nucleótidos de los virus identificados mostró una homología del 99% con la cepa USA/Colorado/2013 de los Estados Unidos (1), además estas secuencias parciales fueron clasificadas en el genotipo 2a (virulento clásico) (Fig. 1). Las secuencias fueron reportadas en el GenBank con los siguientes números de acceso: KJ906601, KJ906602, KJ906603.
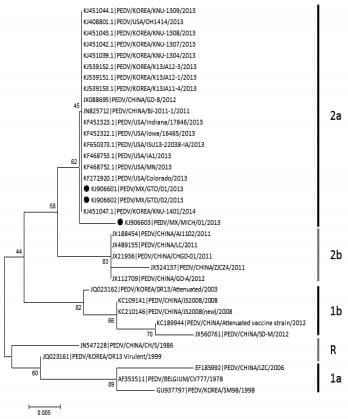
Figura 1. Análisis filogenético de las secuencias parciales (503 pb) del gen S del VDEP detectado en los estados de Michoacán y Guanajuato (Prueba de Neighbour-Joining, empleando 1000 réplicas de bootstrap).
Discusión y conclusiones
El VDEP se logró detectar por RT-PCR a partir de muestras de heces y de tejido intestinal. Los resultados de la secuenciación confirmaron la presencia del VDEP en las muestras, determinando que los virus más emparentados corresponden a cepas de Estados Unidos y cepas Coreanas.
Referencias
1. Marthaer et al., 2013. Genome Announc 1(4):1-2.
2. Ogawa et al., 2009. J Virol Methods 160:210-214.
Temas relacionados
Autores:
INIFAP México
INIFAP México
UNAM - Universidad Nacional Autónoma de México
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.







