INTRODUCCIÓN
El proceso de selección genética en ganadería, ha visto incrementada su eficiencia debido a métodos de predicción como el BLUP (mejor predicción lineal insesgada). El mismo es utilizado para estimar los valores de cría o el mérito genético de los animales; a través de la utilización combinada de información del individuo y de la familia. Al utilizar información familiar, los animales que están emparentados, usualmente tienen valores de cría similares, por lo que tienden a ser seleccionados o eliminados en grupo, aumentando el parentesco entre los individuos y la raza; disminuyendo la varianza genética y la respuesta a la selección (Quinton et al., 1992; Mackinnon, 2003). Adicionalmente a la utilización del BLUP, intensidades de selección elevadas por el uso de biotecnologías reproductivas como la inseminación artificial y la transferencia de embriones, también han conducido al incremento del parentesco dentro de algunas razas, a través de la reducción del número efectivo de padres (McDaniel, 2001; MacKinnon, 2003).
El apareamiento de animales emparentados en una población cerrada es inevitable, lo que conduce a la acumulación de consanguinidad y disminución de la diversidad genética (Falconer y Mackay, 1996); y ha demostrado ser causa de grandes pérdidas económicas cuando se superan ciertos niveles (Ercanbrack y Knight, 1991). Sin embargo, la consanguinidad ha sido considerada como una herramienta poderosa y útil para el mejoramiento de algunas razas ovinas, a través del incremento de la frecuencia de alelos deseables, la producción de líneas para su cruzamiento posterior y el aprovechamiento consecuente del vigor híbrido (Falconer y Mackay, 1996; Ceyhan et al., 2011). La pérdida de diversidad y el incremento resultante de la homocigocidad, pueden resultar en una disminución de la producción y/o la reproducción de animales consanguíneos. En ovinos, el efecto más importante se observa sobre características reproductivas como la fertilidad de los animales (Ercanbrack y Knight, 1991; MacKinnon, 2003). También puede dar lugar a una leve disminución en el peso del vellón, si bien parecería no tener efecto sobre la calidad de la lana (MacKinnon, 2003; Swanepoel et al., 2007). De esta manera, es importante tener en cuenta los efectos de la consanguinidad en poblaciones bajo selección, para ajustar adecuadamente el proceso de mejora por la posible disminución en el desempeño de los animales.
En el año 1999, el Instituto Nacional de Investigación Agropecuaria (INIA), junto al Secretariado Uruguayo de la Lana (SUL) y la Sociedad de Criadores Merino Australiano del Uruguay (SCMAU), basados en un diagnóstico de las tendencias mundiales de la producción y mercado de las lanas finas y superfinas, comenzaron un Proyecto de Investigación y Desarrollo del Merino Fino para Uruguay (PMF) (Montossi et al., 2005). El objetivo de dicho proyecto, fue el de desarrollar una alternativa de producción ovina que permitiera mejorar la sustentabilidad socioeconómica de los productores de lana de las regiones de Basalto y Cristalino. Éste tuvo como componente fundamental la formación del Núcleo Fundacional de Merino Fino de la Unidad Experimental “Glencoe” de INIA Tacuarembó (NFG). EL NFG fue formado inicialmente por 475 borregas (siendo posteriormente inferior el número efectivo de hembras utilizadas) provenientes de 37 productores colaboradores, las que fueron inseminadas con semen perteneciente a carneros importados y nacionales dando lugar, en 1999, a la primera progenie. Las estrategias empleadas para la creación de este Núcleo fueron detalladas por Montossi et al. (2005).
El objetivo de selección del NFG durante los 10 años de funcionamiento del PMF fue disminuir drásticamente el diámetro promedio de la fibra (DPF), manteniendo o incluso permitiendo, pequeñas pérdidas en Peso de Vellón Limpio (PVL). Con este fin se desarrolló un índice de selección (Ind) considerando ambas características (de los Campos et al., 2000). Asimismo, en los últimos años se incluyó, si bien no formalmente, al peso vivo a la esquila (PVE) como criterio de selección.
A nivel del NGF, existen varias causas que determinan un potencial alto riesgo de incrementar la consanguinidad de los animales del mismo, entre ellas podemos citar: (1) el reducido tamaño (un promedio de 285 animales por progenie con datos de diámetro, candidatos a selección), (2) la alta intensidad de selección aplicada en los padres y madres mediante un incremento de la utilización de Transferencia de Embriones (entre los años 2005-2009 los corderos nacidos por medio de esta tecnología osciló entre un 6 y 14%), y (3) la necesidad de manejar, por solicitud de la SCMAU, los apareamientos de Merino astado y mocho por separado, situación aún más preocupante en la población mocha ya que sólo representan en promedio (años 1999-2007) un 21.5% de los animales del NFG. Para la encarnerada del 2008, la SCMAU solicitó no mantener ambas líneas y que se considerase su cruzamiento en los apareamientos dirigidos.
El objetivo del presente trabajo es estudiar el impacto que ha tenido la implementación de un método sencillo para controlar el aumento de los niveles de consanguinidad en el NFG en relación al resto de la población Merino evaluada genéticamente en Uruguay.
MATERIAL Y MÉTODOS
Desde el año 2003 se comenzó a controlar la consanguinidad de los apareamientos dirigidos dentro del NFG. Una vez seleccionadas las madres de la futura progenie (por índice de selección), se les asigna al menos 3 posibles carneros (2 por inseminación artificial intrauterina y 1 como repaso por inseminación cervical con semen fresco), seleccionados también por el índice y otras características evaluadas genética y fenotípicamente. Para cada una de las opciones se genera un cordero “virtual” y se estima el coeficiente de consaguinidad (%F). Luego se descartan los apareamientos consanguíneos (%F>6), eligiendo otras opciones.
Se comparó la evolución (generaciones 1999-2009) del %F y de las tendencias genéticas para Ind, DPF, PVL, y PVE en el NFG y en la población Merino nacional total sin la inclusión del NFG (Población). La evaluación genética se realizó según Ciappesoni et al. (2006).
Las tendencias genéticas fueron calculadas con el procedimiento REG del paquete estadístico SAS (2003). Para el cálculo de la consanguinidad se compararon dos algoritmos: (1) según Wright (1922), el cual calcula como cero la consanguinidad de los animales que tienen al menos un padre desconocido (W); y (2) según VanRaden (1992) donde se le asigna a los animales con padres desconocidos la consanguinidad de sus contemporáneos con genealogía completa (VR). Los contemporáneos fueron, a su vez, diferenciados en dos grupos: animales pedigree (PI) y puros por cruza (PxC), por considerarse que la presión de selección en ambas poblaciones es diferente. Ambos cálculos se realizaron con el programa INBUPGF90 desarrollado por Aguilar y Misztal (2008), utilizando 10 iteraciones para el cálculo por el método de VR.
Para los cálculos de %F se utilizó la genealogía completa de la base de datos empleada en las evaluaciones genéticas, a la cual se le suma toda la información disponible de los carneros importados en la página Web australiana de evaluaciones genéticas (www.sheepgenetics.org.au).
RESULTADOS Y DISCUSIÓN
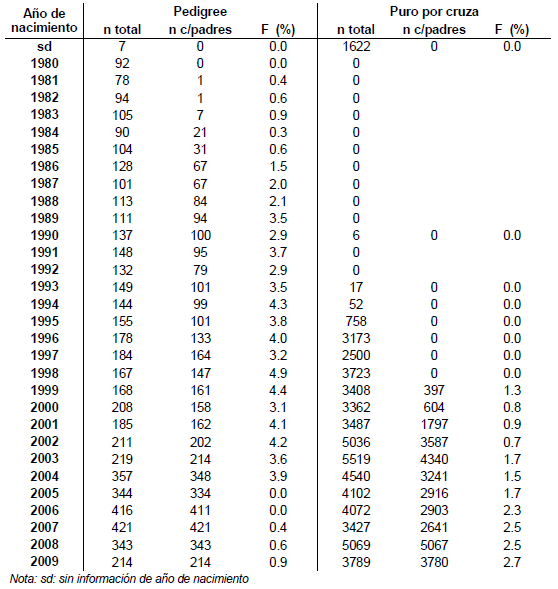
En el Cuadro 1 se presenta el número de animales en la genealogía total y aquellos con padre y madre conocidos, y el promedio de consanguinidad (%F) de estos últimos, según grupo genético (año de nacimiento y origen). Se observa un gran avance en los registros genealógicos en especial en los animales PxC, pasando de un 52% de animales con ambos padres conocidos en el año 2001, a prácticamente el 100% en las últimas dos generaciones (i.e. 2008 y 2009). Como era de esperar la información genealógica en los PI es muy completa, superando en los últimos años el 90% de los animales con padre y madre registrados. Los animales con registros productivos tanto PI como PxC, cuentan casi en su totalidad con información genealógica completa. Por lo tanto, la información faltante tanto de padre como de madre en ambos orígenes, se debe a que estos animales son ascendientes de animales con datos, en general ingresados desde fuera del sistema de evaluación genética nacional (e.g. animales importados, cabañas nuevas). Los promedios de %F fueron siempre superiores en el origen PI, siendo, por ejemplo, desde la generación 1999 entre 1.5 y 4.7 veces mayores que para el PxC. Por lo tanto, es conveniente tratar a ambos orígenes por separado para asignarles el %F promedio a los animales con genealogía faltante, según el método VR.
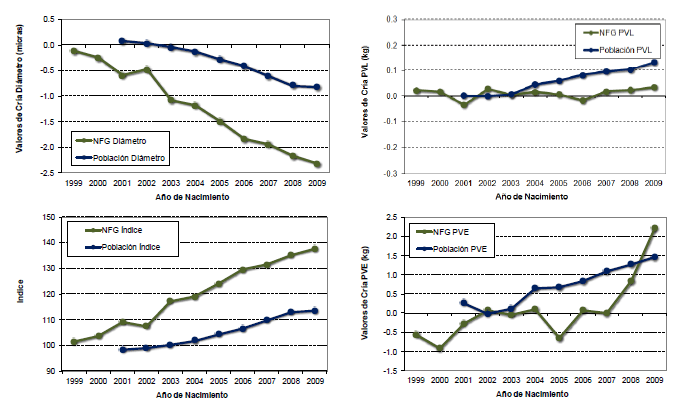
En la Figura 1 se presentan las tendencias genéticas para las características evaluadas. Para la Población, se grafica desde el año 2001 por ser este cuando comenzaron a conectarse las cabañas particulares a la evaluación poblacional. Se observa como se cumplieron los objetivos iniciales del NFG reduciendo el diámetro drásticamente y manteniendo el PVL. Asimismo, es notorio el énfasis que se le ha dado al PVE en los últimos dos años. Las tendencias en la Población y en el NFG siguen una misma orientación, pero se destaca el mayor progreso obtenido por este último en el índice de selección y consecuentemente en el diámetro de la fibra (Cuadro 2). Los incrementos en la consanguinidad anual promedio del NFG son similares a los obtenidos en la Población (Cuadro 2) y baja magnitud no superando el 0.1% anual para ambos algoritmos estudiados. Según Quinton et al. (1992) y McDaniel (2001), un incremento de hasta el 1%F por generación es aceptable en la mayoría de las poblaciones, si bien la magnitud de dicho incremento, usualmente es mayor en la mayoría de los estudios de simulación realizados.
Cuadro 1. Número de animales total (n total) y con padre y madre conocidos (n c/padres) y consanguinidad promedio (F) por grupo genético (año de nacimiento y origen).
Cuadro 2. Incremento anual de la consanguinidad (F) y de los valores genéticos promedio de diámetro de la fibra, peso de vellón limpio (PVL), peso vivo a la esquila (PVE) e índice de selección, para el NFG y la población total evaluada sin el NFG (Población), generaciones 1999 a 2009
Cuadro 3. Consanguinidad promedio (F), para el NFG, la población sin el NFG (Población) y el total de los animales evaluados (Total), generaciones 1999 a 2009.

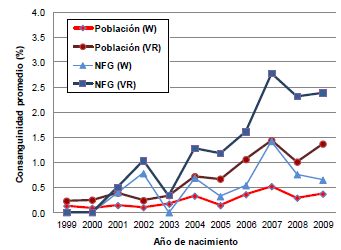
En el Cuadro 3 se observa que tanto para el NFG como para la Población, los valores promedios (generaciones 1999 a 2009) de consanguinidad son bajos. Los promedios aumentan considerablemente, al calcularse la consanguinidad según el método de alternativo (VR). Los animales consanguíneos según el método clásico (W) representan el 8 y 10% para la Población y el NFG, respectivamente. Estos porcentajes aumentan al 90 y 60%, respectivamente, al calcularse la consanguinidad según el método alternativo (VR). En la Figura 2, se presenta la evolución de la consanguinidad promedio a través de los años para el NFG y la Población según ambos métodos estudiados (W y VR). Si bien se observan en el NFG aumentos en %F, en especial desde la generación 2005, los valores se han mantenido por debajo de 1.2 y 2.8% para los métodos W y VR, respectivamente. En el caso de la Población, al existir constantes incorporaciones de animales desde fuera del sistema de evaluación genética, se aprecia como el método VR sería más apropiado para cuantificar el %F promedio.
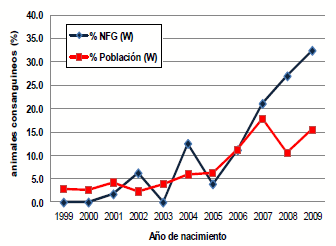
Asimismo, en la Figura 3 se observa el incremento del porcentaje de animales consanguíneos (según el método regular, W) desde la generación 1999 a la 2009. Se observa un incremento de los animales consanguíneos principalmente en los últimos cuatro años, posiblemente causado en parte porque en estas generaciones tanto en la Población como en el NFG, prácticamente no existen animales sin registros de padre y madre. A su vez, para el NFG y las cabañas que logran un marcado progreso genético en DPF, PVL y PVE, ha sido cada vez más dificultoso encontrar carneros importados que superen a los nacionales. Adicionalmente, varios de los carneros más afinadores (menor DEP para DPF), tanto nacionales como importados tienen ancestros en común.
Al incluir los grupos genéticos (por año de nacimiento y origen) con el método VR la mayoría de los animales pasan a ser consanguíneos. En el caso del NFG, en las 11 generaciones evaluadas el promedio fue de 92% de animales que presentan cierto grado de consanguinidad. La generación 2009 presentó el menor nivel de consanguíneos 74. En la Población, en los últimos dos años se supera el 90% de animales consanguíneos calculados según VR.
Si bien en la literatura existen resultados dispares de los efectos de %F sobre las características productivas y reproductivas, tanto en el cordero como en la madre (MacKinnon, 2003), los niveles alcanzados en la Población así como en el NFG no son de gran magnitud, y no estarían comprometiendo el desempeño productivo de los animales. De esta manera, es necesario seguir llevando a cabo un control efectivo de los apareamientos, ya sea por monta natural o inseminación artificial, para que el proceso de mejora genética continúe teniendo resultados exitosos.
Figura 1: Tendencias genéticas de diámetro de la fibra, peso de vellón limpio (PVL), peso vivo a la esquila (PVE) e índice de selección, para el NFG y la población total evaluada sin el NFG (Población), generaciones 1999 a 2009
Figura 2: Coeficiente de consanguinidad promedio (%F) para el NFG y la población total evaluada sin el NFG (Población), generaciones 1999 a 2009
Figura 3: Porcentaje de animales consanguíneos (%F); según algoritmo de Wright (1922) para el NFG y la población total evaluada sin el NFG (Población), generaciones 1999 a 2009.
CONCLUSIONES
Se destaca la importancia de incluir para el cálculo de la consanguinidad en poblaciones con genealogías incompletas, algoritmos como el estudiado (VR) por ser aproximaciones más cercanas a la realidad.
Se demuestra la efectividad del método empleado, que pese a las dificultades expresadas anteriormente, el %F promedio en el NFG se mantuvo siempre por debajo de valores tolerables que no estaría afectando el desempeño productivo de los animales, logrando a su vez importantes progresos genéticos en las características de mayor relevancia económica.
AGRADECIMIENTOS
Este trabajo no hubiese sido posible sin el apoyo de: Asociación Rural del Uruguay, SCMAU, SUL e integrantes, personal de apoyo y técnico de la Unidad Experimental Glencoe de INIA. A Ignacio Aguilar por sus valiosos aportes y facilitar el programa INBUPGF90.
REFERENCIAS BIBLIOGRÁFICAS
AGUILAR I., MISZTAL I. 2008. Technical Note: Recursive Algorithm for Inbreeding Coefficients Assuming Nonzero Inbreeding of Unknown Parents. J. Dairy Sci., 91: 1669-1672.
CEYHAN A., KAYGISIZ A., SEZENLER T. 2011. Effect of inbreeding on preweaning growth traits and survival rate in Sakiz sheep. J. Anim. Plant. Sci., 21: 1-4.
CIAPPESONI G., RAVAGNOLO O., GIMENO D., MONTOSSI F., DE BARBIERI I. 2006. Estimation of genetic parameters and genetic trends for wool production and quality for the uruguayan Merino. Proccedings of the 8th WCGALP, August 13-18, 2006, Bello Horizonte, Brazil. pp 1392-1395.
DE LOS CAMPOS G., DE MATTOS D., MONTOSSI F., SAN JULIÁN R., FRUGONI C. 2000. Incorporación de las señales de mercado a la toma de decisiones en mejora genética. Serie de Actividades de Difusión Nº 246, INIA Tacuarembó.
ERCANBRACK S.K., KNIGHT A.D. 1991. Effects of inbreeding on reproduction and wool production of Rambouillet, Targhee and Columbia ewes. J. Anim. Sci., 69: 4734-4744.
FALCONER D.S., MACKAY T.F.C. 1996. Introduction to Quantitative Genetics. Ed 4. Longmans Green, Harlow, Essex, UK.
MACKINNON K.M. 2003. Analysis of Inbreeding in a Closed Population of Crossbred Sheep. Master Thesis. Animal and Poultry Sciences - Virginia Polytechnic Institute and State University. 54p.
MCDANIEL B.T. 2001. Uncontrolled inbreeding. J. Dairy. Sci., 84:E185-E186.
MONTOSSI F., DE BARBIERI I, CIAPPESONI G., RAVAGNOLO O., DE MATTOS D., PÉREZ JONES J., FROS A., GRATTAROLA M., MEDEROS A., SOARES DE LIMA M. 2005. Núcleo fundacional de merino fino y superfino de la Unidad Experimental “Glencoe” - INIA Uruguay: una experiencia innovadora de mejoramiento genético asociativo y participativo. Agrociencia, Vol. IX-N°1 y 2, pp. 609-616.
QUINTON M., SMITH C., GODDARD M.E. 1992. Comparison of selection methods at the same level of inbreeding. J. Anim. Sci., 70: 1060-1067.
SAS. 2003. SAS version 9.1.3. SAS Inst., Cary, N.C., USA.
SWANEPOEL J.W., VAN WYK J.B., CLOETE S.W.P., DELPORT G.J. 2007. Inbreeding in the Donhe Merino breed in South Africa. Short Communications. South African J. Anim Sci., 37: 176-179.
VANRADEN P.M. 1992. Accounting for inbreeding and crossbreeding in genetic evaluations of large populations. J. Dairy Sci., 75: 3136–3144.
WRIGHT S. 1922. Coefficients of inbreeding and relationship. Amer. Nat., 56: 330–338.