Caracterización molecular de una población de mapeo asociativo de Lotus Tenuis: diversidad y estructura genética
Publicado: 21 de abril de 2017
Por: Luis Inostroza1 y Hernán Acuña2.
1Instituto de Investigaciones Agropecuarias, CRI-Quilamapu, Chillán, Chile
Introducción
Lotus tenuis es una especie leguminosa perenne, diploide (2n=12), de polinización cruzada y autofecundación incompatible. Por otro lado, Lotus japonicus es una leguminosa anual que ha sido ampliamente estudiada como ‘especie-modelo’ en estudios de la biología de la interacción planta/rizobio, dado su simplicidad genética (diploide, autofecundación y ciclo ontogénico corto). Las especies del género Lotus de importancia agrícola (forrajeras) son genéticamente cercanas a la especie modelo (Szczyglowski y Stougaard, 2008), por lo que gran parte de la información molecular puede ser transferida. El objetivo de este trabajo fue caracterizar la diversidad y estructura genética de una población de mapeo asociativo compuesta por 100 genotipos de L. tenuis, con marcadores moleculares tipo secuencias simples repetidas (SSR) identificadas en el genoma de L. japonicus.
Materiales y métodos
La población de mapeo asociativo se compone por 100 genotipos provenientes de dos poblaciones naturalizadas en Chile de Lotus tenuis, las que fueron previamente seleccionadas por su tolerancia a sequía divergente (Acuña et al., 2010). Las poblaciones sensible y tolerante a sequía se denominan Lt14 y Lt4, respectivamente. Cincuenta individuos de cada población conforman el germoplasma de estudio. La caracterización molecular se realizó en el laboratorio de Genómica Forestal de la Universidad de Concepción, Chile. La extracción del ADN genómico se realizó a partir de un brote apical con un kit comercial (DNeasy Plant Mini Kit, Qiagen). Se amplificaron 88 marcadores moleculares tipo secuencias simples repetidas (SSR), los cuales fueron previamente desarrollados en L. japonicus (www.kazusa.or.jp/lotus/). Cincuenta y cuatro partidores fueron seleccionados en estudios previos de transferibilidad entre especies del género Lotus, desarrollados por el Proyecto LOTASSA (Sanjuan y Rebuffo, 2010). El resto de los marcadores fueron seleccionados de tres trabajos en que se reportan QTLs asociados a caracteres de importancia agronómica en L. japonicus. La reacción en cadena de la polimerasa (PCR) se realizó con SSR marcados con etiquetas de fluorescencia bajo las siguientes condiciones: 1ng/μl de ADN, 10x PCR Buffer, 50mM MgCl2, 10mM dNTPs, 10ρmol/μl de cada SSR, 5U/μl de Taq DNA polimerasa y 1mg/ml BSA en un volumen total de 10μl. La PCR se programó con una temperatura inicial de desnaturalización de 95 ºC, seguido por 35 ciclos de 94 ºC por 30 s, la temperatura de alineamiento fue 60 ºC por 45 s, 72 ºC por 20 s, seguido por 35 ciclos de 94 ºC por 30 s, 50 ºC por 60 s, 72 ºC por 60 s y un paso final de extensión a 60 ºC por 30 min. Los productos PCR se genotiparon y cuantificaron usando un secuenciador automático (ABI 3130 xl) y el software GeneMapper® V4.0 (Applied Biosystem inc.), respectivamente. Se determinó el grado de información generado por cada SSR mediante la determinación de la frecuencia alélica, número de genotipos, número de alelos y el contenido de información polimórfica (PIC) usando el software PowerMarker V3.25 (http://statgen.ncsu.edu/powermarker/). La estructura de la población se caracterizó mediante un modelo basado en métodos de agrupamiento según frecuencia alélica implementado en el software Structure V2.3.4.
Resultados y discusión
El análisis de los electroferogramas permitió identificar 268 alelos. El número promedio de alelos por locus fue 3.6. Dieciséis locus mostraron el menor número de alelos (2) mientras que TM1150 mostró el mayor número de alelos (7). El valor de PIC se utiliza para evaluar el poder discriminatorio de un SSR y fluctúa entre 0 y 1. Un alto valor de PIC indica que el SSR es más informativo. El valor promedio de PIC de todos los loci evaluados fue 0.41. El valor máximo de PIC fue 0.68 (TM1150) mientras que el menor fue de 0.10 (TM0014).
La población de mapeo asociativo se encuentra altamente estructurada, información relevante para los análisis de mapeo asociativo. La estructura se compone de 5 grupos o clusters (Figura 1), entre los cuales, los clusters 1 y 2 diferencian la frecuencia alélica de las poblaciones fundadoras (Lt-4 y Lt-14). Sin embargo, algunos individuos comparten alelos de ambas poblaciones lo que origina el resto de los clusters. El cluster 1 agrupa el 82% de los individuos de la población Lt-4 y el Cluster 2 agrupa el 90% de los individuos de la población Lt-14.
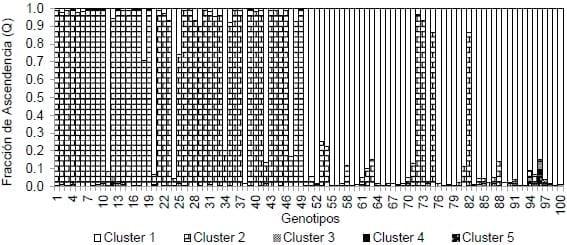
Figura 1. Ascendencia de cada genotipo (Q) que compone la población de mapeo asociativo estimada con Structure V2.3.4. Genotipos del 1 al 50 corresponden a población Lt-4 y del 51 al 100 a Lt-14.
Conclusiones
La población de L. tenuis mostró una alta diversidad genética, la que fue ampliamente descrita por los SSR desarrollos en L. japonicus. La población de mapeo asociativo se encuentra altamente estructurada, dado el fuerte agrupamiento ocasionado por las poblaciones fundadoras (Lt-4 y Lt-14).
Referencias
ACUÑA, H., L. INOSTROZA, M.A.P. SÁNCHEZ, and G. TAPIA. 2010. Drought-tolerant naturalized populations of Lotus tenuis for constrained environments. Acta Agriculturae Scandinavica, Section B - Plant Soil Science 60, 174-181.
SZCZYGLOWSKI, K., and J. STOUGAARD. 2008. Lotus genome: pod of gold for legume research. TRENDS in Plant Science 13, 515-517.
SANJUÁN J y REBUFFO M. 2010. LOTASSA: un puente entre la genómica y las pasturas del siglo XXI. PROCISUR, Montevideo, 70 p.
Actividad financiada por proyecto FONDECYT 11100094 y MINAGRI-INIA-501364-70.
Temas relacionados
Autores:
Únete para poder comentar.
Una vez que te unas a Engormix, podrás participar en todos los contenidos y foros.
* Dato obligatorio
¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Crear una publicación



.jpg&w=3840&q=75)