INTRODUCCIÓN
La garrapata Rhipicephalus (Boophilus) microplus es un ectoparásito del ganado bovino que se alimenta de sangre y tiene la capacidad vectorial de transmitir enfermedades como anaplasmosis y babesiosis (Miranda-Miranda et al., 2010). Este artrópodo representa un serio problema de salud animal en la ganadería bovina desarrollada en regiones tropicales y subtropicales del mundo, debido a su alta capacidad vectorial y el daño físico que recibe la piel del animal (Cossio-Bayugar et al., 2005). Actualmente, se sabe que las presiones ecológicas ejercidas sobre las garrapatas se basan en varios depredadores y organismos patógenos que afectan considerablemente sus poblaciones (Miranda-Miranda et al., 2011, 2012). En trabajos previos se han aislado bacterias a partir de garrapatas del género Rhipicephalus pertenecientes a los géneros, Staphylococcus, Pseudomonas, Acinetobacter, Enterobacter, Enterococcus (Sachman-Ruiz et al., 2014), Aeromonas, Bacillus, Brevibacillus, Castelaniella, Comamonas, Kocuria, Microbacterium, (Zimmer et al., 2013); Anaplasma, Ehrlichia y protozoarios parásitos como Babesia y Theileria (Tonetti et al., 2009). Algunas de estas especies bacterianas se han encontrado en huevos, larvas y hembras ingurgitadas (Amoo et al., 1987). Asimismo, estudios previos han demostrado la presencia de bacterias del género Staphylococcus en garrapatas hembras de R. microplus. Dichos individuos muestran signos de infección caracterizados por un exudado amarillento en el hipostoma y poro genital, así como un cambio de coloración en la cutícula, resultando en la muerte de hembras ingurgitadas por la interrupción de la oviposición (Miranda-Miranda et al., 2010).
Es el objetivo de este estudio, hacer una comparación de los aislados bacterianos obtenidos durante los años 2010 al 2015, presentes en exudados de garrapatas hembras de R. microplus infectadas naturalmente, para determinar si otros microorganismos además de bacterias del género Staphylocoocus estaban presentes en el exudado de las garrapatas con signos de infección que puedan ejercer un sinergismo en la patogenicidad la enfermedad descrita.
MATERIALES Y MÉTODO
Obtención de garrapatas. Se recolectaron las garrapatas obtenidas en tres diferentes infestaciones experimentales realizadas en tres diferentes años (2010, 2013 y 2015), en bovinos estabulados en el CENID-PAVET del INIFAP ubicados en Jiutepec, Morelos. Las garrapatas hembrasrepletas colectadas,se examinaron macro y microscópicamente en busca de características fenotípicas que mostraran signos de infección natural como son: exudado en el área del hipostomaporo genital, cambio de coloración corporal e incapacidad de ovipositar.
Aislamiento bacteriano y extracción de ADN. El aislamiento de bacterias se llevó a cabo mediante siembra con asa bacteriológica, a partir de exudados presentes en el orificio genital de las garrapatas hembras ingurgitadas, mismo exudado que se encuentra ausente en garrapatas sanas. El plaqueo de los exudados, se realizó en medios de cultivo Agar-Tripticasa de soya (TSA) (DifcoBD), siguiendo el protocolo descrito por el fabricante. Una vez sembradas, las cajas Petri fueron incubadas a 37 °C, para inducir el crecimiento de colonias. Estas colonias fueron mantenidas mediante pases consecutivos en medio TSA, hasta obtener Unidades Formadoras de Colonias (UFC). Una vez obtenidas, las UFC se sembraron en 5 ml de medio TSA líquido a 37 °C durante 16 h. De este cultivo, se tomaron alícuotas de 2 ml y se les añadió 0.3 ml. de glicerol, para la obtención de stocks que fueron almacenados a -70 °C. Asimismo, parte de las alícuotas obtenidas, fué usada para la extracción de DNA total con el QIAamp DNA Minikit (Qiagen-USA) de acuerdo a lo descrito por el fabricante.
Reacción en cadena de la polimerasa (PCR) y secuenciación.La amplificación del gen codificador de la secuencia 16S Ribosomal, se llevó a cabo mediante la PCR, a partir de 20 ng de ADN genómico, más 10μM de los oligonucleótidos universales fD1 y rD1 (Weisburg et al., 1991), 2.5U de Taq Polimerasa Platinum (Invitrogen) y 200 mM de dNTP´s. La reacción se realizó en un termociclador ARKTIK (ThermoScientiphic®), según las especificaciones del fabricante y las características de los primers, con 1 ciclo 94 °C 5 min; 30 ciclos de 94 °C por 1 min, 55 °C por 30 s y 72 °C durante 30 s y finalmente 1 ciclo 72 °C por 10 min. Los amplicones obtenidos se corrieron en geles de agarosa al 1 %. El producto obtenido, se purificó utilizando un DNA Clean y Concentrator kit (ZYMO, Research) siguiendo las instrucciones del fabricante. Finalmente, la secuenciación se llevó a cabo en un secuenciador automático de DNA de 16 capilares (Applied Biosystems, modelo 3130xl) en la Unidad de Síntesis y Secuenciación de DNA (USSDNA) del Instituto de Biotecnología de la UNAM.
Construcción filogenética. Las construcciones filogenéticas se llevaron a cabo a través de un análisis in silico de las secuencias obtenidas de los aislados con el gen 16S ribosomal, comparándolas con la base de datos del Gene Bank con del programa BLASTn (http://blast.ncbi.nlm.nih.gov/Blast.cgi). Se obtuvieron secuencias que tuvieron la mayor puntuación en el análisis BLASTn y se alinearon con el programa MUSCLE (www.ebi.ac.uk/Tools/msa/muscle/) para obtener un perfil. Con el alineamiento del perfil se realizó una búsqueda del mejor método de inferencia filogenética con el programa FindModel (www.hiv.lanl.gov/content/sequence/findmodel/findmodel.html). El cual resulto ser para todas las inferencias el método de máxima verosimilitud, con el modelo GRT+G (General Time Reversible plus Gamma distribution). Se alinearon las secuencias obtenidas de los aislados con el perfil obtenido del GenBank con el programa Clustal_X en el modo de alineamiento de perfiles. Con el alineamiento final se hizo la inferencia filogenética con el método mencionado y 1500 réplicas de Boostrap con el programa MEGA6 (Tamura et al., 2013). El árbol filogenético se construyó empleando a las siguientes especies para enraízarlo: Listeria monocitogenes, Psycrobactyer articus y Streptococcus constellatus pharyngis.
RESULTADOS
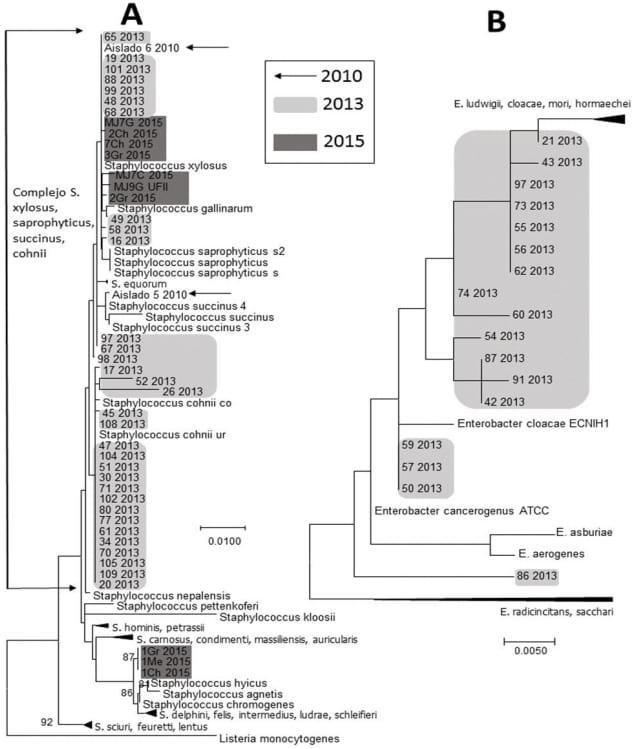
De 900 garrapatas analizadas en los años 2010, 2013 y 2015 se obtuvieron 74 aislados bacterianos a partir de los exudados de las garrapatas infectadas. A partir de la ribotificación encontramos que 44 aislados pertenecen al género Staphylococcus de los cuales 2 se colectaron en el 2010, 32 en el 2013 y 10 en el 2015 (Fig. 1A); 17 al género Enterobacter aislados en el 2013 (Fig. 1B); 8 al género Acinetobacter de los cuales 4 se aislaron en el 2013 y otros 4 en el 2015 (Fig. 2A), 4 al género Enterococcus aislados en el 2015 (Fig. 2B) y uno al género Pseudomonas aislado en el 2013 (Fig. 2C).
DISCUSIÓN
El género de bacterias con mayor incidencia en los aislados obtenidos fue Staphylococcus con 44 aislados, y se encontró de manera consistente en los exudados analizados. En su mayoría estos aislados se relacionan filogenéticamente con el complejo S. xylosus-saprophyticus-gallinarumcohnii. Se sabe que S. xylosus naturalmente se encuentra en carne en proceso de putrefacción y leche, además puede ser ocupado como inoculo inicial para carnes fermentadas. A pesar de que S. xylosus es denominado como no patogénico, algunas cepas están relacionadas con enfermedades oportunistas en animales y humanos (Dordet-Frisoni et al., 2007).Por su parte S. saprophyticus ha sido reportado previamente, como parte de la microbiota bacteriana patógena de la garrapata del ganado, lo cual confirma y es consistente con los resultados obtenidos en este estudio (MirandaMiranda et al., 2010). Las especies del género Acinetobacter son bacterias Gram negativas y catalasa positivas, que naturalmente han sido reportadas en agua, tierra, artrópodos e incluso se han identificado en muestras humanas. Asimismo, algunos reportes (Demir et al., 2012; Sevin et al., 2010) asocian bacterias de este género con actividad entomopatógena. Al respecto, nuestros resultados mostraron que seis de los ocho aislados muestran homología con A. indicus, una bacteria aislada en la India y que está asociada a tiraderos de subproductos derivados de la producción del ixodicida Lindano (Malhotra et al., 2012), cuyo papel se relaciona con la degradación de diferentes isómeros de hexaclorociclohexano por medio de la vía metabólica de Lin (Lal et al., 2010).
Figura 1. Análisis filogenético de secuencias del gen 16S generados con el método de ML bajo el modelo de GTR+G y 1500 boostraps. A) Filogenia del género Staphylococcus. B) Filogenia del género Enterobacter.
Figura 2. Análisis filogenético de secuencias del gen 16S generados con el método de ML bajo el modelo de GTR+G y 1500 boostraps. A) Filogenia del género Acinetobacter. B) Filogenia del género Enterococcus. C) Filogenia del género Pseudomonas.
Este género de bacterias también puede participar como patógenos oportunistas en vertebrados, esta es una característica importante, debido a que las garrapatas son inusuales entre los artrópodos ya que las hembras repletas son en un 90% sangre de bovino, razón por la cual enfermedades infecciosas de la sangre de los bovinos pueden dañar a las garrapatas, por ejemplo los hemopatógenos bovinos Babesia bovis, Babesia bigemina, Anaplasma spp, Theleria spp, también son patógenos para las garrapatas. Debido a estos antecedentes, durante nuestro estudio hemos puesto atención a bacterias patógenas de bovinos que pudieran infectar la sangre del hospedero que constituye el 90 % del peso de las hembras repletas. Nuestro estudio identifica Enterococcus app son usados frecuentemente como bioindicadores de contaminación fecal, pero también pueden ser usados como modelos de infección (Byappanahalli et al., 2012). Nuestros resultados indican que algunos de los aislados se encuentran relacionados filogenéticamente con E. casseliflavus, bacteria que está ampliamente distribuida en diferentes reservorios como el lobo marino (Arctocephalus australis, Arctocephalus tropicalis) (Santestevan et al., 2015), las moscas (Musca domestica) (Ghosh et al., 2014), pollos de granja (Kacániová et al., 2013), en polillas junto con Bacillus thuringiensis (Shao et al., 2011), gasterópodos (Charrier et al., 1998), en el intestino del escarabajo de la patata (Leptinotarsa decemlineata). Además se ha reportado que puede ser patógeno para el humano (Byappanahalli et al., 2012). Las muestras de exudado también mostraron aislados pertenecientes al género Enterobacter spp., mismos que se relacionan filogenéticamente con E. cloacae y con E. sacchari, bacterias encontradas abundantemente en la naturaleza, particularmente en tractos gastrointestinales y en el suelo (Mezzatesta et al., 2012), lo que probablemente se correlacione con la presencia de heces del hospedero bovino, lo cual podría indicar contaminación ambiental de las muestras.
CONCLUSIÓN
El objetivo de este trabajo fue determinar si otros microorganismos además de bacterias del género Staphylocoocus estaban presentes en el exudado de las garrapatas con signos de infección como los descritos previamente (Miranda-Miranda et al., 2010), esto con el propósito de conocer si existe alguna coinfección bacteriana que pudiera resultar en una infección más eficiente y dañina para la garrapata. Falta determinar si los organismos encontrados en los exudados pueden causar daño a las garrapatas por si mimas o potenciar la infección con Staphyloccocus, y encontrar su localización en la garrapata para entender mejor el desarrollo de la infección.
Agradecimientos
Este trabajo fue financiado por fondos fiscales INIFAP SIGI1322633028 y por fondos del proyecto Conacyt 248049.
Literatura Citada
Amoo, A., Dipeolu, O., Akinboade, A. and A. Adeyemi. 1987. Bacterial Isolation From and Transmition by Boophilus decoloratus and Boophilus geigy. Folia Parasitologica, 34: 69–74.
Byappanahalli, M. N., Nevers, M., Korajkic, A., Staley, Z. and V. Harwood. 2012. Enterococci in the enviroment. Microbiology and Molecular Biology Reviews, 76(4): 685–706.
Charrier, M., Combet-Blanc, and B. Ollivier. 1998. Bacterial flora in the gut of Helix aspersa (Gastropoda Pulmonata): evidence for a permanent population with a dominant homolactic intestinal bacterium, Enterococcus casseliflavus. Canadian Journal of Microbiology, 44(1): 20–27.
Cossio-Bayugar, R., Miranda, E. and P. Holman. 2005. Molecular cloning of a phospholipid-hydroperoxide glutathione peroxidase gene from the tick, Boophilus microplus (Acari: Ixodidae). Insect Biochemistry and Molecular Biology, 35(12): 1378–1387.
Demi?r, I., Eryu¨zlu¨, E., and Z. Demi?rbag?. 2012. A study on the characterization and pathogenicity of bacteria from Lymantria dispar L. (Lepidoptera: Lymantriidae). Turkish Journal of Biology, 36: 459–468.
Dordet-Frisoni, E., Dorchies, G., De Araujo, C., Talon, R. and S. Leroy. 2007. Genomic Diversity in Staphylococcus xylosus. Applied and Enviromental Microbiology, 73(22): 7199–7209.
Ghosh, A., Akhtar, M., Holderman, C. and L. Zurek. 2014. Significance and Survival of Enterococci During the House Fly Development. Journal of Medical Entomology, 51(1): 63–67.
Lal, R., Pandey, G., Sharma, P., Kumari, K., Malhotra, S., Pandey, R. and J. Oakeshott. 2010. Biochemistry of microbial degradation of hexachlorocyclohexane and prospects for bioremediation. Microbiology and Molecular Biology Reviews, 74(1): 58–80.
Malhotra, J., Anand, S., Jindal, S., Rajagopal, R. and R. Lal. 2012. Acinetobacter indicus sp. nov., isolated from a hexachlorocyclohexane dump site. International Journal of Systematic and Evolutionary Microbiology, 62: 2883–2890.
Mezzatesta, M., Gona, F. and S. Stefani. 2012. Enterobacter cloacae complex: clinical impact and emerging antibiotic resistance. Future Microbiology, 7(7): 887–902.
Miranda-Miranda, E., Cossío-Bayugar, R., Quezada-Delgado, M., Sachman-Ruiz, B. and R. Reynaud. 2010. Staphylococcus saprophyticus is a pathogen of the cattle tick Rhipicephalus (Boophilus) microplus. Biocontrol Science and Technology, 20: 1055–1067.
Miranda-Miranda, E., Cossío-Bayúgar, R., Martinez-Ibañez, F. and C. Bautista-Garfias. 2011. Megaselia scalaris (Diptera: Phoridae), reared on Rhipicephalus (Boophilus) microplus (Acari: Ixodidae) laboratory cultures. Medical and Veterinary Entomology, 25(3): 344–347.
Miranda-Miranda, E., Cossio-Bayugar, R., Martínez-Ibañez, F., Casasanero-Orduña R. and J. Folch-Mallol. 2012. Natural occurrence of lethal aspergillosis in the cattle tick Rhipicephalus (Boophilus) microplus (Acari:Ixodidae). Parasitology, 138: 1–5.
Kacániová, Rovná, K., Arpášová, H., Hleba, L., Petrová, J., Hašcík, P., Cubon, J., Pavelková, A., Chlebo, R., Bobková, A. and M. Stricík. 2013. The effects of bee pollen extracts on the broiler chicken’s gastrointestinal microflora. Research in Veterinary Science, 95(1): 34–37.
Tonetti, N., Berggoetz, M., Rühle, C., Pretorius, A. M. and L. Gern. 2009. Ticks and tick-borne pathogens from wildlife in the Free State Province, South Africa. Journal of Wildlife Diseases, 45(2): 437– 446.
Tamura, K., Stecher, G., Peterson, D., Filipski, A. and S. Kumar. 2013. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Molecular Biology and Evolution, 30(12): 2725–2729.
Tonetti, N., Berggoetz, M., Rühle, C., Pretorius, M. and L. Gern. 2009. Ticks and tick-borne pathogens from wildlife in the Free State Province, South Africa. Journal of Wildlife Diseases, 45(2): 437–446.
Sachman-Ruiz, B., Reynaud, E., Miranda-Miranda, E., y Cossio-Bayugar, R. 2014. Aislamiento y ribotipificación bacteriana, durante infección de la garrapata de ganado Rhipicephalus (Boophilus) microplus. Entomología mexicana, 1: 38−42.
Santestevan, N., Zvoboda, D., Prichula, J., Inhoque, R., Raffo, G., Almansa, L. and D. Blaese. 2015. Antimicrobial resistance and virulence factor gene profiles of Enterococcus spp. isolates from wild Arctocephalus australis (South American fur seal) and Arctocephalus tropicalis (Subantarctic fur seal). World Journal of Microbiology and Biotechnology, 31(12): 1935–1946.
Sevim, A., Demirbag?, Z. and I. Demir. 2010. A new study on the bacteria of Agrotis segetum Schiff. (Lepidoptera: Noctuidae) and their insecticidal activities. Turkish Journal Agriculture and Forestry, 34: 333–342.
Shao, Y., Spiteller, D., Tang, X., Ping, L., Colesie, C., Münchberg, U. and W. Boland. 2011. Crystallization of α- and β-carotene in the foregut of Spodoptera larvae feeding on a toxic food plant. Insect Biochemistry and Molecular Biology, 41(4): 273–281.
Weisburg, W. G., Barns, S. M., Pelletier, D. A. and D. J. Lane. 1991. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology, 173(2): 697–703.
Zimmer, K. R., Seixas, A., Conceição, J. M., Zvoboda, D. A., Barros, M. P., Tasca, T. and C. Termignoni. 2013. Cattle tick-associated bacteria exert anti-biofilm and anti-Trichomonas foetus activities. Veterinary Microbiology, 164(1-2):171–176.






.jpg&w=3840&q=75)