Caracterización y comparación de cepas de Pasteurella Multocida asociadas con neumonías porcinas y Rinitis atrófica progresiva.
Publicado: 17 de enero de 2017
Por: Fernando Bessone (Instituto Nacional de Tecnología Agropecuaria - INTA); Soriano M. L; Fabrisio Alustiza (Becario CONICET en Univ. Nacional de Rio Cuarto); Piscitelli H; Pablo Tamiozzo (CONICET - Universidad Nacional de Rio Cuarto), Dr. Carlos Perfumo, Gustavo Carlos Zielinski (INTA) Argentina
Introducción
Pasteurella multocida (Pm) es el agente causal de la Rinitis Atrófica Progresiva (PAR) y Pasteurelosis Neumónica (PN) en cerdos (1). Este agente forma parte del Complejo Respiratorio Porcino responsable de importantes pérdidas económicas. Los esquemas de tipificación utilizados con mayor frecuencia para Pm definen 5 tipos capsulares (A, B, D, E, F) (2). Las cepas toxigénicas asociados con PAR son frecuentemente de tipo capsular D, aunque los aislamientos toxigénicos de tipo capsular A también pueden estar involucrados en la enfermedad (3). En contraste con los aislados asociados con PAR, las cepas de Pm aisladas de PN son por lo general no toxigénicas y de tipo capsular A (1). Sin embargo, una pequeña proporción de los aislamientos de pulmón son toxigénicos y / o pueden ser de tipo capsular D (3).
El objetivo del presente estudio fue caracterizar las cepas de Pm que se recuperaron de casos de PN y PAR mediante análisis comparativo para conocer sus tipos capsulares y presencia de toxina (ToxA), a fin de relacionar su tipo capsular y/o expresión toxigénica con sintomatología clínica y/o hallazgos de necropsia.
Material y métodos
Se analizaron 59 aislamientos de Pm, los cuales fueron colectados a través de hisopados nasales y pulmones de cerdos con casos clínicos de PAR y Neumonías. Las cepas fueron cultivadas en placas de agar sangre al 6% e incubadas a 37°C durante 24 hrs. e identificadas por coloración de Gram, morfología (bacilos o cocobacilos) y prueba de oxidasa (positiva). El DNA de los aislados fue extraído mediante un kit comercial, almacenándose el DNA a -20ºC. Posteriormente se llevaron a cabo las diferentes reacciones de amplificación mediante PCR para la caracterización de especie, tipo capsular y detección de ToxA a través de la metodología descripta por Townsend y col., 2001 y Davies y col., 2003.
Resultados
Las 59 cepas estudiadas resultaron positivas a la PCR especie-especifica de Pm. Se observó que el 65% de los aislados fueron tipo capsular A o D, mientras que el 35% restante pertenecían a otros tipos capsulares. También se observó que el 15% de las cepas estudiadas amplificó para el gen ToxA.
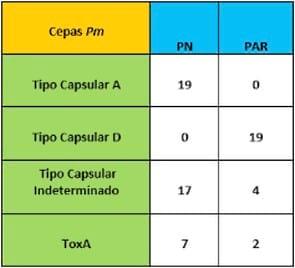
Tabla 1. Tipo capsular y presencia de toxina (ToxA) según sintomatología clínica y/o hallazgos de necropsia.
Discusión
No existe información actualizada, de los últimos años, a cerca de la situación de PAR y PN en nuestro país. La técnica de PCR resulto ser rápida y confiable para determinar los tipos capsulares A y D de Pm (3,4).
De 36 aislamientos asociado con PN 19 (53%) eran de tipo capsular A mientras que 17 (47%) pertenecían a otros tipos capsulares pudiendo ser B, E o F, ya que no se realizó dicha tipificación. En cambio, de las 23 cepas aisladas de PAR se observó que 19 (83%) fueron tipo capsular D y 4 (17%) de ellas no fueron tipificadas al igual que en el caso de las PN. La asociación en cuanto a sintomatología y tipo capsular tanto en los casos de PN con tipo capsular A, como en casos de PAR con tipo capsular D concuerdan con estudios previos (1,3). En cuanto al gen ToxA, se detectó en 7 cepas aisladas de PN y solo en 2 casos de PAR, esto difiere con estudios anteriores (5), ya que es poco frecuente encontrar cepas tipo A positivas aisladas de PN, que por lo general son no-toxígenas.
En estudios recientes (3) se observaron similares hallazgos en cuanto a la tipificación de las cepas en relación a la sintomatología, esto se debe a la transferencia horizontal del gen ToxA que existe entre las Pm (3), donde podrían tener relación a casos de PAR.
Podemos determinar con estos resultados que Pm podría cumplir un rol etiológico primario en la patogenia de las neumonías porcinas y no siempre comportarse como patógeno oportunista como citan en otros trabajos (5). Es por ello que se requiere un mayor número de caracterizaciones de Pm para poder profundizar los conocimientos sobre este patógeno, tanto en cepas aisladas a través de casos de PAR, como también en los aislados de PN y desarrollar modelos de infección experimental a fin de correlacionar la expresión genética de los aislados con la producción de enfermedad en los huéspedes
Bibliografía
1. Pijoan y col. Serotyping of Pasteurella multocida isolated from swine lungs collected at slaughter. J Clin Microbiol 17, 1074–1076, 1983.
2. Leotta y col. Identificación, biotipificación y caracterización de cepas de Pasteurella multocida aisladas en la Argentina. Revista Argentina de Microbiología 38: 125-129, 2006.
3. Davies y col. Characterization and comparison of Pasteurella multocida strains associated with porcine pneumonia and atrophic rhinitis. Journal of Medical Microbiology 52, 59–67, 2003.
4. Townsend y col. Genetic organization of Pasteurella multocida cap loci and development of a multiplex capsular PCR typing system. J Clin Microbiol 39, 924– 929, 2001.
5. Sakano y col. Toxigenic type A Pasteurella multocida as a causative agent of nasal turbinate atrophy in swine. J Vet Med Sci 54, 403–407, 1992.
Contenido del evento:
Temas relacionados:
Autores:
Instituto Nacional de Tecnología Agropecuaria - INTA
Instituto Nacional de Tecnología Agropecuaria - INTA
Universidad Nacional de Rio Cuarto - UNRC
Universidad Nacional de La Plata - UNLP
Instituto Nacional de Tecnología Agropecuaria - INTA
Mostrar más
Recomendar
Comentar
Compartir

¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.









