
Contenido patrocinado por:
Laboratorio Avimex, S. A de C. V.
Análisis genético del virus de la diarrea epidémica porcina (PED) en México 2013 - 2015
Publicado: 26 de noviembre de 2015
Por: Rubén Echeveste García de Alba, Quezada M. F., Cortés F. R., Bernardo Lozano, David Sarfati Mizrahi, Ernesto Soto, Dr. Horacio Lara Puente. Laboratorio Avi-Mex, S. A. de C. V. México
Introducción La presencia de la Diarrea Epidémica Porcina (DEP) se reportó por primera vez en México y oficialmente ante la OIE en mayo del 2014, con base a muestras remitidas para su análisis molecular al SENASICA. Con el objetivo de aportar información sobre la homología genética del virus de DEP (VDEP) y determinar su distribución geográfica, se realizó un análisis de secuencias a partir de muestras provenientes de diferentes estados del país. Material y Método Se analizaron los resultados de 98 secuencias de nucleótidos obtenidas de la región S1 de los VDEP a partir de muestras provenientes de 6 estados de la República que incluyeron Sonora, Jalisco, Puebla, Veracruz, Estado de México y Morelos, y que fueron remitidas entre el 2013 y febrero del 2015. Los resultados se analizaron con el programa Vector NTI ver. 11.0 para la generación de dendogramas. La determinación de una rama o subtipo fue estimada con una diferencia genética ≥1%, mientras que secuencias con una homología genética entre el 0.5% y 0.01% fueron consideradas como una misma variante antigénica o clade. Se tomó como “virus tipo” a las secuencias identificadas como “Illinois 63, 2013” y que proviene de los Estados Unidos de Norteamérica. Resultados y Discusión En el esquema 1 se observa la agrupación de las 98 secuencias genéticas en 3 ramas y más de 18 clades, sin que ello signifique diferencias antigénicas en alguna de las muestras. Las muestras de Jalisco corresponden a las 3 ramas encontradas, mientras que las muestras de Sonora y algunas de Jalisco corresponden a la rama 2; de la rama 3 (hasta el momento la más amplia) se detectan las secuencias de Morelos, Veracruz, Puebla y el Estado de México. En muestras de Jalisco y Veracruz se detectaron secuencias que presentan la mayor variación genética (1.5%). Las secuencias de una muestra proveniente de Jalisco tiene un 100% de homología genética con el VDEP “Illinois 63, 2013”. | Esquema 1 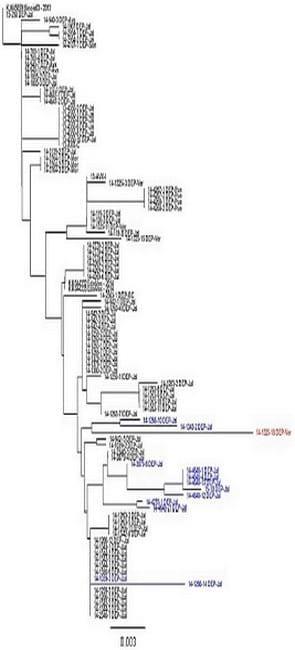 |
Conclusiones
Se determinó que las 98 secuencias analizadas presentan una buena homología genética entre ellas (<1.5%).
Referencias Bibliográficas
1. Heneidi Z. A. 2º Simposio DEP en México, FMVZ UNAM 2014.
2. FAO, IAEA, EIB Animal Pathogens phylogenetics: Basic principles and its application. 2014.
Temas relacionados
Autores:
Mostrar más
Únete para poder comentar.
Una vez que te unas a Engormix, podrás participar en todos los contenidos y foros.
* Dato obligatorio
¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Crear una publicación




