Desarrollo de un ensayo de PCR en tiempo real para la detección de un linaje distinto de IBDV (dIBDV)
Publicado: 28 de octubre de 2015
Por: Gonzalo Tomás, Martín Hernández, Ana Marandino, Diego Hernández, Yanina Panzera, Claudia Techera, Ruben Pérez. Grupo de Investigación en Genética de Microorganismos, Sección Genética Evolutiva, Facultad de Ciencias, Uruguay.
Introducción
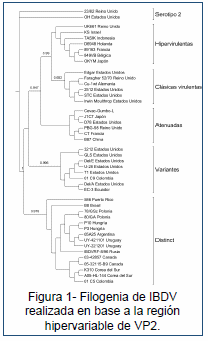
El virus de Gumboro (Birnaviridae, Avibirnavirus), es un patógeno que afecta gravemente la industria avícola mundial, provocando una enfermedad inmunodepresora en aves jóvenes. Su genoma está compuesto por dos segmentos de ARN doble hebra llamados A y B, los que codifican cinco proteínas virales (VP1-5). De acuerdo a criterios antigénicos y patogénicos, IBDV se ha clasificado en cuatro cepas: clásicas virulentas, atenuadas, variantes e hipervirulentas. Estudios filogenéticos recientes basados en la región hipervariable de la proteína VP2 (rhVP2), que incluyen cepas de todo el mundo, indicaron que además de las cuatro cepas recién nombradas existe un quinto linaje claramente definido [1]. A este linaje se le llamó distinct IBDV (dIBDV), y está formado por cepas de Sudamérica, Centroamérica, Norteamérica, Europa y Asia (Fig. 1). Si bien aún no hay estudios de patogenicidad, se ha visto que a nivel de campo los dIBDVs tienen una manifestación subclínica. Desde el punto de vista antigénico, estudios con anticuerpos monoclonales indicaron que los dIBDVs son diferentes a las cuatro cepas conocidas [2]. Esto se corresponde con la presencia de varios cambios aminoacídicos conservados en este grupo viral dentro de la rhVP2 (Fig. 2). Las características genéticas y antigénicas particulares, sumado a su alta prevalencia en algunos países [3] (Tabla I), hacen necesario el desarrollo de herramientas que permitan detectar específicamente este linaje viral para conocer mejor su distribución y prevalencia.


Objetivo
Diseñar, estandarizar y validar un ensayo de PCR en tiempo real que permita la detección rápida y sensible del linaje dIBDV.
Materiales y métodos
Se realizaron alineamientos nucleotídicos de la rhVP2, incluyendo secuencias de cepas clásicas virulentas, atenuadas, variantes e hipervirulentas, además de las secuencias del linaje dIBDV. En base a marcadores nucleotídicos exclusivos de los
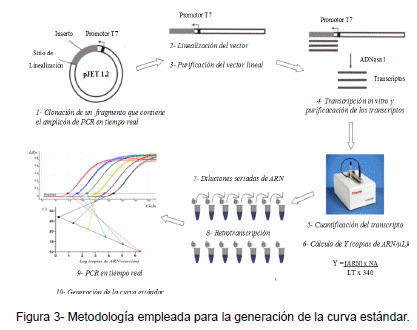
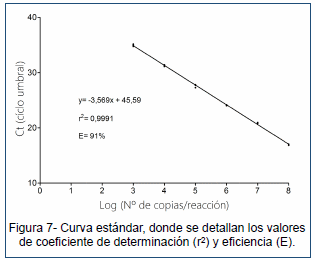
dIBDV, se diseñó una sonda TaqMan-MGB y un juego de cebadores que amplifica un fragmento de 72 pb. La amplificación se puso a punto utilizando un equipo ABI 7500 (Applied Biosystems). La validación del método se realizó mediante análisis de especificidad, sensibilidad clínica y desempeño analítico. Las muestras utilizadas en la validación fueron muestras de campo uruguayas, argentinas y estadounidenses, además de cepas vacunales. El desempeño analítico del ensayo se evaluó generando una curva estándar a partir de transcriptos de ARN (Fig. 3), analizando la eficiencia, el coeficiente de determinación, el rango dinámico y el coeficiente de variación.
dIBDV, se diseñó una sonda TaqMan-MGB y un juego de cebadores que amplifica un fragmento de 72 pb. La amplificación se puso a punto utilizando un equipo ABI 7500 (Applied Biosystems). La validación del método se realizó mediante análisis de especificidad, sensibilidad clínica y desempeño analítico. Las muestras utilizadas en la validación fueron muestras de campo uruguayas, argentinas y estadounidenses, además de cepas vacunales. El desempeño analítico del ensayo se evaluó generando una curva estándar a partir de transcriptos de ARN (Fig. 3), analizando la eficiencia, el coeficiente de determinación, el rango dinámico y el coeficiente de variación.
Resultados
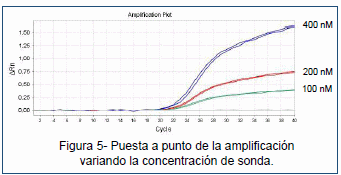
Se diseñó un juego de cebadores y sonda sobre una región genómica con cambios nucleotídicos conservados, capaces de diferenciar este tipo viral del resto de las cepas (Fig. 4). La puesta a punto del ensayo permitió mejorar su funcionamiento,
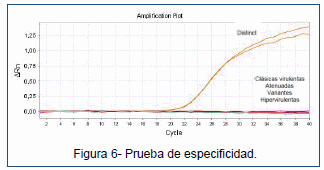
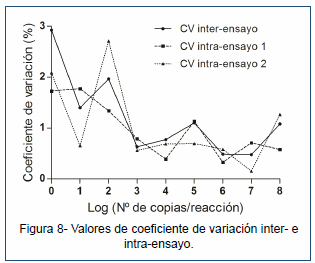
aumentando los niveles de fluorescencia final de forma notoria (Fig. 5). La validación del ensayo demostró que el mismo tiene una buena especificidad, no detectándose amplificación cuando se enfrentó la sonda con muestras de cepa clásica virulenta, atenuada, variante o hipervirulenta (Fig. 6). La curva estándar indicó un valor de eficiencia de 91 %, un coeficiente de determinación de 0,9991 y un rango dinámico establecido entre las 103-108 copias de ADNc por reacción (Fig. 7). Los valores de coeficiente de variación, tanto intra- como inter-ensayo, no superaron el 3 % (Fig. 8), demostrando la gran reproducibilidad del ensayo.
aumentando los niveles de fluorescencia final de forma notoria (Fig. 5). La validación del ensayo demostró que el mismo tiene una buena especificidad, no detectándose amplificación cuando se enfrentó la sonda con muestras de cepa clásica virulenta, atenuada, variante o hipervirulenta (Fig. 6). La curva estándar indicó un valor de eficiencia de 91 %, un coeficiente de determinación de 0,9991 y un rango dinámico establecido entre las 103-108 copias de ADNc por reacción (Fig. 7). Los valores de coeficiente de variación, tanto intra- como inter-ensayo, no superaron el 3 % (Fig. 8), demostrando la gran reproducibilidad del ensayo.

Conclusiones
? Los dIBDV son un grupo viral con características genéticas y antigénicas particulares, diferentes a los demás grupos o cepas virales de IBDV típicamente descritos.
? Los dIBDV tienen una alta prevalencia en los últimos años tanto en Argentina como en Uruguay.
? Se logró el diseño, estandarización y validación de un método basado en PCR en tiempo real capaz de detectar los dIBDV.
? Este método demostró ser altamente específico, sensible y reproducible, permitiendo su utilización en cualquier laboratorio y sobre cualquier tipo de muestra.
? La utilización de este método es una alternativa rápida y sensible para la realización de un screening en busca de virus del grupo dIBDV.
Referencias bibliográficas
1- Hernández M, Tomás G, Marandino A, Iraola G, Maya L, Mattion N, Hernández D, Villegas P, Banda A, Panzera Y, Pérez R. Genetic characterization of South American infectious bursal disease virus reveals the existence of a distinct worldwide-spread genetic lineage. Avian Pathology 44(3):212-21. 2015.
2- Domanska K, Rivallan G, Smietanka K, Toquin D, Minta Z, Eterradossi N. Antigenic characterization of Polish infectious bursal disease virus strains. Bulletin of the Veterinary Institute in Pulawy 46:45-52. 2002.
3- Vera F, Craig MI, Olivera V, Rojas F, König G, Pereda A, Vagnozzi A. Molecular characterization of infectious bursal disease virus (IBDV) isolated in Argentina indicates a regional lineage. Archives of Virology DOI 10.1007/s00705-015-2449 b 4. 2015.
Temas relacionados
Autores:
Únete para poder comentar.
Una vez que te unas a Engormix, podrás participar en todos los contenidos y foros.
* Dato obligatorio
¿Quieres comentar sobre otro tema? Crea una nueva publicación para dialogar con expertos de la comunidad.
Crear una publicación




